
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 5,578,403 – 5,578,553 |
| Length | 150 |
| Max. P | 0.992265 |

| Location | 5,578,403 – 5,578,513 |
|---|---|
| Length | 110 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.76 |
| Mean single sequence MFE | -43.85 |
| Consensus MFE | -36.41 |
| Energy contribution | -36.73 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.873070 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
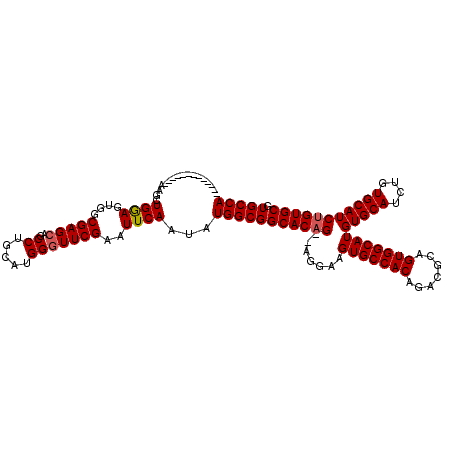
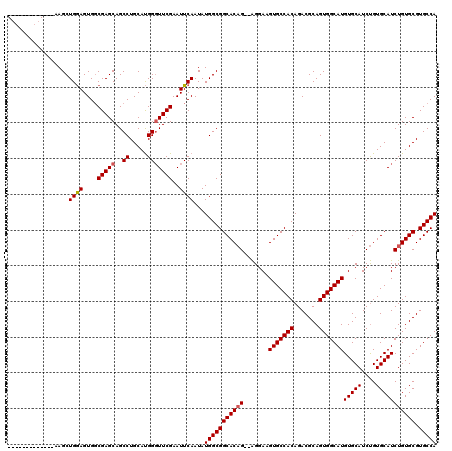
>2L_DroMel_CAF1 5578403 110 + 22407834 -GGGAA-------AAGCUGGAGUGUCGAGCAGCCUGCAUGGGUUCGAAUUCAAUAUGGCGGCACAG--AGGAAGUGCCACAGACGCAGUGGCAUGUGCAUCUGUGCAUCUGUGCGUGCCA -.....-------....((((...((((((..((.....)))))))).))))...(((((((((((--(....(((((((.......))))))).((((....))))))))))).))))) ( -42.70) >DroSec_CAF1 22402 105 + 1 -------------AAGCUGGAGUGGCGAGCAGCCUGCAUGGGUUCGAAUUCAAUAUGGCGGCACAG--AGGAAGUGCCACAGACGCAGUGGCAUGUGCAUCUGUGCAUCUGUGCGUGCCA -------------......((((..(((((..((.....))))))).))))....(((((((((((--(....(((((((.......))))))).((((....))))))))))).))))) ( -41.80) >DroSim_CAF1 24056 105 + 1 -------------AAGCUGGAGUGGCGAGCAGCCUGCAUGGGUUCGAAUUCAAUAUGGCGGCACAG--AGGAAGUGCCACAGACGCAGUGGCAUGUGCAUCUGUGCAUCUGUGCGUGCCA -------------......((((..(((((..((.....))))))).))))....(((((((((((--(....(((((((.......))))))).((((....))))))))))).))))) ( -41.80) >DroYak_CAF1 22280 120 + 1 CGGGAAACCGGGGUAGCUGAAGUGGCGAGCAGCCUGCACGGUUUCGAAUUCAAUAUGGCGGCACUGUGGGGAUGUGCCACAGACGCAGUGGCAUGUGCAUCCGUGCAUCUGUGCGUGCCA ...(((((((..(((((((..........))).)))).)))))))..........(((((((((((((.(((((((((((.......)))))....)))))).))))...)))).))))) ( -49.10) >consensus _____________AAGCUGGAGUGGCGAGCAGCCUGCAUGGGUUCGAAUUCAAUAUGGCGGCACAG__AGGAAGUGCCACAGACGCAGUGGCAUGUGCAUCUGUGCAUCUGUGCGUGCCA .................((((....(((((..((.....)))))))..))))...(((((((((((.......(((((((.......)))))))(((((....))))))))))).))))) (-36.41 = -36.73 + 0.31)

| Location | 5,578,403 – 5,578,513 |
|---|---|
| Length | 110 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.76 |
| Mean single sequence MFE | -37.50 |
| Consensus MFE | -32.19 |
| Energy contribution | -32.00 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.33 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.66 |
| SVM RNA-class probability | 0.970713 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
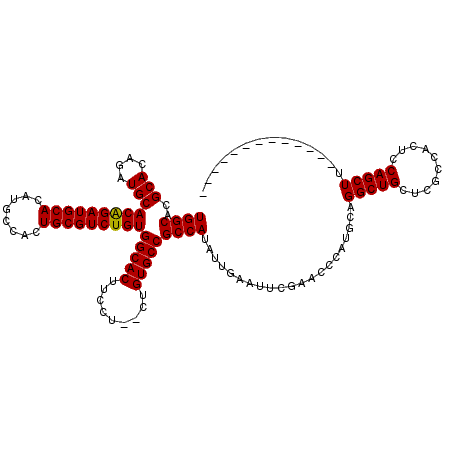
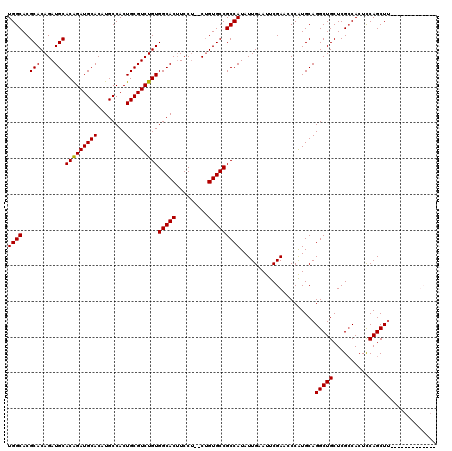
>2L_DroMel_CAF1 5578403 110 - 22407834 UGGCACGCACAGAUGCACAGAUGCACAUGCCACUGCGUCUGUGGCACUUCCU--CUGUGCCGCCAUAUUGAAUUCGAACCCAUGCAGGCUGCUCGACACUCCAGCUU-------UUCCC- ((((..(((((((((((((((((((........))))))))).))).....)--)))))).))))....................((((((..........))))))-------.....- ( -35.90) >DroSec_CAF1 22402 105 - 1 UGGCACGCACAGAUGCACAGAUGCACAUGCCACUGCGUCUGUGGCACUUCCU--CUGUGCCGCCAUAUUGAAUUCGAACCCAUGCAGGCUGCUCGCCACUCCAGCUU------------- ((((..(((((((((((((((((((........))))))))).))).....)--)))))).)))).....................(((.....)))..........------------- ( -35.90) >DroSim_CAF1 24056 105 - 1 UGGCACGCACAGAUGCACAGAUGCACAUGCCACUGCGUCUGUGGCACUUCCU--CUGUGCCGCCAUAUUGAAUUCGAACCCAUGCAGGCUGCUCGCCACUCCAGCUU------------- ((((..(((((((((((((((((((........))))))))).))).....)--)))))).)))).....................(((.....)))..........------------- ( -35.90) >DroYak_CAF1 22280 120 - 1 UGGCACGCACAGAUGCACGGAUGCACAUGCCACUGCGUCUGUGGCACAUCCCCACAGUGCCGCCAUAUUGAAUUCGAAACCGUGCAGGCUGCUCGCCACUUCAGCUACCCCGGUUUCCCG ((((....(((((((((.(...((....))..))))))))))(((((.........)))))))))..........(((((((.(..(((((..........)))))..).)))))))... ( -42.30) >consensus UGGCACGCACAGAUGCACAGAUGCACAUGCCACUGCGUCUGUGGCACUUCCU__CUGUGCCGCCAUAUUGAAUUCGAACCCAUGCAGGCUGCUCGCCACUCCAGCUU_____________ ((((..(((....)))(((((((((........)))))))))(((((.........))))))))).....................(((((..........))))).............. (-32.19 = -32.00 + -0.19)

| Location | 5,578,435 – 5,578,553 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.63 |
| Mean single sequence MFE | -43.66 |
| Consensus MFE | -38.50 |
| Energy contribution | -39.30 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.88 |
| SVM decision value | 1.00 |
| SVM RNA-class probability | 0.898273 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5578435 118 + 22407834 GGUUCGAAUUCAAUAUGGCGGCACAG--AGGAAGUGCCACAGACGCAGUGGCAUGUGCAUCUGUGCAUCUGUGCGUGCCAGUGGGCUCUCUCAUUGAACCAACAGCUUGUGGCGUCAAUU (((((((........(((((((((((--(....(((((((.......))))))).((((....))))))))))).))))).((((....)))))))))))....((.....))....... ( -44.50) >DroSec_CAF1 22429 118 + 1 GGUUCGAAUUCAAUAUGGCGGCACAG--AGGAAGUGCCACAGACGCAGUGGCAUGUGCAUCUGUGCAUCUGUGCGUGCCAGUGGGCUCUCUCAUUGAACCAACAGCUUGUGGCGUCAAUU (((((((........(((((((((((--(....(((((((.......))))))).((((....))))))))))).))))).((((....)))))))))))....((.....))....... ( -44.50) >DroSim_CAF1 24083 118 + 1 GGUUCGAAUUCAAUAUGGCGGCACAG--AGGAAGUGCCACAGACGCAGUGGCAUGUGCAUCUGUGCAUCUGUGCGUGCCAGUGGGCUCUCUCAUUGAACCAACAGCUUGUGGCGUCAAUU (((((((........(((((((((((--(....(((((((.......))))))).((((....))))))))))).))))).((((....)))))))))))....((.....))....... ( -44.50) >DroEre_CAF1 22343 118 + 1 GGCUCGAAUUCAAUAUGGCGGCACAG--AGGAAGUGCCACAGACGCACUGGCAUGUGCAUCUGUGCAUCUGUGCGUGCCAGUGGGCUCUCUCAUUGAACCAACAGCUUGUGGCGUCAAUU ((((((.........(((((((((((--(.....(((.(((((.((((......)))).))))))))))))))).))))).)))))).....(((((.(((........)))..))))). ( -43.10) >DroYak_CAF1 22320 120 + 1 GUUUCGAAUUCAAUAUGGCGGCACUGUGGGGAUGUGCCACAGACGCAGUGGCAUGUGCAUCCGUGCAUCUGUGCGUGCCAGUGGGCGCUCUCAUUGAACCAACAGCUUGUGGCGUCAAUU ...............(((((.(((((((.(((((((((((.......)))))....)))))).)))).(((((.((..(((((((....))))))).))).))))...))).)))))... ( -41.70) >consensus GGUUCGAAUUCAAUAUGGCGGCACAG__AGGAAGUGCCACAGACGCAGUGGCAUGUGCAUCUGUGCAUCUGUGCGUGCCAGUGGGCUCUCUCAUUGAACCAACAGCUUGUGGCGUCAAUU (((((((........(((((((((((.......(((((((.......)))))))(((((....))))))))))).))))).((((....)))))))))))....((.....))....... (-38.50 = -39.30 + 0.80)

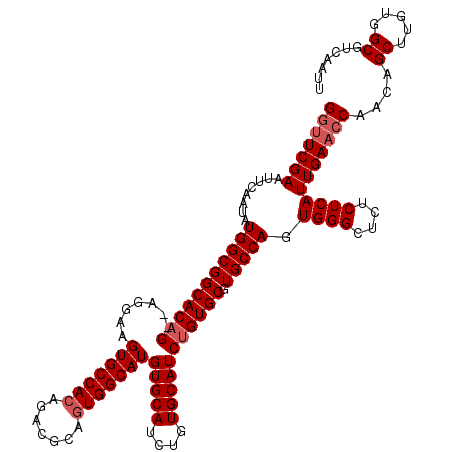
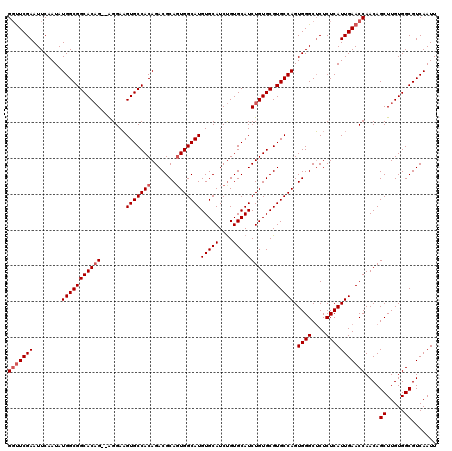
| Location | 5,578,435 – 5,578,553 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.63 |
| Mean single sequence MFE | -39.98 |
| Consensus MFE | -36.02 |
| Energy contribution | -35.86 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.32 |
| SVM RNA-class probability | 0.992265 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5578435 118 - 22407834 AAUUGACGCCACAAGCUGUUGGUUCAAUGAGAGAGCCCACUGGCACGCACAGAUGCACAGAUGCACAUGCCACUGCGUCUGUGGCACUUCCU--CUGUGCCGCCAUAUUGAAUUCGAACC ..((((...........((.(((((.......))))).))((((..(((((((((((((((((((........))))))))).))).....)--)))))).))))........))))... ( -39.60) >DroSec_CAF1 22429 118 - 1 AAUUGACGCCACAAGCUGUUGGUUCAAUGAGAGAGCCCACUGGCACGCACAGAUGCACAGAUGCACAUGCCACUGCGUCUGUGGCACUUCCU--CUGUGCCGCCAUAUUGAAUUCGAACC ..((((...........((.(((((.......))))).))((((..(((((((((((((((((((........))))))))).))).....)--)))))).))))........))))... ( -39.60) >DroSim_CAF1 24083 118 - 1 AAUUGACGCCACAAGCUGUUGGUUCAAUGAGAGAGCCCACUGGCACGCACAGAUGCACAGAUGCACAUGCCACUGCGUCUGUGGCACUUCCU--CUGUGCCGCCAUAUUGAAUUCGAACC ..((((...........((.(((((.......))))).))((((..(((((((((((((((((((........))))))))).))).....)--)))))).))))........))))... ( -39.60) >DroEre_CAF1 22343 118 - 1 AAUUGACGCCACAAGCUGUUGGUUCAAUGAGAGAGCCCACUGGCACGCACAGAUGCACAGAUGCACAUGCCAGUGCGUCUGUGGCACUUCCU--CUGUGCCGCCAUAUUGAAUUCGAGCC ..............(((((.(((((.......))))).))((((..((((((((((((((((((((......)))))))))).))).....)--)))))).))))...........))). ( -44.50) >DroYak_CAF1 22320 120 - 1 AAUUGACGCCACAAGCUGUUGGUUCAAUGAGAGCGCCCACUGGCACGCACAGAUGCACGGAUGCACAUGCCACUGCGUCUGUGGCACAUCCCCACAGUGCCGCCAUAUUGAAUUCGAAAC .((..((((.....)).))..))((((((.(.(((((....)))....(((((((((.(...((....))..))))))))))(((((.........)))))))).))))))......... ( -36.60) >consensus AAUUGACGCCACAAGCUGUUGGUUCAAUGAGAGAGCCCACUGGCACGCACAGAUGCACAGAUGCACAUGCCACUGCGUCUGUGGCACUUCCU__CUGUGCCGCCAUAUUGAAUUCGAACC .(((((.((((........)))))))))..(((..(....((((..(((....)))(((((((((........)))))))))(((((.........)))))))))....)..)))..... (-36.02 = -35.86 + -0.16)

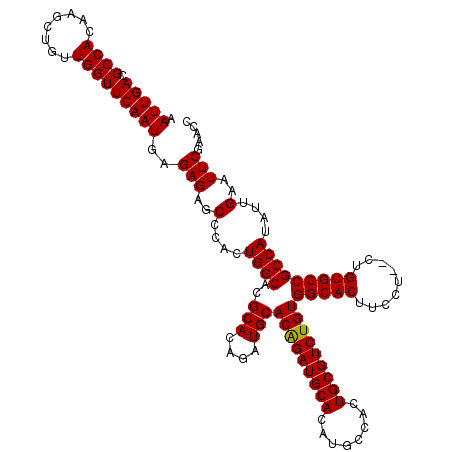
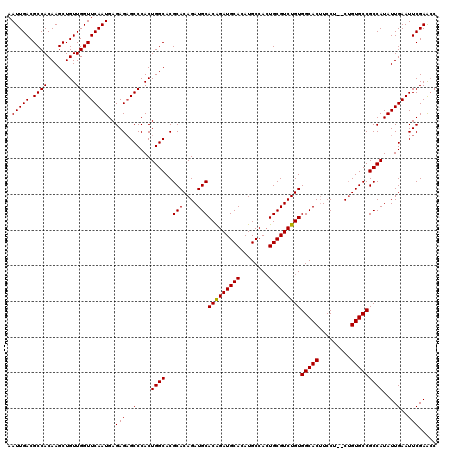
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:18:58 2006