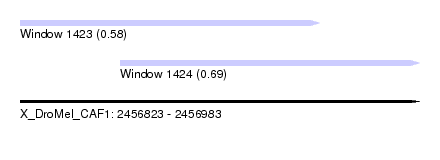
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 2,456,823 – 2,456,983 |
| Length | 160 |
| Max. P | 0.689792 |

| Location | 2,456,823 – 2,456,943 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.78 |
| Mean single sequence MFE | -46.77 |
| Consensus MFE | -29.30 |
| Energy contribution | -30.00 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.581724 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
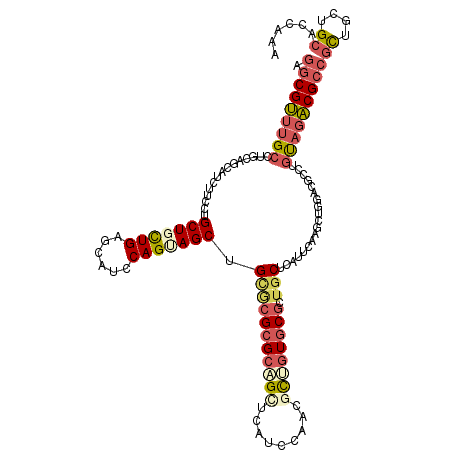
>X_DroMel_CAF1 2456823 120 + 22224390 CGCCAGCAUUUGCUCUUCAUGCUGGGAUUGGAGCAGACGAAGGCGUUUGCUUGCCGCAUCUUCCUGCUGUUGAGCAUCCAGUAGCUGCGCGCGCAGCUCAUCCAACGUUGUGCGCUGCUC .((((((((((((((..((.((.((((((((((((((((....))))))))..))).)))..)).))))..)))))...))).)))).(((((((((.........))))))))).)).. ( -45.80) >DroSec_CAF1 893 120 + 1 CGCCAGCAUUUGCUCUUCAUGCUGGACUUCGAGCAGACGAAGGCGUUUGCCUGCCAAAUCUUCCUGCUGUUGAGCUUCCAGUAGCUGCACGCGCAGGUCAUCCAACGUUGUGCGCUGCUC ..(((((((.........))))))).....((((((.((..(((((((((((((.........((((((.........))))))........))))).))...))))))...)))))))) ( -40.43) >DroSim_CAF1 1721 120 + 1 CGCCAGCAUUUCCUCUUCAUGCUGGACUUGGAGCAGACGAAGGCGUUUGCCUGCCAAAUCUUCCUGCUGUUGAGCAUCCAGUAGCUGCACGCGCAGGUCAUCCAACGUUGUGCGCUGCUC ..(((((((.........)))))))..((((.(((((((....)))))))...)))).......((((....))))...((((((.(((((((..((....))..)).))))))))))). ( -42.70) >DroEre_CAF1 16381 120 + 1 GGCCAGCAUUUGUUCUUCAUGCUCGGAUUGGAGCAGACGGAUGCGUUUGCUCAAAGUCUCCUCUUGCUGCUGAGCAUCCAGGAGCUGUGCGCGCUGCUCAUCCAACGCUGUGCGCUGCUC ....((((...((((((.((((((((....(((((((((....))))))))).(((......)))....))))))))..))))))...((((((.((.........)).)))))))))). ( -45.70) >DroYak_CAF1 14783 120 + 1 GGCCAGCAUCUGCUCUUCAUGCUCGGAUUGGACCAGACGGAGGCGUUUGCUUGCAGCCUCCCCCUGCUGCUGAGCAUCCAGGAGCUGUGCGCGCUGCUCAUCCAACGCUGUGCGCUGCUC ....((((...((((((.((((((((...((..(((..((((((((......)).))))))..))))).))))))))..))))))...((((((.((.........)).)))))))))). ( -48.10) >DroPer_CAF1 8439 120 + 1 GGCGAGCAUUUGUUCCUCGUGGUUGGCCUCGACCUGGCGCAGCCGCUUGGCCAGAGCCUCGUGCCGCUGCUGGGUCUCCAGCAGCUGGCAGCGCUGUUCUUCCAUGGCGAUACGCUGCUC (((((((....))))...((((((((((.......))).)))))))...))).((((..((((((((((((((....)))))))).)))..((((((......))))))..)))..)))) ( -57.90) >consensus CGCCAGCAUUUGCUCUUCAUGCUGGGAUUGGAGCAGACGAAGGCGUUUGCCUGCAGCAUCUUCCUGCUGCUGAGCAUCCAGUAGCUGCGCGCGCAGCUCAUCCAACGCUGUGCGCUGCUC ..(((((((.........))))))).......(((((((....)))))))...............(((((((......))))))).(((((((((((.........)))))))).))).. (-29.30 = -30.00 + 0.70)

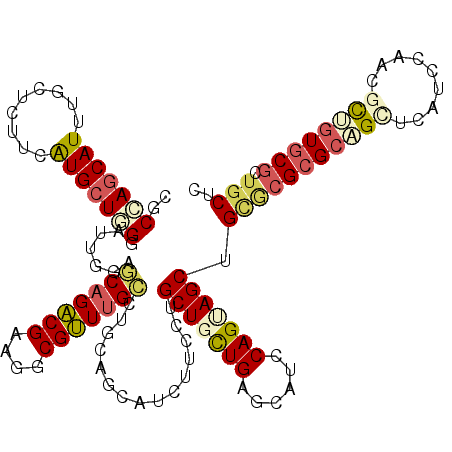
| Location | 2,456,863 – 2,456,983 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.78 |
| Mean single sequence MFE | -47.18 |
| Consensus MFE | -27.90 |
| Energy contribution | -28.93 |
| Covariance contribution | 1.03 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.33 |
| SVM RNA-class probability | 0.689792 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2456863 120 + 22224390 AGGCGUUUGCUUGCCGCAUCUUCCUGCUGUUGAGCAUCCAGUAGCUGCGCGCGCAGCUCAUCCAACGUUGUGCGCUGCUCAUUCAAGCUGGACGCCUGCAGACGCCGUUGCUGCACCAAA .(((((((((..((.(.....(((.(((..((((((....((....))(((((((((.........)))))))))))))))....))).))))))..))))))))).............. ( -47.60) >DroSec_CAF1 933 120 + 1 AGGCGUUUGCCUGCCAAAUCUUCCUGCUGUUGAGCUUCCAGUAGCUGCACGCGCAGGUCAUCCAACGUUGUGCGCUGCUCAUUCAAGAUGGAGGCCUGUAGACGCCGCUGCUGCACCAAA .(((((((((..(((..(((((...(((....)))....((((((.(((((((..((....))..)).))))))))))).....)))))...)))..)))))))))((....))...... ( -45.30) >DroSim_CAF1 1761 120 + 1 AGGCGUUUGCCUGCCAAAUCUUCCUGCUGUUGAGCAUCCAGUAGCUGCACGCGCAGGUCAUCCAACGUUGUGCGCUGCUCAUUCAUGAUGGAGGCCUGUAGACGCCGCUGCUGCACCAAA .(((((((((..(((.........((((....))))(((((((((.(((((((..((....))..)).))))))))))(((....))))))))))..)))))))))((....))...... ( -47.00) >DroEre_CAF1 16421 120 + 1 AUGCGUUUGCUCAAAGUCUCCUCUUGCUGCUGAGCAUCCAGGAGCUGUGCGCGCUGCUCAUCCAACGCUGUGCGCUGCUCAUUCAUGCUGGCCGCCUGUAUACGCCGCUGCUGCACCAAA ..((((.(((...(((......)))((.(((.(((((....((((...((((((.((.........)).)))))).))))....)))))))).))..))).)))).((....))...... ( -39.80) >DroYak_CAF1 14823 120 + 1 AGGCGUUUGCUUGCAGCCUCCCCCUGCUGCUGAGCAUCCAGGAGCUGUGCGCGCUGCUCAUCCAACGCUGUGCGCUGCUCAUUCAAACUGGCCGCCUGUAGACGCCGCUGCUGCACCAAA .(((((((((..((.(((......((((....)))).....((((...((((((.((.........)).)))))).)))).........))).))..)))))))))((....))...... ( -48.50) >DroPer_CAF1 8479 120 + 1 AGCCGCUUGGCCAGAGCCUCGUGCCGCUGCUGGGUCUCCAGCAGCUGGCAGCGCUGUUCUUCCAUGGCGAUACGCUGCUCGAAAAUGUUGUGCGCCUGCUGGCGCCGCUGCUGGACGAGG .(((....))).....((((((.((((((((((....)))))))).(((((((.........(((..(((........)))...)))....(((((....)))))))))))))))))))) ( -54.90) >consensus AGGCGUUUGCCUGCAGCAUCUUCCUGCUGCUGAGCAUCCAGUAGCUGCGCGCGCAGCUCAUCCAACGCUGUGCGCUGCUCAUUCAAGCUGGACGCCUGUAGACGCCGCUGCUGCACCAAA .(((((((((...............(((((((......))))))).(((((((((((.........)))))))).)))...................)))))))))((....))...... (-27.90 = -28.93 + 1.03)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:56:03 2006