
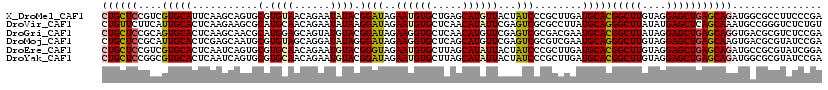
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,229,928 – 16,230,048 |
| Length | 120 |
| Max. P | 0.710671 |

| Location | 16,229,928 – 16,230,048 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.39 |
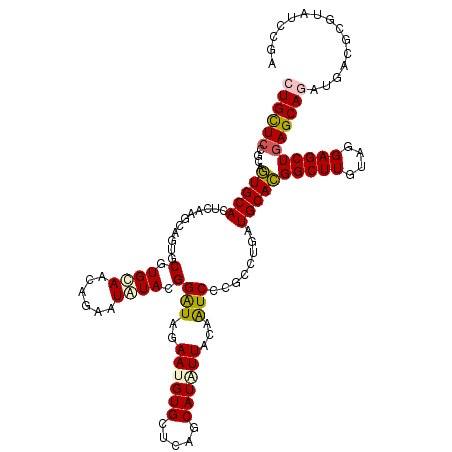
| Mean single sequence MFE | -45.95 |
| Consensus MFE | -20.91 |
| Energy contribution | -20.80 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.65 |
| Structure conservation index | 0.46 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.710671 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16229928 120 - 22224390 CUGCUCCGUCGUGCAUUCAAGCAGUGCGUGUAACAGAAUAUACGGAUAGAAUGUGCUGAGCAUGUUACUAUCCCGCUUGAUGCACGGCUUGUAGGAGCUGAGCAGAUGGCGCCUUCCCGA ..((.(((((((((((.(((((.....(((((......)))))(((((((((((((...))))))).)))))).)))))))))))(((((....))))).....))))).))........ ( -47.40) >DroVir_CAF1 4680 120 - 1 CUGUUCUUCAUUGCACUCAAGAAGCGCAUGCAACAGAAUAUAAGGAUAGAAUGUGCUCAACAUAUUCGAGUCGCGCCUUAUGCAGGGCUUAUAUGAGCUCAGCAAAUGCCGGGUCUCUGU .(((.((((.(((....))))))).)))....(((((...........(((((((.....)))))))((.(((.((.((.(((.((((((....)))))).))))).))))).))))))) ( -33.70) >DroGri_CAF1 4992 120 - 1 CUGCUCCGCAGUGCACUCAAGCAACGCAUGGAGCAGUAUGUACGGAUAGAAGGUGCUCAACAUGUUCGAGUCGCGACGAAUGCACGGCUUAUAGGAGCUGAGCAGGUGACGCGUCUCCGA (((((((((..(((......)))..))..)))))))......((((..(((.(((.....))).)))..(.((((.((..(((.((((((....)))))).)))..)).)))).))))). ( -48.00) >DroMoj_CAF1 4820 120 - 1 CUGCUCCGCAUUGCACUCGAGCAAUGCGUGUAGCAGGAUAUAGGGAUAGAAGGUGCUCAGCAUGUUCGAGUCGCGUCGAAUGCAGGGCUUGUAGGAGCUGAGCAAGUGACGCGUAUCCGA ((((((((((((((......)))))))).).))))).......((((((((.((((...)))).)))....(((((((..(((..(((((....)))))..)))..)))))))))))).. ( -54.20) >DroEre_CAF1 4628 120 - 1 CUGCUCCGUCGUGCACUCAAUCAGUGCGUGCAACAGAAUGUACGGGUAGAAUGUGCUUAGCAUAUUACUAUCCCGCUUGAUGCACGGCUUGUAGGAGCUGAGCAGAUGCCGCGUAUCGGA ..(((((((((((((.((((.(.((((((........))))))(((((((((((((...))))))).)))))).).)))))))))))).....)))))......(((((...)))))... ( -46.50) >DroYak_CAF1 4357 120 - 1 CUGCUCCGGCGUGCACUCAAUCAGUGCGUGCAACAGAAUGUACGGAUAGAAUGUGCUUAGCAUAUUACUAUCCCGCUUGAUGCACGGCUUGUAGGAGCUGAGCAGAUGGCGCGUAUCCGA ......((((((((..((.(((((.(((((((......)))))(((((((((((((...))))))).)))))).)))))))((.((((((....)))))).)).))..)))))...))). ( -45.90) >consensus CUGCUCCGCAGUGCACUCAAGCAGUGCGUGCAACAGAAUAUACGGAUAGAAUGUGCUCAGCAUAUUACAAUCCCGCCUGAUGCACGGCUUGUAGGAGCUGAGCAGAUGACGCGUAUCCGA ((((((....(((((...........(.((((......)))).)(((..((((((.....))))))...)))........)))))(((((....)))))))))))............... (-20.91 = -20.80 + -0.11)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:14:33 2006