
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 1,517,109 – 1,517,297 |
| Length | 188 |
| Max. P | 0.991601 |

| Location | 1,517,109 – 1,517,217 |
|---|---|
| Length | 108 |
| Sequences | 3 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 98.77 |
| Mean single sequence MFE | -23.97 |
| Consensus MFE | -23.30 |
| Energy contribution | -23.30 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.97 |
| SVM decision value | 0.77 |
| SVM RNA-class probability | 0.846359 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1517109 108 - 22224390 UCGCGUGUGUAUAUGUAUGUGUAUAUAAAAAAGUGAUUUUAAUUUCUGUUACUUUAUUCGUCGUAUUCGUGAAUAAGUAGAAAAAAUUACUUUCCUACUGCAUACUUG ....(((((((((((((....))))))..(((((((((((...(((((....((((((((.((....)))))))))))))))))))))))))).....)))))))... ( -24.30) >DroSec_CAF1 6572 108 - 1 GCGCGUGUGUAUAUGUAUGUGUAUAUAAAAAAGUGAUUUUAAUUUCUGUUACUUUAUUCGUCGUAUUCGUGAAUAAGUAGAAAAAAUUACUUUCCUACUGCAUCCUUG ..((.(((((((((....)))))))))..(((((((((((...(((((....((((((((.((....))))))))))))))))))))))))))......))....... ( -23.30) >DroSim_CAF1 7829 108 - 1 GCGCGUGUGUAUAUGUAUGUGUAUAUAAAAAAGUGAUUUUAAUUUCUGUUACUUUAUUCGUCGUAUUCGUGAAUAAGUAGAAAAAAUUACUUUCCUACUGCAUACUUG ....(((((((((((((....))))))..(((((((((((...(((((....((((((((.((....)))))))))))))))))))))))))).....)))))))... ( -24.30) >consensus GCGCGUGUGUAUAUGUAUGUGUAUAUAAAAAAGUGAUUUUAAUUUCUGUUACUUUAUUCGUCGUAUUCGUGAAUAAGUAGAAAAAAUUACUUUCCUACUGCAUACUUG ..((.(((((((((....)))))))))..(((((((((((...(((((....((((((((.((....))))))))))))))))))))))))))......))....... (-23.30 = -23.30 + 0.00)

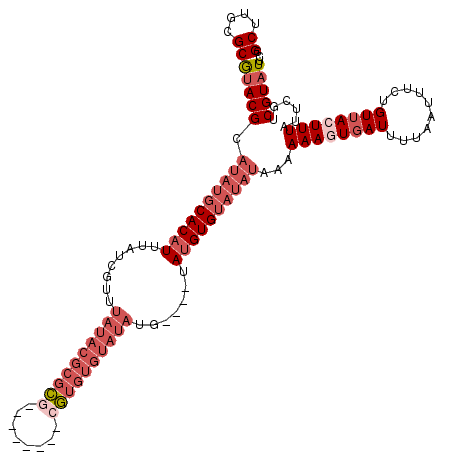
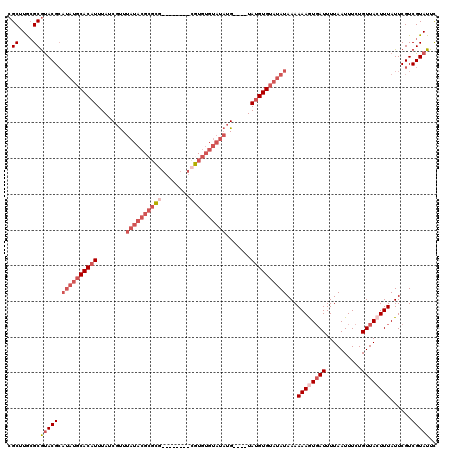
| Location | 1,517,149 – 1,517,257 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.99 |
| Mean single sequence MFE | -26.14 |
| Consensus MFE | -17.94 |
| Energy contribution | -21.30 |
| Covariance contribution | 3.36 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.27 |
| SVM RNA-class probability | 0.938715 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1517149 108 - 22224390 AGCUUGCGCAUACGCAUAUGCACAUUUAUCGUUUAUACGCUCG--------CGUGUGUAUAUG----UAUGUGUAUAUAAAAAAGUGAUUUUAAUUUCUGUUACUUUAUUCGUCGUAUUC .((....))((((((((((((((((....(((.(((((((...--------.))))))).)))----.))))))))))...((((((((..........))))))))....).))))).. ( -28.90) >DroSec_CAF1 6612 108 - 1 CGCUUGCGCGUACGCAUAUGCACAUUUAUCGUUUAUACGCGCG--------CGUGUGUAUAUG----UAUGUGUAUAUAAAAAAGUGAUUUUAAUUUCUGUUACUUUAUUCGUCGUAUUC (((....)))(((((((((((((((....(((.(((((((...--------.))))))).)))----.))))))))))...((((((((..........))))))))....).))))... ( -29.90) >DroSim_CAF1 7869 108 - 1 CGCUUGCGCGUACGCAUAUGCACAUUUAUCGUUUAUACGCGCG--------CGUGUGUAUAUG----UAUGUGUAUAUAAAAAAGUGAUUUUAAUUUCUGUUACUUUAUUCGUCGUAUUC (((....)))(((((((((((((((....(((.(((((((...--------.))))))).)))----.))))))))))...((((((((..........))))))))....).))))... ( -29.90) >DroEre_CAF1 6654 110 - 1 CGCUUACGCGUACGCAUAUGCACAUUUAUCGCUUAUACGCGUUU--UGUUAUAU--------GUGUAUACGUGUAUAUAAAAAAAUGAUUUUAAUUUCUGUUACUUUAUUCGUCGUAUUU ....((((((((((((((((.(((.....(((......)))...--))))))))--------))))..))))))).........(((((..(((...........)))...))))).... ( -23.00) >DroYak_CAF1 8189 114 - 1 CGCUUGAGCGCACG------CACAUUUAUCGUUUAUACGCGUUAUAUGUUAUAUGUGUAUAUGUGUAUAUGUGUAUAUAAAAAAAUCAUUUUAAUUUCUGUUACUUUAUUCGUCGUAUUU .......(((.(((------(((((.((.((..(((((((((..........)))))))))..)))).))))))...(((((......)))))..................))))).... ( -19.00) >consensus CGCUUGCGCGUACGCAUAUGCACAUUUAUCGUUUAUACGCGCG________CGUGUGUAUAUG____UAUGUGUAUAUAAAAAAGUGAUUUUAAUUUCUGUUACUUUAUUCGUCGUAUUC .((....))(((((.((((((((((........((((((((((........)))))))))).......))))))))))...((((((((..........))))))))......))))).. (-17.94 = -21.30 + 3.36)

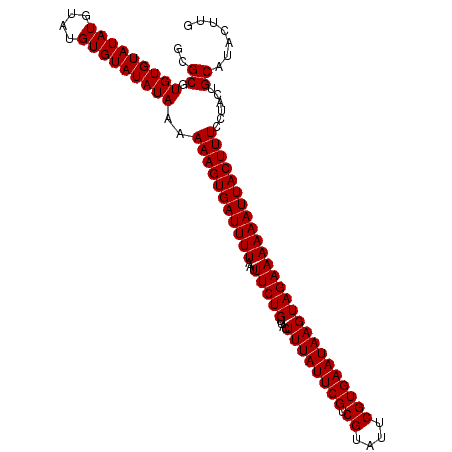
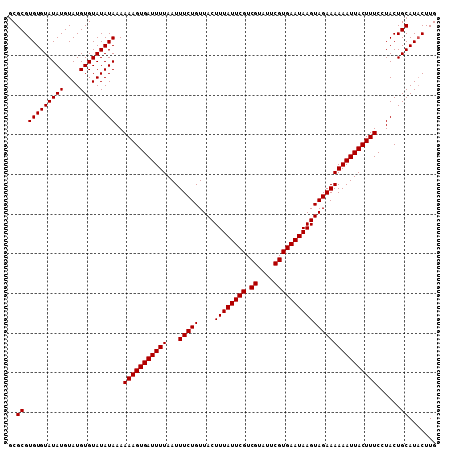
| Location | 1,517,189 – 1,517,297 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.47 |
| Mean single sequence MFE | -25.37 |
| Consensus MFE | -14.90 |
| Energy contribution | -16.30 |
| Covariance contribution | 1.40 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.45 |
| Structure conservation index | 0.59 |
| SVM decision value | 2.28 |
| SVM RNA-class probability | 0.991601 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1517189 108 + 22224390 UUAUAUACACAUA----CAUAUACACACG--------CGAGCGUAUAAACGAUAAAUGUGCAUAUGCGUAUGCGCAAGCUAAGCGAAACAACUAAACUAAUGUCUAUGUAUACAUGUAUA ...((((((.(((----((((.(((..((--------(.(((((....))......((((((((....)))))))).)))..)))...............))).)))))))...)))))) ( -23.33) >DroSec_CAF1 6652 108 + 1 UUAUAUACACAUA----CAUAUACACACG--------CGCGCGUAUAAACGAUAAAUGUGCAUAUGCGUACGCGCAAGCGAAGAGAAACAAUUAAACUAAAGUCUAUGUAUACAUGUAUA ...((((((.(((----((((.((.....--------.((((((((..(((.....)))..)))))))).(((....))).....................)).)))))))...)))))) ( -27.70) >DroSim_CAF1 7909 108 + 1 UUAUAUACACAUA----CAUAUACACACG--------CGCGCGUAUAAACGAUAAAUGUGCAUAUGCGUACGCGCAAGCGAAGAGAAACAACUAAACUAAAGUCUAUGUAUACAUGUAUA ...((((((.(((----((((.((.....--------.((((((((..(((.....)))..)))))))).(((....))).....................)).)))))))...)))))) ( -27.70) >DroEre_CAF1 6694 110 + 1 UUAUAUACACGUAUACAC--------AUAUAACA--AAACGCGUAUAAGCGAUAAAUGUGCAUAUGCGUACGCGUAAGCGAUGAGAAACAACUAUACUAAAGUCUAUGUAUACAUGUAUA ...((((((.(((((((.--------........--..((((((((..(((.....)))..)))))))).(((....)))..........................))))))).)))))) ( -28.70) >DroYak_CAF1 8229 114 + 1 UUAUAUACACAUAUACACAUAUACACAUAUAACAUAUAACGCGUAUAAACGAUAAAUGUG------CGUGCGCUCAAGCGAAGAGAAACAACUAUACUAAAGUCUAUGUAUACAUGUAUA ...........((((((..((((((..............((((((((.........))))------))))(((....)))..........................))))))..)))))) ( -19.40) >consensus UUAUAUACACAUA____CAUAUACACACG________CGCGCGUAUAAACGAUAAAUGUGCAUAUGCGUACGCGCAAGCGAAGAGAAACAACUAAACUAAAGUCUAUGUAUACAUGUAUA .((((((.((............................((((((((..(((.....)))..)))))))).(((....))).....................)).)))))).......... (-14.90 = -16.30 + 1.40)

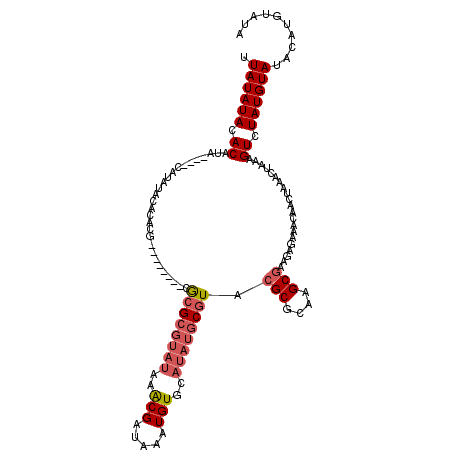
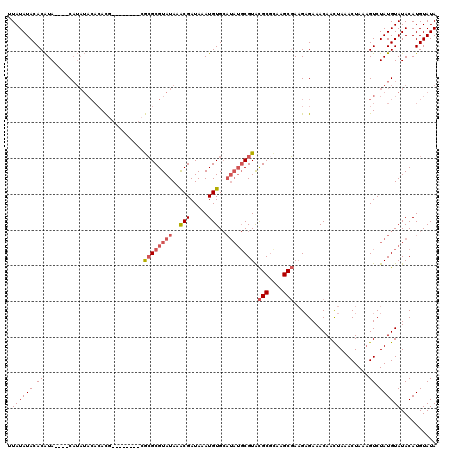
| Location | 1,517,189 – 1,517,297 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.47 |
| Mean single sequence MFE | -30.46 |
| Consensus MFE | -16.50 |
| Energy contribution | -19.66 |
| Covariance contribution | 3.16 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.60 |
| Structure conservation index | 0.54 |
| SVM decision value | 2.20 |
| SVM RNA-class probability | 0.990215 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1517189 108 - 22224390 UAUACAUGUAUACAUAGACAUUAGUUUAGUUGUUUCGCUUAGCUUGCGCAUACGCAUAUGCACAUUUAUCGUUUAUACGCUCG--------CGUGUGUAUAUG----UAUGUGUAUAUAA ......((((((((((...............((..(((.......)))...))((((((((((((....(((....)))....--------.)))))))))))----).)))))))))). ( -29.00) >DroSec_CAF1 6652 108 - 1 UAUACAUGUAUACAUAGACUUUAGUUUAAUUGUUUCUCUUCGCUUGCGCGUACGCAUAUGCACAUUUAUCGUUUAUACGCGCG--------CGUGUGUAUAUG----UAUGUGUAUAUAA (((((((((((((((((((....)))))............(((..(((((((((.(((........))))))....)))))))--------))))))))))))----))))......... ( -31.10) >DroSim_CAF1 7909 108 - 1 UAUACAUGUAUACAUAGACUUUAGUUUAGUUGUUUCUCUUCGCUUGCGCGUACGCAUAUGCACAUUUAUCGUUUAUACGCGCG--------CGUGUGUAUAUG----UAUGUGUAUAUAA (((((((((((((((((((....)))))............(((..(((((((((.(((........))))))....)))))))--------))))))))))))----))))......... ( -31.10) >DroEre_CAF1 6694 110 - 1 UAUACAUGUAUACAUAGACUUUAGUAUAGUUGUUUCUCAUCGCUUACGCGUACGCAUAUGCACAUUUAUCGCUUAUACGCGUUU--UGUUAUAU--------GUGUAUACGUGUAUAUAA ((((((((((((((((((((.......))))..........((..(((((((.(((((........))).))...)))))))..--.))....)--------)))))))))))))))... ( -31.40) >DroYak_CAF1 8229 114 - 1 UAUACAUGUAUACAUAGACUUUAGUAUAGUUGUUUCUCUUCGCUUGAGCGCACG------CACAUUUAUCGUUUAUACGCGUUAUAUGUUAUAUGUGUAUAUGUGUAUAUGUGUAUAUAA (((((((((((((((((.(((.(((..((........))..))).))))(((((------.((((.((.(((......))).)).))))....))))).))))))))))))))))..... ( -29.70) >consensus UAUACAUGUAUACAUAGACUUUAGUUUAGUUGUUUCUCUUCGCUUGCGCGUACGCAUAUGCACAUUUAUCGUUUAUACGCGCG________CGUGUGUAUAUG____UAUGUGUAUAUAA .......((((...(((((....))))).............((....))))))..((((((((((........((((((((((........)))))))))).......)))))))))).. (-16.50 = -19.66 + 3.16)

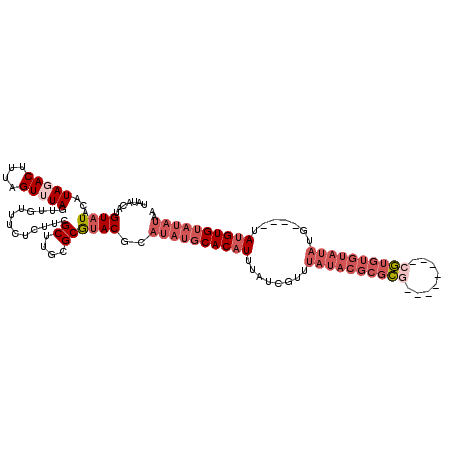
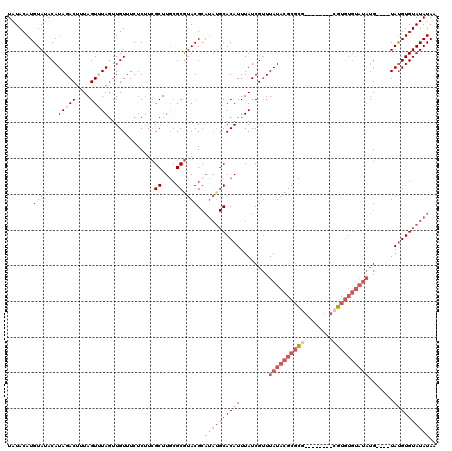
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:47:01 2006