
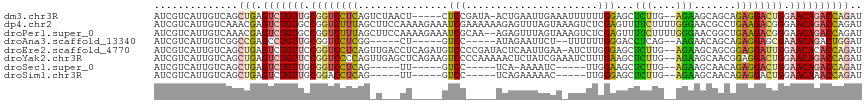
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 9,945,627 – 9,945,742 |
| Length | 115 |
| Max. P | 0.999956 |

| Location | 9,945,627 – 9,945,737 |
|---|---|
| Length | 110 |
| Sequences | 8 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 65.82 |
| Shannon entropy | 0.66844 |
| G+C content | 0.47054 |
| Mean single sequence MFE | -38.97 |
| Consensus MFE | -18.84 |
| Energy contribution | -18.65 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.54 |
| Mean z-score | -2.88 |
| Structure conservation index | 0.48 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 4.13 |
| SVM RNA-class probability | 0.999646 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 9945627 110 + 27905053 AUCGUCAUUGUCAGCUGAGUCUGUUGCGGUCCUCAGUCUAACU-----CUCCGAUA-ACUGAAUUGAAAUUUUUUGGAGCUCUUG--AGAAGCAGCAGAGGACUGGAACAGACCAGAU ..............(((.(((((((.((((((((.........-----.(((((..-...((((....)))).)))))(((((..--...)).))).)))))))).)))))))))).. ( -36.70, z-score = -1.86, R) >dp4.chr2 17352700 118 - 30794189 AUCGUCAUUGUCAAACGAGUCUGUGCCGGUCUUUAGCUUCCAAAAGAAAUCGAAAAAAGAGUUUAGUAAAGUCUCGAGUUUUCUUUUGGGAACGCCUGAAGACGGGAACAGACCAGAU .((((.........))))((((((.((.(((((((((((((((((((((((((.....((.((.....)).))))))..))))))))))))..).)))))))).)).))))))..... ( -39.80, z-score = -3.46, R) >droPer1.super_0 7007882 116 + 11822988 AUCGUCAUUGUCAAACGAGUCUGUGCCGGUCUUUAGCUUCCAAAAGAAAUCGCAA--AGAGUUUAGUAAAGUCUCGAGUUUUCUUUUGGGAACGGCUGAAGACGGGAACAGACCAGAU .((((.........))))((((((.((.((((((((((((((((((((((((...--(((.((.....)).))))))..)))))))))))...)))))))))).)).))))))..... ( -44.00, z-score = -4.53, R) >droAna3.scaffold_13340 3178125 99 - 23697760 AUCGUCAUUGUCGGCCGAGCCUGUUGCGGUCCUCGG-----CU-----GUCC-----AUAGAAUUCU--UUUUUUGGGACCUCAG--AAGAACAGCAGAGGAGCGAAACAGACUGGAU ...(((...((((((...)))((((.((.(((((.(-----((-----((..-----..((....))--((((((((....))))--))))))))).))))).)).)))))))..))) ( -33.00, z-score = -1.10, R) >droEre2.scaffold_4770 9494440 115 + 17746568 AUCGUCAUUGUCAGCUGAGUCUGUUCCGGUCCUCAGUUGACCUCAGAUGUCCCCGAUACUCAAUUGAA-AUCUUGGGAGCUCUUG--AGAAGCAGCGGAGGAUUGGAACACACCAGAU ..............(((.((.(((((((((((((.((((..(((((..(..(((((...((....)).-...)))))..)..)))--))...)))).))))))))))))).))))).. ( -45.50, z-score = -3.35, R) >droYak2.chr3R 12348906 116 - 28832112 AUCGUCAUUGUCAGCUGAGUCUGUUCCGGUCCCCAGUUGAGCUCAGAAGUCCCCAAAAACUCUAUCGAAAUCUUUGAAGCUCUUG--AGAAGCAACGGAGGACUGGAACAGACCAGAU ..............(((.((((((((((((((((.((((..((((..(((...((((...((....))....))))..)))..))--))...)))))).))))))))))))))))).. ( -45.80, z-score = -4.53, R) >droSec1.super_0 9049959 95 - 21120651 AUCGUCAUUGUCAGCUGAGUCUGUUGCGGUCCUCAG-----UU-----GUCC-----UCA-AAAAUC-----UUGGAAGCUCUUG--AGAAGCAACAGAGGACUGGAACAGACCAGAU ..............(((.(((((((.((((((((.(-----((-----((.(-----(((-(.....-----..........)))--))..))))).)))))))).)))))))))).. ( -38.36, z-score = -3.65, R) >droSim1.chr3R 16033749 96 - 27517382 AUCGUCAUUGUCAGCUGAGUCUGUUGCGGACCUCAG-----UU-----GUCC-----UCAGAAAAAC-----UUGGGAGCUCUUG--AGAAGCAACAGAGGACUGGAACAAACCAGAU ...........((((((((((((...)))).)))))-----))-----)(((-----((.......(-----(..((....))..--))........)))))((((......)))).. ( -28.56, z-score = -0.56, R) >consensus AUCGUCAUUGUCAGCUGAGUCUGUUCCGGUCCUCAG_U___CU_____GUCC_____ACAGAAUAGUA_AUUUUGGAAGCUCUUG__AGAAGCAGCAGAGGACUGGAACAGACCAGAU ..............(((.(((((((.((((((((...............(((......................)))...(((....))).......)))))))).)))))))))).. (-18.84 = -18.65 + -0.19)
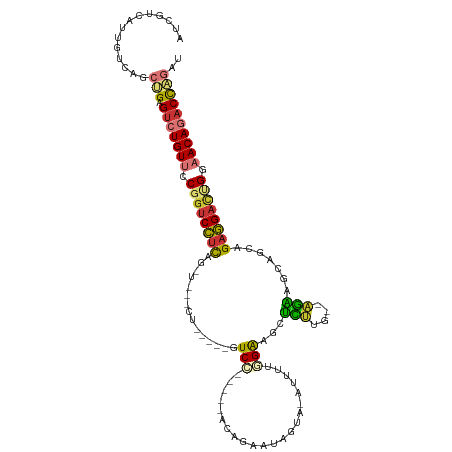
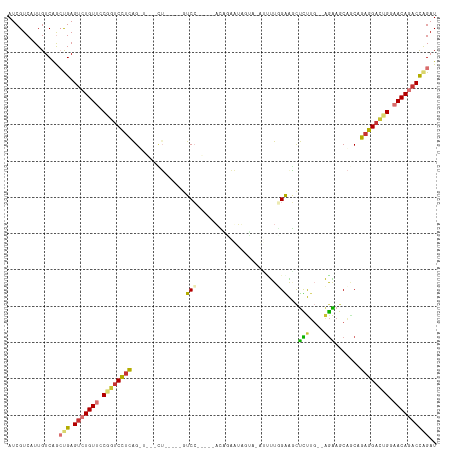
| Location | 9,945,627 – 9,945,737 |
|---|---|
| Length | 110 |
| Sequences | 8 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 65.82 |
| Shannon entropy | 0.66844 |
| G+C content | 0.47054 |
| Mean single sequence MFE | -37.64 |
| Consensus MFE | -14.02 |
| Energy contribution | -14.36 |
| Covariance contribution | 0.34 |
| Combinations/Pair | 1.35 |
| Mean z-score | -3.05 |
| Structure conservation index | 0.37 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.50 |
| SVM RNA-class probability | 0.998821 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 9945627 110 - 27905053 AUCUGGUCUGUUCCAGUCCUCUGCUGCUUCU--CAAGAGCUCCAAAAAAUUUCAAUUCAGU-UAUCGGAG-----AGUUAGACUGAGGACCGCAACAGACUCAGCUGACAAUGACGAU ..((((((((((.(.((((((..(((.((((--(...((((.................)))-)....)))-----)).)))...)))))).).))))))).))).............. ( -32.43, z-score = -1.52, R) >dp4.chr2 17352700 118 + 30794189 AUCUGGUCUGUUCCCGUCUUCAGGCGUUCCCAAAAGAAAACUCGAGACUUUACUAAACUCUUUUUUCGAUUUCUUUUGGAAGCUAAAGACCGGCACAGACUCGUUUGACAAUGACGAU .....((((((.((.(((((..(((....((((((((((..((((((................))))))))))))))))..))).))))).)).))))))(((((.......))))). ( -37.89, z-score = -3.80, R) >droPer1.super_0 7007882 116 - 11822988 AUCUGGUCUGUUCCCGUCUUCAGCCGUUCCCAAAAGAAAACUCGAGACUUUACUAAACUCU--UUGCGAUUUCUUUUGGAAGCUAAAGACCGGCACAGACUCGUUUGACAAUGACGAU .....((((((.((.(((((.(((.....((((((((((.(.(((((.((.....)).)).--))).).))))))))))..))).))))).)).))))))(((((.......))))). ( -35.70, z-score = -3.51, R) >droAna3.scaffold_13340 3178125 99 + 23697760 AUCCAGUCUGUUUCGCUCCUCUGCUGUUCUU--CUGAGGUCCCAAAAAA--AGAAUUCUAU-----GGAC-----AG-----CCGAGGACCGCAACAGGCUCGGCCGACAAUGACGAU ..((((((((((.((.(((((.(((((((..--..(((.((........--.)).)))...-----))))-----))-----).))))).)).)))))))).)).............. ( -35.10, z-score = -2.92, R) >droEre2.scaffold_4770 9494440 115 - 17746568 AUCUGGUGUGUUCCAAUCCUCCGCUGCUUCU--CAAGAGCUCCCAAGAU-UUCAAUUGAGUAUCGGGGACAUCUGAGGUCAACUGAGGACCGGAACAGACUCAGCUGACAAUGACGAU ..(((((.((((((..(((((.(.((..(((--((.((..((((..(((-(((....))).))).))))..))))))).)).).)))))..)))))).)).))).............. ( -38.40, z-score = -1.52, R) >droYak2.chr3R 12348906 116 + 28832112 AUCUGGUCUGUUCCAGUCCUCCGUUGCUUCU--CAAGAGCUUCAAAGAUUUCGAUAGAGUUUUUGGGGACUUCUGAGCUCAACUGGGGACCGGAACAGACUCAGCUGACAAUGACGAU ..((((((((((((.((((((.((((...((--((..((((((((((((((.....)))))))))))).))..))))..)))).)))))).))))))))).))).............. ( -51.70, z-score = -4.96, R) >droSec1.super_0 9049959 95 + 21120651 AUCUGGUCUGUUCCAGUCCUCUGUUGCUUCU--CAAGAGCUUCCAA-----GAUUUU-UGA-----GGAC-----AA-----CUGAGGACCGCAACAGACUCAGCUGACAAUGACGAU ..((((((((((.(.((((((.((((.((((--((((((((....)-----).))))-)))-----))))-----))-----).)))))).).))))))).))).............. ( -38.50, z-score = -4.22, R) >droSim1.chr3R 16033749 96 + 27517382 AUCUGGUUUGUUCCAGUCCUCUGUUGCUUCU--CAAGAGCUCCCAA-----GUUUUUCUGA-----GGAC-----AA-----CUGAGGUCCGCAACAGACUCAGCUGACAAUGACGAU ..((((((((((.(.(.((((.((((((((.--.(((((((....)-----))))))..))-----)).)-----))-----).)))).).).))))))).))).............. ( -31.40, z-score = -1.96, R) >consensus AUCUGGUCUGUUCCAGUCCUCUGCUGCUUCU__CAAGAGCUCCAAAAAU_UACAAUUCUGU_____GGAC_____AG___A_CUGAGGACCGCAACAGACUCAGCUGACAAUGACGAU ..((((((((((.(.((((((...............................................................)))))).).))))))).))).............. (-14.02 = -14.36 + 0.34)

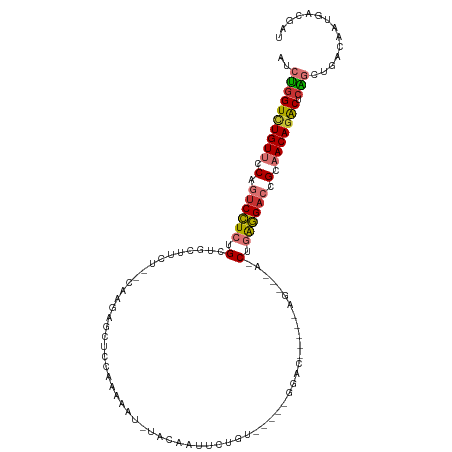
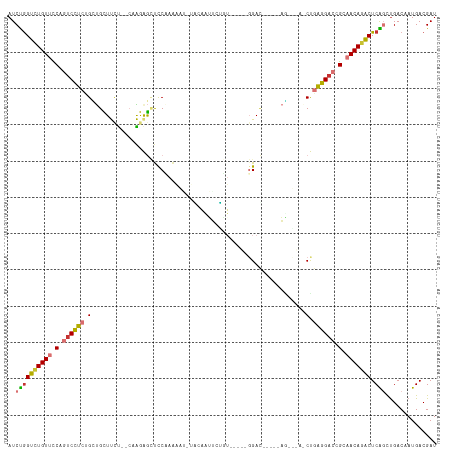
| Location | 9,945,632 – 9,945,742 |
|---|---|
| Length | 110 |
| Sequences | 8 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 67.73 |
| Shannon entropy | 0.62861 |
| G+C content | 0.47168 |
| Mean single sequence MFE | -41.70 |
| Consensus MFE | -22.23 |
| Energy contribution | -22.37 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.37 |
| Mean z-score | -4.05 |
| Structure conservation index | 0.53 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 5.21 |
| SVM RNA-class probability | 0.999956 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 9945632 110 + 27905053 CAUUGUCAGCUGAGUCUGUUGCGGUCCUCAGUCUAACUCUCCGAUAACUGAA---------UUGAAAUUUUUUGGAGCUCUU-GAGAAGCAGCAGAGGACUGGAACAGACCAGAUGAUGC ....((((.(((.(((((((.((((((((..........(((((.....(((---------(....)))).)))))(((((.-....)).))).)))))))).)))))))))).)))).. ( -40.60, z-score = -3.15, R) >dp4.chr2 17352705 118 - 30794189 CAUUGUCAAACGAGUCUGUGCCGGUCUUUAGCU--UCCAAAAGAAAUCGAAAAAAGAGUUUAGUAAAGUCUCGAGUUUUCUUUUGGGAACGCCUGAAGACGGGAACAGACCAGAUAAUGC (((((((....(.((((((.((.((((((((((--(((((((((((((((.....((.((.....)).))))))..))))))))))))..).)))))))).)).))))))).))))))). ( -42.90, z-score = -4.83, R) >droPer1.super_0 7007887 116 + 11822988 CAUUGUCAAACGAGUCUGUGCCGGUCUUUAGCU--UCCAAAAGAAAUCGCAAA--GAGUUUAGUAAAGUCUCGAGUUUUCUUUUGGGAACGGCUGAAGACGGGAACAGACCAGAUAAUGC (((((((....(.((((((.((.((((((((((--((((((((((((((...(--((.((.....)).))))))..)))))))))))...)))))))))).)).))))))).))))))). ( -47.10, z-score = -5.83, R) >droAna3.scaffold_13340 3178130 99 - 23697760 CAUUGUCGGCCGAGCCUGUUGCGGUCCUCGGCU--GUCCAUAGA-----------------AUUCUUUUUUUGGGACCUCA--GAAGAACAGCAGAGGAGCGAAACAGACUGGAUAAUGC ((((((((((...)))((((.((.(((((.(((--((....((.-----------------...))((((((((....)))--)))))))))).))))).)).)))).....))))))). ( -34.40, z-score = -1.93, R) >droEre2.scaffold_4770 9494445 115 + 17746568 CAUUGUCAGCUGAGUCUGUUCCGGUCCUCAGUU--GACCUCAGAUGUCCCCGAU-A-CUCAAUUGAAAUCUUGGGAGCUCUU-GAGAAGCAGCGGAGGAUUGGAACACACCAGAUAAUGC (((((((...((.((.(((((((((((((.(((--(..(((((..(..(((((.-.-.((....))....)))))..)..))-)))...)))).))))))))))))).))))))))))). ( -47.70, z-score = -4.36, R) >droYak2.chr3R 12348911 116 - 28832112 CAUUGUCAGCUGAGUCUGUUCCGGUCCCCAGUU--GAGCUCAGAAGUCCCCAAA-AACUCUAUCGAAAUCUUUGAAGCUCUU-GAGAAGCAACGGAGGACUGGAACAGACCAGAUAAUGC (((((((...((.((((((((((((((((.(((--(..((((..(((...((((-...((....))....))))..)))..)-)))...)))))).))))))))))))))))))))))). ( -48.00, z-score = -5.53, R) >droSec1.super_0 9049964 95 - 21120651 CAUUGUCAGCUGAGUCUGUUGCGGUCCUCAGUU--GUCCUCAAA----------------------AAUCUUGGAAGCUCUU-GAGAAGCAACAGAGGACUGGAACAGACCAGAUAAUGC (((((((...((.(((((((.((((((((.(((--((.(((((.----------------------..............))-)))..))))).)))))))).)))))))))))))))). ( -40.56, z-score = -4.66, R) >droSim1.chr3R 16033754 96 - 27517382 CAUUGUCAGCUGAGUCUGUUGCGGACCUCAGUU--GUCCUCAGA---------------------AAAACUUGGGAGCUCUU-GAGAAGCAACAGAGGACUGGAACAAACCAGAUAAUGC ((((((((((((((((((...)))).)))))))--((((((...---------------------....((..((....)).-.))........))))))(((......)))))))))). ( -32.36, z-score = -2.12, R) >consensus CAUUGUCAGCUGAGUCUGUUCCGGUCCUCAGUU__GCCCUCAGA__UC___A_________AUUAAAAUCUUGGGAGCUCUU_GAGAAGCAGCAGAGGACUGGAACAGACCAGAUAAUGC (((((((...((.(((((((.((((((((.(((.........................................................))).)))))))).)))))))))))))))). (-22.23 = -22.37 + 0.14)

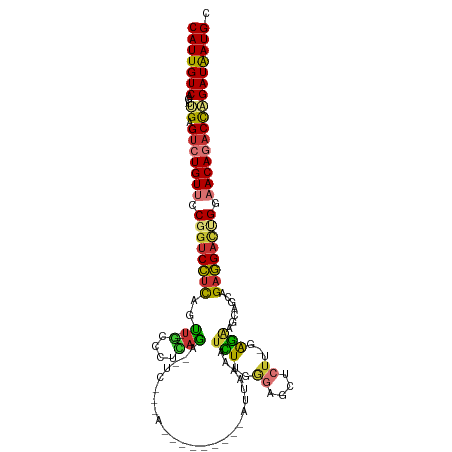
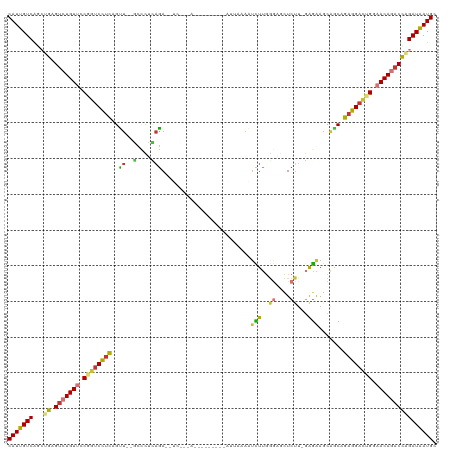
| Location | 9,945,632 – 9,945,742 |
|---|---|
| Length | 110 |
| Sequences | 8 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 67.73 |
| Shannon entropy | 0.62861 |
| G+C content | 0.47168 |
| Mean single sequence MFE | -38.16 |
| Consensus MFE | -16.16 |
| Energy contribution | -17.02 |
| Covariance contribution | 0.86 |
| Combinations/Pair | 1.38 |
| Mean z-score | -3.70 |
| Structure conservation index | 0.42 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 4.46 |
| SVM RNA-class probability | 0.999813 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 9945632 110 - 27905053 GCAUCAUCUGGUCUGUUCCAGUCCUCUGCUGCUUCUC-AAGAGCUCCAAAAAAUUUCAA---------UUCAGUUAUCGGAGAGUUAGACUGAGGACCGCAACAGACUCAGCUGACAAUG ...(((.((((((((((.(.((((((..(((.(((((-...((((..............---------...))))....))))).)))...)))))).).))))))).))).)))..... ( -35.63, z-score = -2.83, R) >dp4.chr2 17352705 118 + 30794189 GCAUUAUCUGGUCUGUUCCCGUCUUCAGGCGUUCCCAAAAGAAAACUCGAGACUUUACUAAACUCUUUUUUCGAUUUCUUUUGGA--AGCUAAAGACCGGCACAGACUCGUUUGACAAUG .((((.((.(((((((.((.(((((..(((....((((((((((..((((((................)))))))))))))))).--.))).))))).)).))))))).....)).)))) ( -36.39, z-score = -4.05, R) >droPer1.super_0 7007887 116 - 11822988 GCAUUAUCUGGUCUGUUCCCGUCUUCAGCCGUUCCCAAAAGAAAACUCGAGACUUUACUAAACUC--UUUGCGAUUUCUUUUGGA--AGCUAAAGACCGGCACAGACUCGUUUGACAAUG .((((.((.(((((((.((.(((((.(((.....((((((((((.(.(((((.((.....)).))--.))).).)))))))))).--.))).))))).)).))))))).....)).)))) ( -34.20, z-score = -3.65, R) >droAna3.scaffold_13340 3178130 99 + 23697760 GCAUUAUCCAGUCUGUUUCGCUCCUCUGCUGUUCUUC--UGAGGUCCCAAAAAAAGAAU-----------------UCUAUGGAC--AGCCGAGGACCGCAACAGGCUCGGCCGACAAUG .......((((((((((.((.(((((.(((((((...--.(((.((.........)).)-----------------))...))))--))).))))).)).)))))))).))......... ( -35.10, z-score = -3.49, R) >droEre2.scaffold_4770 9494445 115 - 17746568 GCAUUAUCUGGUGUGUUCCAAUCCUCCGCUGCUUCUC-AAGAGCUCCCAAGAUUUCAAUUGAG-U-AUCGGGGACAUCUGAGGUC--AACUGAGGACCGGAACAGACUCAGCUGACAAUG ...(((.(((((.((((((..(((((.(.((..((((-(.((..((((..((((((....)))-.-))).))))..))))))).)--).).)))))..)))))).)).))).)))..... ( -39.40, z-score = -2.19, R) >droYak2.chr3R 12348911 116 + 28832112 GCAUUAUCUGGUCUGUUCCAGUCCUCCGUUGCUUCUC-AAGAGCUUCAAAGAUUUCGAUAGAGUU-UUUGGGGACUUCUGAGCUC--AACUGGGGACCGGAACAGACUCAGCUGACAAUG ...(((.((((((((((((.((((((.((((...(((-(..((((((((((((((.....)))))-))))))).))..))))..)--))).)))))).))))))))).))).)))..... ( -52.70, z-score = -5.70, R) >droSec1.super_0 9049964 95 + 21120651 GCAUUAUCUGGUCUGUUCCAGUCCUCUGUUGCUUCUC-AAGAGCUUCCAAGAUU----------------------UUUGAGGAC--AACUGAGGACCGCAACAGACUCAGCUGACAAUG ...(((.((((((((((.(.((((((.((((.(((((-(((((((....)).))----------------------)))))))))--))).)))))).).))))))).))).)))..... ( -39.50, z-score = -4.93, R) >droSim1.chr3R 16033754 96 + 27517382 GCAUUAUCUGGUUUGUUCCAGUCCUCUGUUGCUUCUC-AAGAGCUCCCAAGUUUU---------------------UCUGAGGAC--AACUGAGGUCCGCAACAGACUCAGCUGACAAUG ...(((.((((((((((.(.(.((((.((((((((..-(((((((....))))))---------------------)..)))).)--))).)))).).).))))))).))).)))..... ( -32.40, z-score = -2.80, R) >consensus GCAUUAUCUGGUCUGUUCCAGUCCUCUGCUGCUUCUC_AAGAGCUCCCAAGAUUUUAAU_________U___GA__UCUGAGGAC__AACUGAGGACCGCAACAGACUCAGCUGACAAUG ...(((.((((((((((.(.((((((.(((.........(((..................................)))........))).)))))).).))))))).))).)))..... (-16.16 = -17.02 + 0.86)


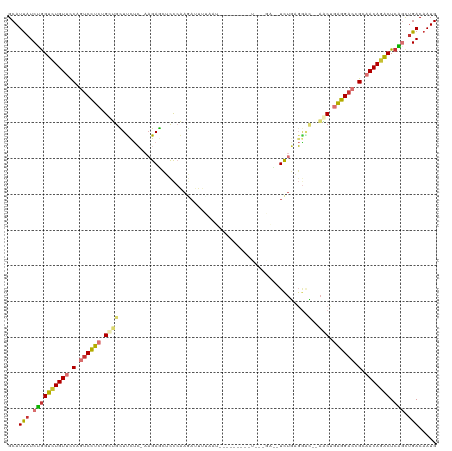
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:14:15 2011