
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 9,454,172 – 9,454,289 |
| Length | 117 |
| Max. P | 0.657223 |

| Location | 9,454,172 – 9,454,289 |
|---|---|
| Length | 117 |
| Sequences | 12 |
| Columns | 126 |
| Reading direction | reverse |
| Mean pairwise identity | 79.53 |
| Shannon entropy | 0.44604 |
| G+C content | 0.40640 |
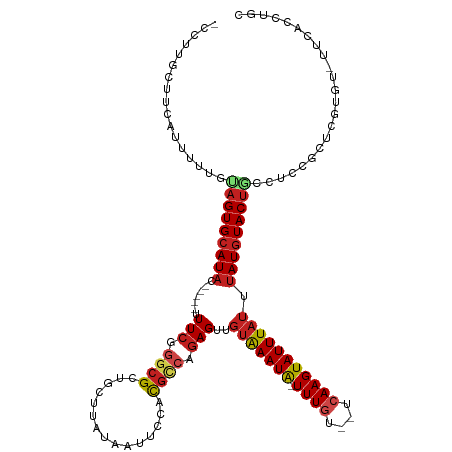
| Mean single sequence MFE | -26.93 |
| Consensus MFE | -14.37 |
| Energy contribution | -14.74 |
| Covariance contribution | 0.38 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.53 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.35 |
| SVM RNA-class probability | 0.657223 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
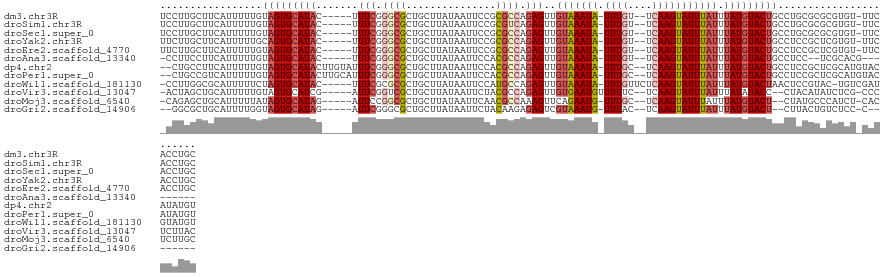
>dm3.chr3R 9454172 117 - 27905053 UCCUUGCUUCAUUUUUGUAGUGCAUAC-----UUUCGGGCGCUGCUUAUAAUUCCGCGCCAGAGUUGUAAAUA-UUUGU--UCAAGUAUUUAUUUAUGUACUGCCUGCGCGCGUGU-UUCACCUGC ................((((((((((.-----.(((.(((((.............))))).)))..(((((((-(((..--..)))))))))).))))))))))....(((.(((.-..))).))) ( -30.02, z-score = -1.63, R) >droSim1.chr3R 15534942 117 + 27517382 UCCUUGCUUCAUUUUUGUAGUGCAUAC-----UUUCGGGCGCUGCUUAUAAUUCCGCGUCAGAGUUGUAAAUA-UUUGU--UCAAGUAUUUAUUUAUGUACUGCCUGCGCGCGUGU-UUCACCUGC ................((((((((((.-----.(((.(((((.............))))).)))..(((((((-(((..--..)))))))))).))))))))))....(((.(((.-..))).))) ( -27.32, z-score = -0.94, R) >droSec1.super_0 8567609 117 + 21120651 UCCUUGCUUCAUUUUUGUAGUGCAUAC-----UUUCGGGCGCUGCUUAUAAUUCCGCGCCAGAGUUGUAAAUA-UUUGU--UCAAGUAUUUAUUUAUGUACUGCCUGCGCGCGUGU-UUCACCUGC ................((((((((((.-----.(((.(((((.............))))).)))..(((((((-(((..--..)))))))))).))))))))))....(((.(((.-..))).))) ( -30.02, z-score = -1.63, R) >droYak2.chr3R 13762974 117 - 28832112 UUCUUGCUUCAUUUUUGCAGUGCAUAC-----UUUCGGGCGCUGCUUAUAAUUCCGCGCCAGAGUUGUAAAUA-UUUGU--UCAAGUAUUUAUUUAUGUACUGCCUCCGCUCGUGU-UUCACCUGC ................((((((((((.-----.(((.(((((.............))))).)))..(((((((-(((..--..)))))))))).))))))))))....((..(((.-..)))..)) ( -31.12, z-score = -3.03, R) >droEre2.scaffold_4770 5565179 117 + 17746568 UUCUUGCUUCAUUUUUGUAGUGCAUAC-----UUUCGGGCGCUGCUUAUAAUUCCGCGCCAGAGUUGUAAAUA-UUUGU--UCAAGUAUUUAUUUAUGUACUGCCUCCGCUCGUGU-UUCACCUGC ................((((((((((.-----.(((.(((((.............))))).)))..(((((((-(((..--..)))))))))).))))))))))....((..(((.-..)))..)) ( -29.12, z-score = -2.70, R) >droAna3.scaffold_13340 2002143 106 - 23697760 -CCUUCCUUCAUUUUUGUAGUGCAUAC-----UUUCGGGCGCUGCUUAUAAUUCCACGCCAGAGUUGUAAAUA-UUUGU--UCAAGUAUUUAUUUAUGUACUGCCUCC--UCGCACG--------- -...............((((((((((.-----.(((.((((.((..........)))))).)))..(((((((-(((..--..)))))))))).))))))))))....--.......--------- ( -24.10, z-score = -2.87, R) >dp4.chr2 16892986 121 - 30794189 --CUGCCUUCAUUUUUGUAGUGCAUACUUGUAUUUCGGGCGCUGCUUAUAAUUCCACGCCAGAGUUGUAAAUA-UUUGC--UCAAGUAUUUAUUUAUGUACUGCCUCCGCUCGCAUGUACAUAUGU --..............((((((((((...((((((.(((((....(((((((((.......)))))))))...-..)))--)))))))).....))))))))))........(((((....))))) ( -26.80, z-score = -0.84, R) >droPer1.super_0 8324808 121 + 11822988 --CUGCCGUCAUUUUUGUAGUGCAUACUUGCAUUUCGGGCGCUGCUUAUAAUUCCACGCCAGAGUUGUAAAUA-UUUGC--UCAAGUAUUUAUUUAUGUACUGCCUCCGCUCGCAUGUACAUAUGU --(((.(((.(((...(((((((...(.........).)))))))....)))...))).)))....(((((((-(((..--..)))))))))).((((((((((........))).)))))))... ( -27.60, z-score = -0.46, R) >droWil1.scaffold_181130 6535228 118 - 16660200 -CCUUGGCGCAUUUUUCUAGUGCAUAC-----UUUCGCGCGCUGCUUAUAAUUCCAUGCCAGAGUUGUAAAUA-UUUGUUCUCAAGUAUUUAUUUAUGUACUAACUCCGUAC-UGUCGAUGUAUGU -...(((((((((.....)))))....-----....((.....))............))))((((((((((((-((((....))))))))))).........)))))(((((-.......))))). ( -25.40, z-score = -1.42, R) >droVir3.scaffold_13047 9228526 115 - 19223366 -ACUAGCUGCAUUUUUGUAGUGCAUCG-----AUUCGGUCGCUGCUUAUAAUUCUACGCCAGAGUUGUGAAUGUUUUUC--UCAAGUAUUUAUUUAUAUACC--CUACAUAUCUCG-CCCUCUUAC -....((((((....))))))(((.((-----((...)))).)))((((((((((.....)))))))))).........--....((((........)))).--............-......... ( -19.80, z-score = -0.71, R) >droMoj3.scaffold_6540 2742462 114 + 34148556 -CAGAGCUGCAUUUUUAUAGUGCAUAG-----AUUCCGGCGCUGCUUAUAAUUCAACGCCAAAGUUCAGAAUG-UUUGC--UCAAGUAUUUAUUUAUGUACU--CUAUGCCCAUCU-CACUCUUGC -..(((..((((......(((((((((-----((....((...((..(((.((((((......)))..)))))-)..))--....))....)))))))))))--..))))....))-)........ ( -22.70, z-score = -0.30, R) >droGri2.scaffold_14906 10154140 105 + 14172833 --GGCGCUGCAUUUUGGUAGUGCAUAG-----AUUCGGGCGCUGCUUAUAAUUCUACAAGAGAGUCGUAAAUG-UUUAC--UCAAGUAUUUAUUUAUGUACU--CUUACUGUCUCC-C-------- --.(((((((......))))))).(((-----(...((((...)))).....))))...((((((((((((((-..(((--....)))..)))))))).)))--))).........-.-------- ( -29.20, z-score = -2.46, R) >consensus _CCUUGCUUCAUUUUUGUAGUGCAUAC_____UUUCGGGCGCUGCUUAUAAUUCCACGCCAGAGUUGUAAAUA_UUUGU__UCAAGUAUUUAUUUAUGUACUGCCUCCGCUCGUGU_UUCACCUGC ................(((((((...............)))))))(((((((((.......)))))))))..............(((((........)))))........................ (-14.37 = -14.74 + 0.38)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:12:40 2011