
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 8,451,482 – 8,451,602 |
| Length | 120 |
| Max. P | 0.794190 |

| Location | 8,451,482 – 8,451,602 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.75 |
| Shannon entropy | 0.25262 |
| G+C content | 0.53167 |
| Mean single sequence MFE | -36.77 |
| Consensus MFE | -29.40 |
| Energy contribution | -28.17 |
| Covariance contribution | -1.23 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.80 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.794190 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
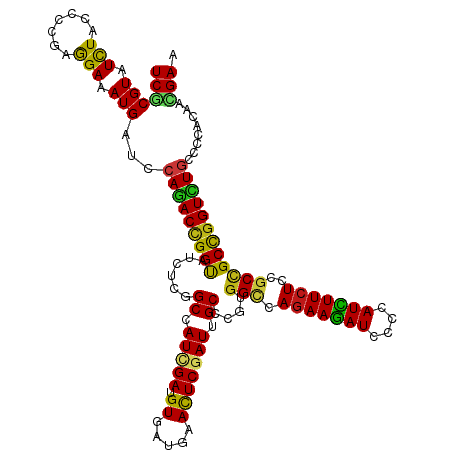
>dm3.chr3R 8451482 120 - 27905053 UCGCGUAUCUACCCCGAGGAGAUGAUCCAGACCGGUAUCUCGGCCAUCGAUGUGAUGAACUCGAUUGCCCGUGGCCAGAAGAUCCCCAUCUUCUCCGCCGCCGGUCUGCCCCACAACGAA (((..(((((.((....)))))))...(((((((((.....(((.(((((.((.....))))))).)))...(((.(((((((....)))))))..))))))))))))........))). ( -44.60, z-score = -3.20, R) >droSim1.chr3R 14543797 120 + 27517382 UCGCGUAUCUACCCCGAGGAGAUGAUCCAGACCGGUAUCUCGGCCAUCGAUGUGAUGAACUCGAUUGCCCGUGGCCAGAAGAUCCCCAUCUUCUCCGCCGCCGGUCUGCCCCACAACGAA (((..(((((.((....)))))))...(((((((((.....(((.(((((.((.....))))))).)))...(((.(((((((....)))))))..))))))))))))........))). ( -44.60, z-score = -3.20, R) >droSec1.super_0 7584610 120 + 21120651 UCGCGUAUCUACCCCGAGGAGAUGAUCCAGACCGGUAUCUCGGCCAUCGAUGUGAUGAACUCGAUUGCCCGUGGCCAGAAGAUCCCCAUCUUCUCCGCCGCCGGUUUGCCCCACAACGAA (((..(((((.((....)))))))...(((((((((.....(((.(((((.((.....))))))).)))...(((.(((((((....)))))))..))))))))))))........))). ( -42.00, z-score = -2.62, R) >droYak2.chr3R 12738058 120 - 28832112 UCGCGUAUCUACCCCGAGGAGAUGAUCCAGACCGGUAUCUCGGCCAUCGAUGUGAUGAACUCGAUUGCCCGUGGUCAGAAGAUCCCCAUCUUCUCCGCCGCCGGUCUGCCCCACAACGAA (((..(((((.((....)))))))...(((((((((.....(((.(((((.((.....))))))).))).((((..(((((((....))))))))))).)))))))))........))). ( -44.50, z-score = -3.04, R) >droEre2.scaffold_4770 4545964 120 + 17746568 UCGCGUAUCUACCCCGAGGAGAUGAUCCAGACCGGUAUCUCGGCCAUCGAUGUGAUGAACUCGAUUGCCCGUGGCCAGAAGAUCCCCAUCUUCUCCGCCGCCGGUCUGCCCCACAACGAA (((..(((((.((....)))))))...(((((((((.....(((.(((((.((.....))))))).)))...(((.(((((((....)))))))..))))))))))))........))). ( -44.60, z-score = -3.20, R) >droAna3.scaffold_12911 3387130 120 - 5364042 UCCCGUAUUUACCCCGAGGAAAUGAUCCAAACCGGUAUCUCGGCCAUUGAUGUGAUGAACUCCAUUGCCCGUGGUCAGAAGAUCCCCAUCUUCUCCGCCGCUGGUCUGCCCCACAACGAA ..(((....((((....(((.....))).....))))...)))...(((.(((((((.....))).(((.(((((.(((((((....)))))))..))))).)))......)))).))). ( -29.60, z-score = -0.54, R) >dp4.chr2 2559790 120 - 30794189 UCUCGUAUCUACCCCGAGGAAAUGAUCCAGACUGGCAUCUCUGCCAUCGAUGUGAUGAACUCGAUUGCCCGUGGCCAGAAGAUCCCCAUUUUCUCGGCUGCCGGUCUGCCCCACAACGAA ..((((.....((....))........(((((((((......((.(((((.((.....))))))).))....(((((((((((....))))))).))))))))))))).......)))). ( -38.50, z-score = -2.30, R) >droPer1.super_7 2730589 120 - 4445127 UCUCGUAUCUACCCCGAGGAAAUGAUCCAGACUGGCAUCUCUGCCAUCGAUGUGAUGAACUCGAUUGCCCGUGGCCAGAAGAUCCCCAUUUUCUCGGCUGCCGGUCUGCCCCACAACGAA ..((((.....((....))........(((((((((......((.(((((.((.....))))))).))....(((((((((((....))))))).))))))))))))).......)))). ( -38.50, z-score = -2.30, R) >droWil1.scaffold_181130 2294565 120 - 16660200 UCACGUAUCUAUCCAGAGGAAAUGAUUCAAACCGGUAUUUCGGCCAUCGAUGUGAUGAACUCGAUUGCCCGUGGCCAGAAGAUUCCUAUUUUCUCAGCUGCUGGUUUACCGCACAAUGAA (((.(.(((..(((...)))...))).)....(((((..(((((.(((((.((.....))))))).))).(..((.(((((((....)))))))..))..).))..))))).....))). ( -31.70, z-score = -0.91, R) >droVir3.scaffold_12822 5216 120 + 4096053 UCGCGUAUCUAUCCCGAGGAAAUGAUCCAGACUGGUAUCUCUGCCAUUGAUGUGAUGAACUCCAUUGCCCGUGGUCAGAAGAUUCCAAUCUUCUCAGCUGCUGGUCUGCCACAUAACGAA ...(((.....(((...))).(((...(((((..(((.((..(((((....(..(((.....)))..)..))))).(((((((....))))))).)).)))..)))))...))).))).. ( -30.90, z-score = -0.50, R) >droMoj3.scaffold_6540 16800058 120 + 34148556 UCGCGUAUCUAUCCCGAAGAAAUGAUCCAGACUGGUAUCUCUGCUAUUGAUGUGAUGAACUCGAUUGCUCGUGGUCAGAAGAUUCCCAUCUUCUCUGCUGCUGGUCUGCCGCACAAUGAA ..(((..(((.......))).......(((((..(((.....((.(((((.((.....))))))).))..(..(..(((((((....))))))))..))))..))))).)))........ ( -30.70, z-score = -0.66, R) >droGri2.scaffold_14906 12553553 120 + 14172833 UCGCGUAUCUAUCCCGAGGAAAUGAUCCAGACCGGUAUCUCUGCCAUUGAUGUGAUGAACUCGAUUGCCCGUGGUCAGAAGAUUCCCAUCUUCUCGGCGGCUGGUCUGCCGCACAACGAA ..(((.(((..(((...)))...))).((((((((.......((.(((((.((.....))))))).))((((.(..(((((((....)))))))).)))))))))))).)))........ ( -35.30, z-score = -0.18, R) >anoGam1.chr2R 34053261 120 + 62725911 UCGCGUAUCUACCCGGAGGAAAUGAUCCAGACCGGUAUCUCCGCCAUCGACGUGAUGAACUCGAUCGCCCGUGGUCAGAAGAUUCCGAUCUUCUCGGCUGCCGGUCUGCCGCACAAUGAA ..(((.........(((........)))((((((((......((.(((((.((.....))))))).))....(((((((((((....))))))).))))))))))))..)))........ ( -40.50, z-score = -0.88, R) >apiMel3.GroupUn 15322031 120 - 399230636 UCACGUAUCUAUCCUAAAGAAAUGAUUCAAACAGGUAUCUCGGCUAUUGAUGUUAUGAAUUCUAUUGCUCGUGGACAAAAAAUUCCUAUUUUCUCAGCUGCUGGUUUACCACACAAUGAG ((((((.(((.......))).))).........((((..(((((..((((((((((((..........)))).)))).(((((....))))).))))..)))))..))))......))). ( -19.30, z-score = 0.35, R) >triCas2.ChLG9 3497525 120 - 15222296 UCGCGUAUCUACCCUGAAGAAAUGAUCCAGACCGGUAUUUCUGCCAUUGAUGUUAUGAACUCGAUCGCUCGAGGGCAGAAAAUCCCGAUUUUCUCUGCCGCCGGUCUGCCGCACAACGAA ..(((..(((.......))).......(((((((((.......................(((((....)))))((((((((((....))))..))))))))))))))).)))........ ( -36.20, z-score = -1.99, R) >consensus UCGCGUAUCUACCCCGAGGAAAUGAUCCAGACCGGUAUCUCGGCCAUCGAUGUGAUGAACUCGAUUGCCCGUGGCCAGAAGAUCCCCAUCUUCUCCGCCGCCGGUCUGCCCCACAACGAA ((((((.(((.......))).)))...(((((((((......((.(((((.((.....))))))).))....(((.(((((((....)))))))..))))))))))))........))). (-29.40 = -28.17 + -1.23)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:09:39 2011