
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 6,946,778 – 6,946,872 |
| Length | 94 |
| Max. P | 0.762617 |

| Location | 6,946,778 – 6,946,872 |
|---|---|
| Length | 94 |
| Sequences | 12 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 79.97 |
| Shannon entropy | 0.38628 |
| G+C content | 0.47840 |
| Mean single sequence MFE | -29.47 |
| Consensus MFE | -18.11 |
| Energy contribution | -17.58 |
| Covariance contribution | -0.53 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.61 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.61 |
| SVM RNA-class probability | 0.762617 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 6946778 94 + 27905053 UGAUAGCG------UUGGCAUUAGUCUCCAGUG------AACUGGCACAUAAAAGCACAGUUAUGGACUGAUUGUCGGG-------UCCGCUAUCCGACGUACGUGUAAAUUG---- .(((((((------((((((((((((.((((..------..))))..(((((........))))))))))).)))))).-------..)))))))..................---- ( -28.40, z-score = -1.54, R) >droSim1.chr3R 13097111 94 - 27517382 UGAUAGCG------AUGGCAUUAGUCUCCAAUG------AACUGGCACAUAAAAGCACAGUUAUGGACUGAUUGUCGGG-------ACCGCUAUCCGACGUACGUGUAAAUUG---- .(((((((------.((((((((((((......------(((((((........)).)))))..))))))).)))))..-------..)))))))..................---- ( -28.00, z-score = -1.98, R) >droSec1.super_0 6103413 94 - 21120651 UGAUAGCG------AUGGCAUUAGUCUCCAGUG------AACUGGCACAUAAAAGCACAGUUAUGGACUGAUUGUCGGG-------ACCGCUAUCCGACGUACGUGUAAAUUG---- .(((((((------.(((((((((((.((((..------..))))..(((((........))))))))))).)))))..-------..)))))))..................---- ( -28.70, z-score = -1.88, R) >droYak2.chr3R 10977645 94 + 28832112 UGAUAGCG------AUGGCAUUAGUCUCCAGUG------AACUGGCACAUAAAAGCACAGCUAUGGACUGAUUGUCGGG-------UCCGCUAUCCGACGUACGUGUAAAUUG---- .(((((((------.((((((((((((((((..------..))))........(((...)))..))))))).)))))..-------..)))))))..................---- ( -27.80, z-score = -1.27, R) >droEre2.scaffold_4770 3076983 94 - 17746568 UGAUAGCG------AUGGCAUUAGUCUCCAGUG------AACUGGCACAUAAAAGCACAGCUAUGGACUGAUUGUCGGG-------UCUGCUAUCCAACGUACGUGUAAAUUG---- .(((((((------.((((((((((((((((..------..))))........(((...)))..))))))).)))))..-------..)))))))..................---- ( -26.10, z-score = -0.95, R) >droAna3.scaffold_13340 11511310 94 - 23697760 AGAUAGCG------AUGGCAUUAGUCUCCAGUG------AACUGACACAUAAAAGCACAGUUAUGGACUGAUUGUCGGG-------GCCGCUAUCCGGCGUACGUGUCAAUUG---- .(((((((------.(((((((((((..(((..------..)))...(((((........))))))))))).)))))..-------..))))))).((((....)))).....---- ( -28.70, z-score = -1.30, R) >dp4.chr2 2355188 107 - 30794189 CGAUAGUGCGCGAUGUGGCAUUAGUCUCCAGUG------AACUGGCACAUAAAAGCACAGUUAUGGACUGAUUGUCGGGCUGCCCCACUGCUAUCCGACGUACGUGUAAAUUG---- ((((..((((((((((((((((((((.((((..------..))))..(((((........))))))))))).))))(((..((......))..))).)))).)))))).))))---- ( -33.20, z-score = -0.98, R) >droPer1.super_6 2370844 107 - 6141320 CGAUAGUGCGCGAUGUGGCAUUAGUCUCCAGUG------AACUGGCACAUAAAAGCACAGUUAUGGACUGAUUGUCGGGCUGCCCCACUGCUAUCCGACGUACGUGUAAAUUG---- ((((..((((((((((((((((((((.((((..------..))))..(((((........))))))))))).))))(((..((......))..))).)))).)))))).))))---- ( -33.20, z-score = -0.98, R) >droWil1.scaffold_181130 7683348 100 + 16660200 UGAUAGCGU----CGUGGCAUUAGUCUCCAGUGUGCGCGAACUGGCAUAUAAAAGCCCAGUUAUAGACUGAUUGUCGGU--------CCACCAAC-AAUGUAAGUGUAAAUUG---- .....((((----.(((((((((((((...((....)).(((((((........).))))))..))))))).))))((.--------...)).))-.))))............---- ( -22.50, z-score = 0.05, R) >droMoj3.scaffold_6540 21488838 95 + 34148556 UGAUAGCG------CCAGCAUUAGUCUCCAGUG------AGCUGGCACAUAAAAGCGCAGUUAUAGACUGAUUGUUGG----------CGCACUCACACACACACACACAUACUCUA (((..(((------(((((((((((((......------(((((((........)).)))))..))))))).))))))----------)))..)))..................... ( -31.90, z-score = -3.45, R) >droGri2.scaffold_14906 3817610 88 - 14172833 UGAUAGCG------CCGGCAUUAGUCUCCAGUG------AGCUGGCACAUAAAUGCACAGUUAUGGACUGAUUGUCGG----------CGCAC-CACACUCACACACAAUU------ .....(((------((((((((((..((((...------((((((((......))).))))).)))))))).))))))----------)))..-.................------ ( -34.20, z-score = -4.19, R) >droVir3.scaffold_13047 14299677 88 - 19223366 UGAUAGCG------UCGGCAUUAGUCUCCAGUG------AGCUGGCAUAUAAAAGCGCAGUUAUGGACUGAUUGUCGG----------CGCGC-CAAACAUGCAUACACUA------ .....(((------((((((((((..((((...------(((((((........)).))))).)))))))).))))))----------)))((-.......))........------ ( -30.90, z-score = -2.03, R) >consensus UGAUAGCG______AUGGCAUUAGUCUCCAGUG______AACUGGCACAUAAAAGCACAGUUAUGGACUGAUUGUCGGG________CCGCUAUCCGACGUACGUGUAAAUUG____ .....(((.......(((((((((((.(((((........)))))..(((((........))))))))))).)))))...........))).......................... (-18.11 = -17.58 + -0.53)

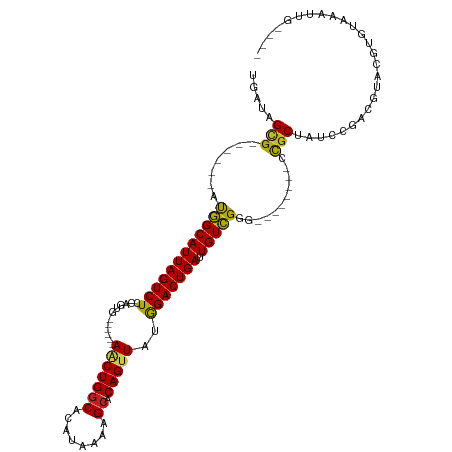

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:05:18 2011