| Sequence ID | dm3.chr2L |
|---|---|
| Location | 6,920,716 – 6,920,856 |
| Length | 140 |
| Max. P | 0.968118 |

| Location | 6,920,716 – 6,920,856 |
|---|---|
| Length | 140 |
| Sequences | 12 |
| Columns | 142 |
| Reading direction | forward |
| Mean pairwise identity | 97.18 |
| Shannon entropy | 0.05683 |
| G+C content | 0.28156 |
| Mean single sequence MFE | -25.85 |
| Consensus MFE | -24.22 |
| Energy contribution | -24.25 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.94 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.79 |
| SVM RNA-class probability | 0.968118 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
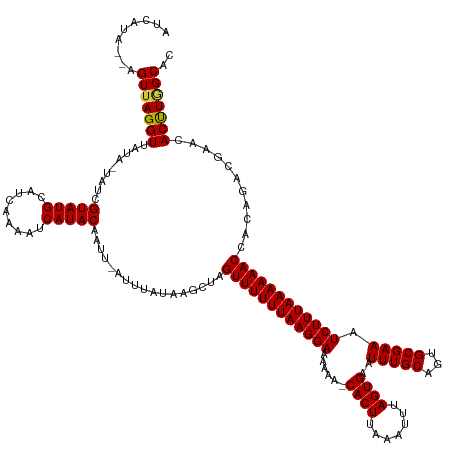
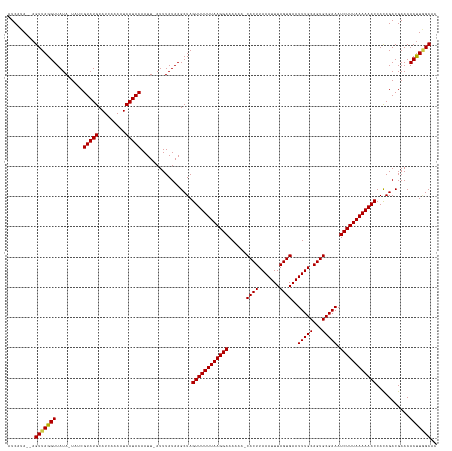
>dm3.chr2L 6920716 140 + 23011544 AUCAUAUAAGUUAGGUUAUAAUAUCGUAUGCAUCAAAAUCAUACAAUU-AUUUAUAAGCUAGUUUUUUAAGGAAAAAA-CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUGGCAC ......((((((((.((((((.((.(((((.((....))))))).)).-..)))))).)))((((((((((((.....-((((.......))))..(((((...))))).))))))))))))...........))))).... ( -26.60, z-score = -2.47, R) >droEre2.scaffold_4929 15835684 140 + 26641161 AUCAUAUAAGUUAGGUUAUAAUAUCGUAUGCAUCAAAAUCAUACAAUU-AUUUAUAAGCUAGUUUUUUAAGGAAAAAA-CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUGGCAC ......((((((((.((((((.((.(((((.((....))))))).)).-..)))))).)))((((((((((((.....-((((.......))))..(((((...))))).))))))))))))...........))))).... ( -26.60, z-score = -2.47, R) >droYak2.chr2L 16335599 140 - 22324452 AUCAUAUAAGUUAGGUUAUAAUAUCGUAUGCAUCAAAAUCAUACAAUU-AUUUAUAAGCUAGUUUUUUAAGGAAAAAA-CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUGGCAC ......((((((((.((((((.((.(((((.((....))))))).)).-..)))))).)))((((((((((((.....-((((.......))))..(((((...))))).))))))))))))...........))))).... ( -26.60, z-score = -2.47, R) >droSec1.super_3 2454380 140 + 7220098 AUCAUAUAAGUUAGGUUAUAAUAUCGUAUGCAUCAAAAUCAUACAAUU-AUUUAUAAGCUAGUUUUUUAAGGAAAAAA-CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUUGCAC .((........(((.((((((.((.(((((.((....))))))).)).-..)))))).)))((((((((((((.....-((((.......))))..(((((...))))).)))))))))))).......))........... ( -24.90, z-score = -2.00, R) >droSim1.chr2L 6717155 140 + 22036055 AUCAUAUAAGUUAGGUUAUAAUAUCGUAUGCAUCAAAAUCAUACAAUU-AUUUAUAAGCUAGUUUUUUAAGGAAAAAA-CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUUGCAC .((........(((.((((((.((.(((((.((....))))))).)).-..)))))).)))((((((((((((.....-((((.......))))..(((((...))))).)))))))))))).......))........... ( -24.90, z-score = -2.00, R) >droWil1.scaffold_180772 6767219 139 + 8906247 AUCAUA--AGUUAGGUUAUA-UAUCGUAUGCAUCAAAAUCAUACAAUUUAUUUAUAAGCUAGUUUUUUAAGGAAAAAAACACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCAUAGACGAACACCUGGCAC ......--.(((((((....-....(((((.((....))))))).............((((((((((((((((......((((.......))))..(((((...))))).))))))))))))..))).)....))))))).. ( -28.20, z-score = -2.94, R) >droPer1.super_5 4915417 135 + 6813705 AUCAUA--AGUUAGGUUAUA-U--CGUAUGCAUCAAAAUCAUACAAUU-AUUUAUAAGCUAGUUUUUUAAGGAAAAAA-CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUGGCAC ......--.(((((((....-(--((((((.((....)))))))....-............((((((((((((.....-((((.......))))..(((((...))))).)))))))))))).......))..))))))).. ( -25.00, z-score = -2.05, R) >dp4.chr4_group5 417255 135 + 2436548 AUCAUA--AGUUAGGUUAUA-U--CGUAUGCAUCAAAAUCAUACAAUU-AUUUAUAAGCUAGUUUUUUAAGGAAAAAA-CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUGGCAC ......--.(((((((....-(--((((((.((....)))))))....-............((((((((((((.....-((((.......))))..(((((...))))).)))))))))))).......))..))))))).. ( -25.00, z-score = -2.05, R) >droAna3.scaffold_12916 8501522 135 + 16180835 AUCAUA--AGUUAGGUUAUA-U--CGUAUGCAUCAAAAUCAUACAAUU-AUUUAUAAGCUAGUUUUUUAAGGAAAAAA-CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUGGCAC ......--.(((((((....-(--((((((.((....)))))))....-............((((((((((((.....-((((.......))))..(((((...))))).)))))))))))).......))..))))))).. ( -25.00, z-score = -2.05, R) >droVir3.scaffold_12963 5456263 136 - 20206255 AUCAUA--AGUUAGGUUAUA-UAUCGUAUGCAUCAAA-UCAUACAAUU-AUUUAUAAGCUAGUUUUUUAAGGAAAAAA-CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUGGCAC ....((--((((((.(((((-.((.(((((.......-.))))).)).-...))))).)))((((((((((((.....-((((.......))))..(((((...))))).))))))))))))...........))))).... ( -25.70, z-score = -2.24, R) >droMoj3.scaffold_6500 14663155 136 - 32352404 AUCAUA--AGUUAGGUUAUA-UAUCGUAUGCAUCAAA-UCAUACAAUU-AUUUAUAAGCUAGUUUUUUAAGGAAAAAA-CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUGGCAC ....((--((((((.(((((-.((.(((((.......-.))))).)).-...))))).)))((((((((((((.....-((((.......))))..(((((...))))).))))))))))))...........))))).... ( -25.70, z-score = -2.24, R) >droGri2.scaffold_15126 4613679 136 + 8399593 AUCAUA--AGUUAGGUUAUA-UAUCGUAUGCAUCAAA-UCAUACAAUU-AUUUAUAAGCUAGUUUUUUAAGGAAAAAA-CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUAGCAC ....((--((((((.(((((-.((.(((((.......-.))))).)).-...))))).)))((((((((((((.....-((((.......))))..(((((...))))).))))))))))))...........))))).... ( -26.00, z-score = -2.67, R) >consensus AUCAUA__AGUUAGGUUAUA_UAUCGUAUGCAUCAAAAUCAUACAAUU_AUUUAUAAGCUAGUUUUUUAAGGAAAAAA_CACUUAAAUUUAGUGAAUUUGCAGUGCGAAAUCUUUAAAAAACCACAGACGAACACUUGGCAC .........(((((((.........(((((.........))))).................((((((((((((......((((.......))))..(((((...))))).))))))))))))...........))))))).. (-24.22 = -24.25 + 0.03)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:22:32 2011