
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 6,388,021 – 6,388,135 |
| Length | 114 |
| Max. P | 0.903547 |

| Location | 6,388,021 – 6,388,135 |
|---|---|
| Length | 114 |
| Sequences | 11 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.93 |
| Shannon entropy | 0.46668 |
| G+C content | 0.50153 |
| Mean single sequence MFE | -34.54 |
| Consensus MFE | -17.22 |
| Energy contribution | -17.62 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.50 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.00 |
| SVM RNA-class probability | 0.870261 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
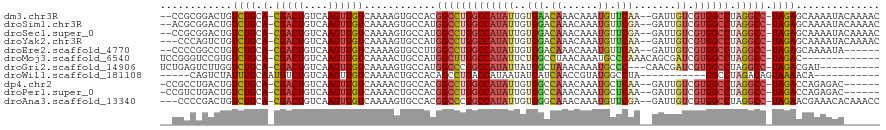
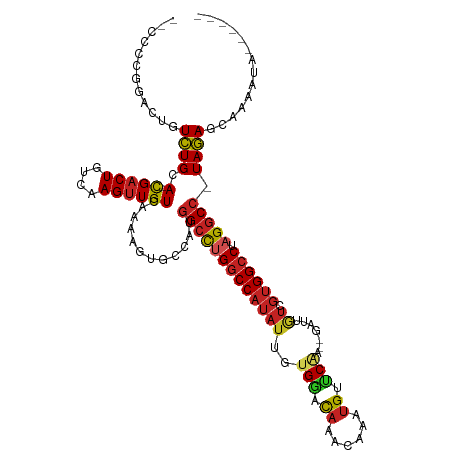
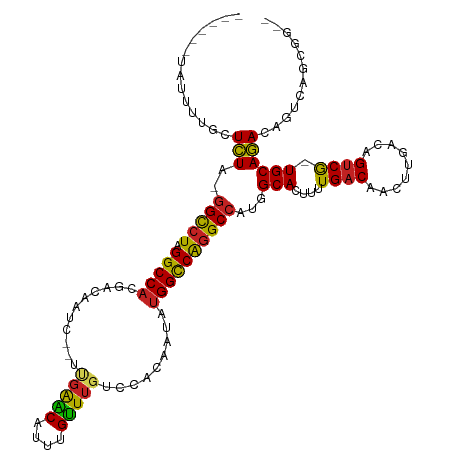
>dm3.chr3R 6388021 114 + 27905053 --CCGCGGACUGUCUGCA-CGACUGUCAAGUUGUCAAAAGUGCCACGGCCUGGCCAUAUUGUGAACAAACAAAUGUUCAA--GAUUGUCGUGGCCUAGGCC-UAGAGCAAAAUACAAAAC --..(((((((...((.(-(((((....))))))))..))).))..((((((((((((((.((((((......)))))).--)))....)))))).)))))-....))............ ( -40.10, z-score = -3.58, R) >droSim1.chr3R 12533990 114 - 27517382 --ACGCGGACUGUCUGCA-CGACUGUCAAGUUGUCAAAAGUGCCAUGGCCUGGCCAUAUUGUGGACAAACAAAUGUUCGA--GAUUGUCGUGGCCUAGGCC-UAGAGCAAAAUACAAAAC --..(((((((...((.(-(((((....))))))))..))).))..((((((((((((((.((((((......)))))).--)))....)))))).)))))-....))............ ( -38.60, z-score = -2.58, R) >droSec1.super_0 5566280 114 - 21120651 --CCGCGGACUGUCUGCA-CGACUGUCAAGUUGUCAAAAGUGCCAUGGCCUGGCCAUAUUGUGGACAAACAAAUGUUCGA--GAUUGUCGUGGCCUAGGCC-UAGAGCAAAAUACAAAAC --..(((((((...((.(-(((((....))))))))..))).))..((((((((((((((.((((((......)))))).--)))....)))))).)))))-....))............ ( -38.60, z-score = -2.61, R) >droYak2.chr3R 10418085 113 + 28832112 ---CCCAGUCUGUCUGCA-CGACUGUCAAGUUGUCAAAAGUGCCAUGGCCUGGCCAUAUUGUGGACAAACAAAUGUUCAA--GAUUGUCGUGGCCUAGGCC-UAGAGCAAAAUACAAAAC ---..(((((((....))-.))))).....((((......((((..((((((((((((((.((((((......)))))).--)))....)))))).)))))-..).)))....))))... ( -36.30, z-score = -2.74, R) >droEre2.scaffold_4770 2524332 108 - 17746568 --CCCCGGCCUGUCUGCA-CGACUGUCAAGUUGUCAAAAGUGCCUUGGCCUGGCCAUAUUGUGGACAAACAAAUGUUCAA--GAUUGUCGUGGCCUAGGCC-UAGAGCAAAAUA------ --....(((((...((.(-(((((....))))))))..)).)))..((((((((((((((.((((((......)))))).--)))....)))))).)))))-............------ ( -38.20, z-score = -2.81, R) >droMoj3.scaffold_6540 5432540 105 + 34148556 UCCGGGUCCGUGUCUGCA-CGACUGUCAAGUUGUCAAAACUGCCAUGGCUUGGCCAUAUUCUGGCCUAACAAAUGCCCAAACAGCGAUCGUGGCCUAGGCC-UAGAC------------- ((..((((.((...((.(-(((((....))))))))..)).(((((((...(((((.....))))).......(((.......))).)))))))...))))-..)).------------- ( -32.50, z-score = -0.86, R) >droGri2.scaffold_14906 9285499 105 - 14172833 UCUGAGUCUUGGUCUGCA-CGACUGUCAAGUUGUCAAAAGUGCCAUGGCCCGGCCAUAUUAUGGCUAAACAAAUGCCC---CAACGAUCGUGGCCUAGGCC-UAGACGAU---------- .....((((.((((((.(-(((((....)))))))...((.((((((..(.((((((...))))))........(...---...))..)))))))))))))-.))))...---------- ( -33.50, z-score = -1.70, R) >droWil1.scaffold_181108 180960 93 + 4707319 -----CAGUCUAUUUGCAAUGUCUGUCAAGUUGUCAAAACUGCCACAGCCUGACCAUAAUAUGAUCAACCGUAUGGCCUA-----------GGCCUAGACAGUAAAACA----------- -----.......(((((..(((((((((.(((((((....))..))))).))))....(((((......)))))(((...-----------.))).))))))))))...----------- ( -21.30, z-score = -1.17, R) >dp4.chr2 23715578 109 - 30794189 -CCGCCUGACUGUCUGCA-CGACUGUCAAGUUGUCAAAACUGCCACGGCCUGGCCAUAUUGUGGCCAAACAAAUGCUCAA--GAUUGUCGUGGCCUAGGCC-UAGACCAGAGAC------ -..(((((((.(((....-.))).))).((((.....))))((((((((.(((((((...)))))))........(....--)...))))))))..)))).-............------ ( -34.70, z-score = -1.36, R) >droPer1.super_0 10890946 109 - 11822988 -CCGUCUGACUGUCUGCA-CGACUGUCAAGUUGUCAAAACUGCCACGGCCUGGCCAUAUUGUGGCCAAACAAAUGCUCAA--GAUUGUCGUGGCCUAGGCC-UAGACCAGAGAC------ -..((((..(((((((.(-((((.(((.((((.....))))((....(..(((((((...)))))))..)....))....--))).)))))(((....)))-)))).)))))))------ ( -34.80, z-score = -1.42, R) >droAna3.scaffold_13340 22981600 113 - 23697760 ---CCCCGACUGUCUGCA-CGACUGUCAAGUUGUCAAAAGUGCCACGGCCCGGCCAUAUUGUGGGCAAACAAAUGUUCGA--GAUUGUCGUGGCCUAGGCC-UAGAACGAAACACAAACC ---....(((.(((....-.))).)))..(((.((...((.(((..((((((((...(((.((((((......)))))).--))).)))).))))..))))-).))....)))....... ( -31.30, z-score = -0.26, R) >consensus __CCCCGGACUGUCUGCA_CGACUGUCAAGUUGUCAAAAGUGCCAUGGCCUGGCCAUAUUGUGGACAAACAAAUGUUCAA__GAUUGUCGUGGCCUAGGCC_UAGAGCAAAAUA______ ............((((...(((((....))))).............(((((((((((................................)))))).))))).)))).............. (-17.22 = -17.62 + 0.40)
| Location | 6,388,021 – 6,388,135 |
|---|---|
| Length | 114 |
| Sequences | 11 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.93 |
| Shannon entropy | 0.46668 |
| G+C content | 0.50153 |
| Mean single sequence MFE | -34.29 |
| Consensus MFE | -23.62 |
| Energy contribution | -22.99 |
| Covariance contribution | -0.63 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.34 |
| Structure conservation index | 0.69 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.903547 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
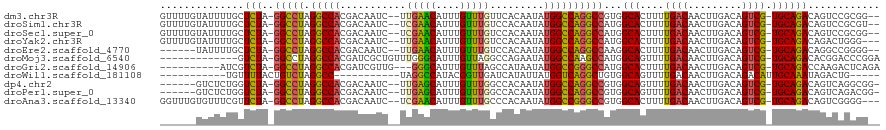
>dm3.chr3R 6388021 114 - 27905053 GUUUUGUAUUUUGCUCUA-GGCCUAGGCCACGACAAUC--UUGAACAUUUGUUUGUUCACAAUAUGGCCAGGCCGUGGCACUUUUGACAACUUGACAGUCG-UGCAGACAGUCCGCGG-- ..................-(((((.(((((.(.....)--.((((((......)))))).....))))))))))((((.(((((((((.(((....))).)-).)))).)))))))..-- ( -35.40, z-score = -1.82, R) >droSim1.chr3R 12533990 114 + 27517382 GUUUUGUAUUUUGCUCUA-GGCCUAGGCCACGACAAUC--UCGAACAUUUGUUUGUCCACAAUAUGGCCAGGCCAUGGCACUUUUGACAACUUGACAGUCG-UGCAGACAGUCCGCGU-- ...((((....((((...-(((((.(((((........--.(((((....))))).........))))))))))..))))......))))...(((.(((.-....))).))).....-- ( -33.07, z-score = -1.36, R) >droSec1.super_0 5566280 114 + 21120651 GUUUUGUAUUUUGCUCUA-GGCCUAGGCCACGACAAUC--UCGAACAUUUGUUUGUCCACAAUAUGGCCAGGCCAUGGCACUUUUGACAACUUGACAGUCG-UGCAGACAGUCCGCGG-- ...((((....((((...-(((((.(((((........--.(((((....))))).........))))))))))..))))......))))...(((.(((.-....))).))).....-- ( -33.07, z-score = -1.32, R) >droYak2.chr3R 10418085 113 - 28832112 GUUUUGUAUUUUGCUCUA-GGCCUAGGCCACGACAAUC--UUGAACAUUUGUUUGUCCACAAUAUGGCCAGGCCAUGGCACUUUUGACAACUUGACAGUCG-UGCAGACAGACUGGG--- ...((((....((((...-(((((.(((((........--.(((((....))))).........))))))))))..))))......)))).....((((((-(....)).)))))..--- ( -31.67, z-score = -1.00, R) >droEre2.scaffold_4770 2524332 108 + 17746568 ------UAUUUUGCUCUA-GGCCUAGGCCACGACAAUC--UUGAACAUUUGUUUGUCCACAAUAUGGCCAGGCCAAGGCACUUUUGACAACUUGACAGUCG-UGCAGACAGGCCGGGG-- ------.......(((..-(((((.(((((........--.(((((....))))).........))))))))))..(((.((((((((.(((....))).)-).)))).)))))))).-- ( -32.67, z-score = -1.25, R) >droMoj3.scaffold_6540 5432540 105 - 34148556 -------------GUCUA-GGCCUAGGCCACGAUCGCUGUUUGGGCAUUUGUUAGGCCAGAAUAUGGCCAAGCCAUGGCAGUUUUGACAACUUGACAGUCG-UGCAGACACGGACCCGGA -------------(((..-(((....))).....((.((((((.((..(((((((((((.....)))))..((....)).............))))))..)-).)))))))))))..... ( -35.80, z-score = -1.02, R) >droGri2.scaffold_14906 9285499 105 + 14172833 ----------AUCGUCUA-GGCCUAGGCCACGAUCGUUG---GGGCAUUUGUUUAGCCAUAAUAUGGCCGGGCCAUGGCACUUUUGACAACUUGACAGUCG-UGCAGACCAAGACUCAGA ----------...((((.-(((((.(((((.....((((---.(((.........))).)))).))))))))))...((((..(((.(.....).)))..)-)))......))))..... ( -32.90, z-score = -0.93, R) >droWil1.scaffold_181108 180960 93 - 4707319 -----------UGUUUUACUGUCUAGGCC-----------UAGGCCAUACGGUUGAUCAUAUUAUGGUCAGGCUGUGGCAGUUUUGACAACUUGACAGACAUUGCAAAUAGACUG----- -----------.........((((((((.-----------...))).(((((((((((((...))))))).))))))(((((((((.(.....).)))).)))))...)))))..----- ( -27.40, z-score = -1.76, R) >dp4.chr2 23715578 109 + 30794189 ------GUCUCUGGUCUA-GGCCUAGGCCACGACAAUC--UUGAGCAUUUGUUUGGCCACAAUAUGGCCAGGCCGUGGCAGUUUUGACAACUUGACAGUCG-UGCAGACAGUCAGGCGG- ------(((.(((((...-.))).))((((((.((...--.)).......(((((((((.....)))))))))))))))......)))..((((((.(((.-....))).))))))...- ( -40.40, z-score = -1.85, R) >droPer1.super_0 10890946 109 + 11822988 ------GUCUCUGGUCUA-GGCCUAGGCCACGACAAUC--UUGAGCAUUUGUUUGGCCACAAUAUGGCCAGGCCGUGGCAGUUUUGACAACUUGACAGUCG-UGCAGACAGUCAGACGG- ------(((....(((.(-(((....((((((.((...--.)).......(((((((((.....))))))))))))))).)))).)))....((((.(((.-....))).)))))))..- ( -38.70, z-score = -1.67, R) >droAna3.scaffold_13340 22981600 113 + 23697760 GGUUUGUGUUUCGUUCUA-GGCCUAGGCCACGACAAUC--UCGAACAUUUGUUUGCCCACAAUAUGGCCGGGCCGUGGCACUUUUGACAACUUGACAGUCG-UGCAGACAGUCGGGG--- ......((((..((.(((-(((((.(((((........--.(((((....))))).........)))))))))).))).))....)))).((((((.(((.-....))).)))))).--- ( -36.07, z-score = -0.72, R) >consensus ______UAUUUUGCUCUA_GGCCUAGGCCACGACAAUC__UUGAACAUUUGUUUGUCCACAAUAUGGCCAGGCCAUGGCACUUUUGACAACUUGACAGUCG_UGCAGACAGUCAGCGG__ ..............(((..(((((.(((((...........(((((....))))).........))))))))))...(((....((((.........)))).))))))............ (-23.62 = -22.99 + -0.63)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:03:40 2011