
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 6,234,009 – 6,234,117 |
| Length | 108 |
| Max. P | 0.999217 |

| Location | 6,234,009 – 6,234,102 |
|---|---|
| Length | 93 |
| Sequences | 11 |
| Columns | 98 |
| Reading direction | forward |
| Mean pairwise identity | 86.85 |
| Shannon entropy | 0.26544 |
| G+C content | 0.39269 |
| Mean single sequence MFE | -29.89 |
| Consensus MFE | -26.99 |
| Energy contribution | -26.24 |
| Covariance contribution | -0.76 |
| Combinations/Pair | 1.17 |
| Mean z-score | -4.25 |
| Structure conservation index | 0.90 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.72 |
| SVM RNA-class probability | 0.999217 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
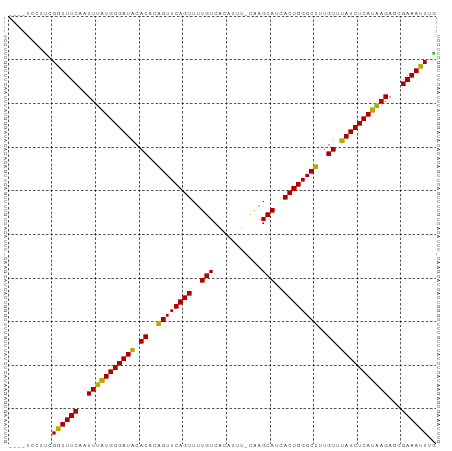
>dm3.chr3R 6234009 93 + 27905053 ----UCCUUCGGUUUCAGUUUAUGGGAUACACACAGUUCAGUUUUGUCACACUU-CAAGCAUCACUGGGCUUUGUUUAUCUCAUGAGAGCGAAAUUUA ----...((((.(((((.....(((((((.(((.((((((((..(((.......-...)))..)))))))).))).)))))))))))).))))..... ( -29.50, z-score = -4.79, R) >droSim1.chr3R 12358144 93 - 27517382 ----UCCUUCGGUUUCAGUUUAUGGGAUACACACAGUUCAGUUUUGUCACACUU-UAAGCAUCACUGGGCUUUGUUUAUCUCAUGAGAGCGAAAUUUA ----...((((.(((((.....(((((((.(((.((((((((..(((.......-...)))..)))))))).))).)))))))))))).))))..... ( -29.50, z-score = -4.91, R) >droSec1.super_0 5411247 93 - 21120651 ----UCCUUCGGUUUCAGUUUAUGGGAUACACACAGUUCAGUUUUGUCACACUU-CAAGCAUCACUGGGCUUUGUUUAUCUCAUGAGAGCGAAAUUUA ----...((((.(((((.....(((((((.(((.((((((((..(((.......-...)))..)))))))).))).)))))))))))).))))..... ( -29.50, z-score = -4.79, R) >droYak2.chr3R 10262524 93 + 28832112 ----UCCUCCGGUUUCAGUUUAUGGGAUACACACAGUUCAGUUUUGUCGCACUA-CAAGCAUCACUGGGCUUUGUUUAUCUCAUGAGAGCGAAAUUUG ----......((((((..(((((((((((.(((.((((((((..(((.......-...)))..)))))))).))).)))))))))))...)))))).. ( -29.10, z-score = -3.75, R) >droEre2.scaffold_4770 2362579 93 - 17746568 ----UCCUCCGGUUUCAGUUUAUGGGAUACACACAGUUCAGUUUUGUCACACUU-CAAGCAUCACUGGGCUUUGUUUAUCUCAUGAGAGCGAAAUUUG ----......((((((..(((((((((((.(((.((((((((..(((.......-...)))..)))))))).))).)))))))))))...)))))).. ( -29.10, z-score = -4.04, R) >droAna3.scaffold_13340 22830465 94 - 23697760 ----UCCUCCGGUUUCAAUUUGUGGGAUACACACGGUUCAGUUGUGUCACUUUUGCGAGCAUCACUGGGCUUUGUUUAUCUCAUAAGACUGAAAUUUG ----......(((((((.(((((((((((.(((.((((((((.(((((.(....).)).))).)))))))).))).)))))))))))..))))))).. ( -32.10, z-score = -4.15, R) >dp4.chr2 23560755 97 - 30794189 UGCCUCUUUUGGUUUCAAUUUAUGGGAUGCACCAAGUUCAGUUUUGUCACAUUU-CGAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGCGAAAUUGA ........(..(((((..(((((((((((.((.(((((((((..((((......-.)).))..))))))))).)).)))))))))))...)))))..) ( -29.40, z-score = -3.10, R) >droPer1.super_0 10728872 97 - 11822988 UGCCUCUUUUGGUUUCAAUUUAUGGGAUGCACCAAGUUCAGUUUUGUCACAUUU-CGAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGCGAAAUUGA ........(..(((((..(((((((((((.((.(((((((((..((((......-.)).))..))))))))).)).)))))))))))...)))))..) ( -29.40, z-score = -3.10, R) >droVir3.scaffold_13047 1194318 93 + 19223366 -----AUUUGGAUUUCAAUUUAUGGGAUACACCAAGUUCAGUUUUGUCAGAUGUCCAAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGUGAAAUUUG -----....((((((((.(((((((((((.((.(((((((((.......(((((....)))))))))))))).)).)))))))))))..)))))))). ( -31.81, z-score = -4.76, R) >droGri2.scaffold_14906 9095445 94 - 14172833 ----AUUUCGGAUUUCAAUUUAUGGGAUACACCAAGUUCAGUUUUGUCAGAUGUCCAAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGCGAAAUUAG ----......((((((..(((((((((((.((.(((((((((.......(((((....)))))))))))))).)).)))))))))))...)))))).. ( -29.51, z-score = -4.65, R) >droMoj3.scaffold_6540 5225329 94 + 34148556 ----AUUAAGGAUUUCAAUUUAUGGGAUACACUAAGUUCAGUUUUGUCAGAUGUCCAAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGCGAAAUUUG ----.....(((((((..(((((((((((.((.(((((((((.......(((((....)))))))))))))).)).)))))))))))...))))))). ( -29.91, z-score = -4.69, R) >consensus ____UCCUUCGGUUUCAAUUUAUGGGAUACACACAGUUCAGUUUUGUCACAUUU_CAAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGCGAAAUUUG ..........((((((..(((((((((((.((..((((((((..(((...........)))..))))))))..)).)))))))))))...)))))).. (-26.99 = -26.24 + -0.76)


| Location | 6,234,024 – 6,234,117 |
|---|---|
| Length | 93 |
| Sequences | 11 |
| Columns | 98 |
| Reading direction | forward |
| Mean pairwise identity | 84.06 |
| Shannon entropy | 0.32922 |
| G+C content | 0.39018 |
| Mean single sequence MFE | -27.38 |
| Consensus MFE | -22.69 |
| Energy contribution | -22.13 |
| Covariance contribution | -0.56 |
| Combinations/Pair | 1.17 |
| Mean z-score | -3.67 |
| Structure conservation index | 0.83 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.65 |
| SVM RNA-class probability | 0.999104 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
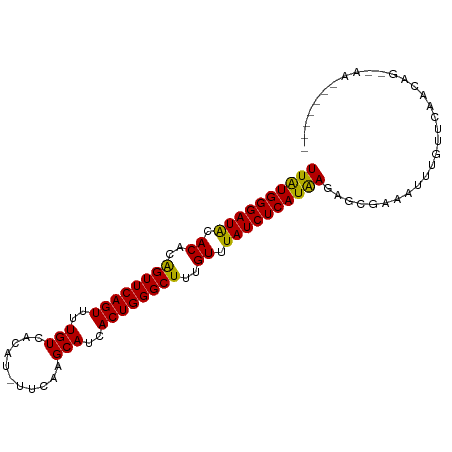
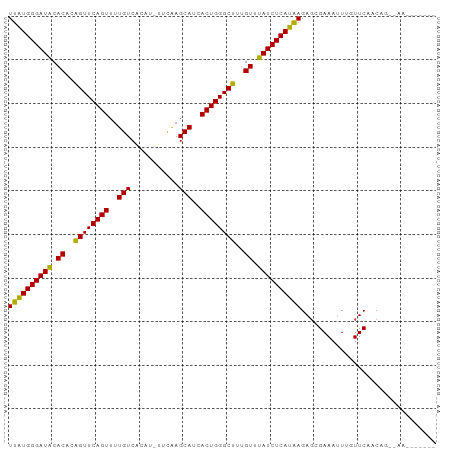
>dm3.chr3R 6234024 93 + 27905053 UUAUGGGAUACACACAGUUCAGUUUUGUCACAC-UUCAAGCAUCACUGGGCUUUGUUUAUCUCAUGAGAGCGAAAUUUAUUCAACAGGAAACCU---- ((((((((((.(((.((((((((..(((.....-.....)))..)))))))).))).))))))))))...................(....)..---- ( -25.50, z-score = -3.51, R) >droSim1.chr3R 12358159 93 - 27517382 UUAUGGGAUACACACAGUUCAGUUUUGUCACAC-UUUAAGCAUCACUGGGCUUUGUUUAUCUCAUGAGAGCGAAAUUUAUUCAACAGGGAACCU---- ((((((((((.(((.((((((((..(((.....-.....)))..)))))))).))).))))))))))...........................---- ( -24.70, z-score = -3.18, R) >droSec1.super_0 5411262 93 - 21120651 UUAUGGGAUACACACAGUUCAGUUUUGUCACAC-UUCAAGCAUCACUGGGCUUUGUUUAUCUCAUGAGAGCGAAAUUUAUUCAACAGGGAACCU---- ((((((((((.(((.((((((((..(((.....-.....)))..)))))))).))).))))))))))...........................---- ( -24.70, z-score = -3.02, R) >droYak2.chr3R 10262539 92 + 28832112 UUAUGGGAUACACACAGUUCAGUUUUGUCGCAC-UACAAGCAUCACUGGGCUUUGUUUAUCUCAUGAGAGCGAAAUUUGUUCAACAG-AAACCU---- ((((((((((.(((.((((((((..(((.....-.....)))..)))))))).))).))))))))))((((((...)))))).....-......---- ( -28.60, z-score = -3.87, R) >droEre2.scaffold_4770 2362594 92 - 17746568 UUAUGGGAUACACACAGUUCAGUUUUGUCACAC-UUCAAGCAUCACUGGGCUUUGUUUAUCUCAUGAGAGCGAAAUUUGUUCAACAUGAAAGU----- ((((((((((.(((.((((((((..(((.....-.....)))..)))))))).))).))))))))))((((((...))))))...........----- ( -28.60, z-score = -3.79, R) >droAna3.scaffold_13340 22830480 87 - 23697760 UUGUGGGAUACACACGGUUCAGUUGUGUCACUUUUGCGAGCAUCACUGGGCUUUGUUUAUCUCAUAAGACUGAAAUUUGUUCAACUU----------- ((((((((((.(((.((((((((.(((((.(....).)).))).)))))))).))).))))))))))...((((.....))))....----------- ( -27.90, z-score = -3.49, R) >dp4.chr2 23560774 97 - 30794189 UUAUGGGAUGCACCAAGUUCAGUUUUGUCACAU-UUCGAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGCGAAAUUGAUUCAACUGUUUCGGGUACU ((((((((((.((.(((((((((..((((....-...)).))..))))))))).)).))))))))))...((((((.(......).))))))...... ( -28.00, z-score = -2.83, R) >droPer1.super_0 10728891 97 - 11822988 UUAUGGGAUGCACCAAGUUCAGUUUUGUCACAU-UUCGAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGCGAAAUUGAUUCAACUGUUUCGGUUACU ((((((((((.((.(((((((((..((((....-...)).))..))))))))).)).))))))))))((.((((((.(......).)))))).))... ( -29.30, z-score = -3.52, R) >droVir3.scaffold_13047 1194332 85 + 19223366 UUAUGGGAUACACCAAGUUCAGUUUUGUCAGAUGUCCAAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGUGAAAUUUGUUCAAC------------- ((((((((((.((.(((((((((.......(((((....)))))))))))))).)).))))))))))(((..(...)..)))...------------- ( -26.21, z-score = -3.44, R) >droGri2.scaffold_14906 9095460 85 - 14172833 UUAUGGGAUACACCAAGUUCAGUUUUGUCAGAUGUCCAAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGCGAAAUUAGUUCAAC------------- ((((((((((.((.(((((((((.......(((((....)))))))))))))).)).))))))))))((((.......))))...------------- ( -28.71, z-score = -4.86, R) >droMoj3.scaffold_6540 5225344 85 + 34148556 UUAUGGGAUACACUAAGUUCAGUUUUGUCAGAUGUCCAAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGCGAAAUUUGUUCAAC------------- ((((((((((.((.(((((((((.......(((((....)))))))))))))).)).))))))))))((((((...))))))...------------- ( -28.91, z-score = -4.82, R) >consensus UUAUGGGAUACACACAGUUCAGUUUUGUCACAU_UUCAAGCAUCACUGGGCUUUGUUUAUCUCAUAAGAGCGAAAUUUGUUCAACAG__AA_______ ((((((((((.((..((((((((..(((...........)))..))))))))..)).))))))))))............................... (-22.69 = -22.13 + -0.56)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:02:51 2011