| Sequence ID | dm3.chr3R |
|---|---|
| Location | 5,925,737 – 5,925,846 |
| Length | 109 |
| Max. P | 0.999243 |

| Location | 5,925,737 – 5,925,846 |
|---|---|
| Length | 109 |
| Sequences | 14 |
| Columns | 121 |
| Reading direction | forward |
| Mean pairwise identity | 86.11 |
| Shannon entropy | 0.31280 |
| G+C content | 0.44817 |
| Mean single sequence MFE | -39.47 |
| Consensus MFE | -35.70 |
| Energy contribution | -35.09 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.34 |
| Mean z-score | -4.16 |
| Structure conservation index | 0.90 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.73 |
| SVM RNA-class probability | 0.999243 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
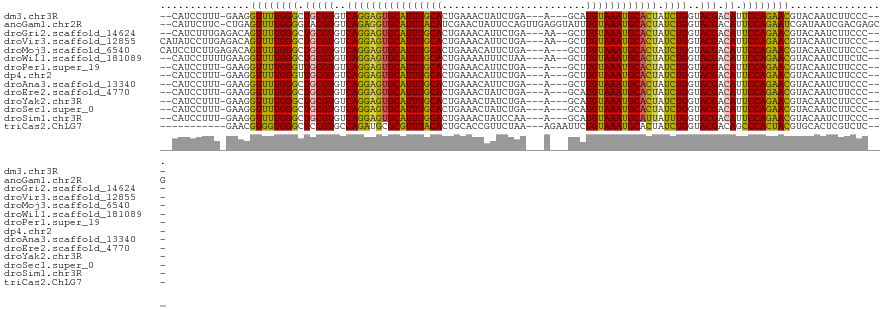
>dm3.chr3R 5925737 109 + 27905053 --CAUCCUUU-GAAGGUUUUGGGCUGCGUGUCAGGAGUGCAUUUGCACUGAAACUAUCUGA---A---GCAUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCCC--- --......((-(.(.(((((((..(((((..((((((((((((((((.((...(.....).---.---.)))))))))))))).))))..))).))..))))))).).))).......--- ( -40.50, z-score = -4.52, R) >anoGam1.chr2R 34491289 118 - 62725911 --CAUUCUUC-CUGAGUUUUGGGGUACGUGUCAGAGGUGCAUUUACAUCGAACUAUUCCAGUUGAGGUAUUUGUAAAUGCACUAUCUGGUACGACAUUCCAGAAUCGAUAAUCGACGAGCG --..(((...-.....(((((((((.(((..((((((((((((((((((.((((.....))))..))....)))))))))))).))))..)))..)))))))))(((.....))).))).. ( -36.50, z-score = -2.64, R) >droGri2.scaffold_14624 2266186 111 + 4233967 --CAUCUUUGAGACAGUUUUGGGCUGCGUGUCAGGAGUGCAUUUGCACUGAAACAUUCUGA---AA--GCUUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCCC--- --.............(((((((..(((((..((((((((((((((((((............---.)--)..)))))))))))).))))..))).))..))))))).............--- ( -37.72, z-score = -3.50, R) >droVir3.scaffold_12855 7476887 113 - 10161210 CAUAUCCUUGAGACAGUUUUGGGCUGCGUGUCAGGAGUGCAUUUGCACUGAAACAUUCUGA---AA--GCUUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCCC--- ...............(((((((..(((((..((((((((((((((((((............---.)--)..)))))))))))).))))..))).))..))))))).............--- ( -37.72, z-score = -3.48, R) >droMoj3.scaffold_6540 22263356 112 - 34148556 CAUCCUCUUGAGACAGUUUUGGGCUGCGUGUCAGGAGUGCAUUUGCACUGAAACAUUCUGA---A---GCUUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCCC--- ...............(((((((..(((((..((((((((((((((((((............---)---)..)))))))))))).))))..))).))..))))))).............--- ( -37.80, z-score = -3.34, R) >droWil1.scaffold_181089 1201427 111 - 12369635 --CAUCCUUUUGAAGGUUUUGGGCUGCGUGUCAGGAGUGCAUUUGCACUGAAAAUUUCUAA---AA--GCUUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCUC--- --.......(((.(.(((((((..(((((..((((((((((((((((((............---.)--)..)))))))))))).))))..))).))..))))))).).))).......--- ( -39.02, z-score = -4.82, R) >droPer1.super_19 1143400 109 - 1869541 --CAUCCUUU-GAAGGUUUUGGGUUGCGUGUCAGGAGUGCAUUUGCACUGAAACAUUCUGA---A---GCUUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCCC--- --......((-(.(.(((((((..(((((..((((((((((((((((((............---)---)..)))))))))))).))))..))).))..))))))).).))).......--- ( -39.10, z-score = -4.16, R) >dp4.chr2 25773489 109 - 30794189 --CAUCCUUU-GAAGGUUUUGGGUUGCGUGUCAGGAGUGCAUUUGCACUGAAACAUUCUGA---A---GCUUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCCC--- --......((-(.(.(((((((..(((((..((((((((((((((((((............---)---)..)))))))))))).))))..))).))..))))))).).))).......--- ( -39.10, z-score = -4.16, R) >droAna3.scaffold_13340 6488989 109 + 23697760 --CAUCCUUU-GAAGGUUUUGGGCUGCGUGUCAGGAGUGCAUUUGCACUGAAACAUUCUGA---A---GCUUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCCC--- --......((-(.(.(((((((..(((((..((((((((((((((((((............---)---)..)))))))))))).))))..))).))..))))))).).))).......--- ( -39.10, z-score = -4.07, R) >droEre2.scaffold_4770 2072169 109 - 17746568 --CAUCCUUU-GAAGGUUUUGGGCUGCGUGUCAGGAGUGCAUUUGCACUGAAACUAUCUGA---A---GCAUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCCC--- --......((-(.(.(((((((..(((((..((((((((((((((((.((...(.....).---.---.)))))))))))))).))))..))).))..))))))).).))).......--- ( -40.50, z-score = -4.52, R) >droYak2.chr3R 9973467 109 + 28832112 --CAUCCUUU-GAAGGUUUUGGGCUGCGUGUCAGGAGUGCAUUUGCACUGAAACUAUCUGA---A---GCAUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCCC--- --......((-(.(.(((((((..(((((..((((((((((((((((.((...(.....).---.---.)))))))))))))).))))..))).))..))))))).).))).......--- ( -40.50, z-score = -4.52, R) >droSec1.super_0 5132754 109 - 21120651 --CAUCCUUU-GAAGGUUUUGGGCUGCGUGUCAGGAGUGCAUUUGCACUGAAACUAUCUGA---A---GCAUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCCC--- --......((-(.(.(((((((..(((((..((((((((((((((((.((...(.....).---.---.)))))))))))))).))))..))).))..))))))).).))).......--- ( -40.50, z-score = -4.52, R) >droSim1.chr3R 12066954 109 - 27517382 --CAUCCUUU-GAAGGUUUUGGGCUGCGUGUCAGGAGUGCAUUUGCACUGAAACUAUCCAA---A---GCAUGUAAAUGCAUUAUUUGGUACGACAUUCCAGAACGUACAAUCUUCCC--- --......((-(.(.(((((((..(((((..((((((((((((((((.((...........---.---.)))))))))))))).))))..))).))..))))))).).))).......--- ( -34.72, z-score = -3.11, R) >triCas2.ChLG7 2753065 104 - 17478683 -----------GAACGUGGUGGGCUCCGUGCCAGAUGCGCGUUUACACUGCACCGUUCUAA---AGAAUUCUGUAAAUGCACUAUCUGGUACGACAGCCCACUACGUGCACUCGUCUC--- -----------(.((((((((((((.(((((((((((.(((((((((.......((((...---.))))..))))))))).).))))))))))..)))))))))))).).........--- ( -49.80, z-score = -6.90, R) >consensus __CAUCCUUU_GAAGGUUUUGGGCUGCGUGUCAGGAGUGCAUUUGCACUGAAACAUUCUGA___A___GCUUGUAAAUGCACUAUCUGGUACGACAUUCCAGAACGUACAAUCUUCCC___ ...............((((((((.(((((..((((((((((((((((........................)))))))))))).))))..))).)).))))))))................ (-35.70 = -35.09 + -0.61)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:02:15 2011