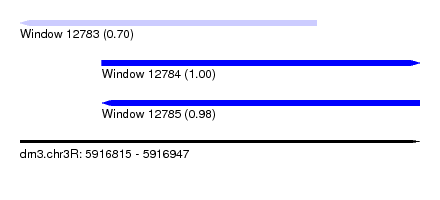
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 5,916,815 – 5,916,947 |
| Length | 132 |
| Max. P | 0.997577 |

| Location | 5,916,815 – 5,916,913 |
|---|---|
| Length | 98 |
| Sequences | 13 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 74.37 |
| Shannon entropy | 0.56826 |
| G+C content | 0.47552 |
| Mean single sequence MFE | -28.52 |
| Consensus MFE | -15.95 |
| Energy contribution | -15.98 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.13 |
| Mean z-score | -0.89 |
| Structure conservation index | 0.56 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.700264 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 5916815 98 - 27905053 AGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAAUGGCAGUUGCAUAUGGA----AAAUUC------GCUUUUCGCA-AAAAGCUCAGC .((((....(((((((((((............)))))))...)))-)....(((...((....))...))).----......------((((((....-)))))).)))) ( -24.00, z-score = 0.27, R) >droGri2.scaffold_14624 2253091 99 - 4233967 AGCUGUGUUCACUUGCUUACACAAUAUUCAAAGCGAGCACAGGGCAAUAUCCCAGUGGCAAUUGCACAGCGA---AAAGCUU------UUUUUCAACG--AAAGCUUAGU .(((((((...(((((((............)))))))(((.(((......))).)))......)))))))..---.((((((------((.......)--)))))))... ( -30.10, z-score = -2.78, R) >droMoj3.scaffold_6540 22249573 98 + 34148556 AGCUGUGUUCACUUGC-AACAUAA-ACUCAAAGCGAGCACAGGGU-GUAUCCCAGCGGCAAUUGCACAGAGA---GAAAGCU------UACUCAGAUGAAAAAGCUUAGC ..((((((....((((-..(....-.(((.....)))....(((.-....))).)..))))..))))))...---..(((((------(..((....))..))))))... ( -24.60, z-score = -0.26, R) >droVir3.scaffold_12855 7462555 92 + 10161210 AGCUGUGUUCACUUGCUUGCACA--AUUCAAAGCGAGCACAGGGU-GUAUCCCAGCGGCAAUUGCAC--UGA---AAAAGCU------UUUU--AGCG--AAAGCUUAGC .(((((.......(((((((...--.......)))))))..(((.-....))).)))))........--...---....(((------....--(((.--...))).))) ( -28.00, z-score = -0.69, R) >droWil1.scaffold_181089 1189444 100 + 12369635 AGCUGUGUUCACUUGUUUGCACAAUAUUCAAAGCGAGCACAGGGU-GUAUCCCAGUGGCAGUUGCACAGA------AGAAUCUAGAAAGCUUUUUG---GAGCUUUUAGC ..((((((((((((((((((............)))))))..(((.-....)))))))).....)))))).------........((((((((....---))))))))... ( -28.70, z-score = -0.84, R) >droPer1.super_19 1133895 108 + 1869541 AGCUGUGUUCACUUGCUUGCACCAUAUUCAAAGCGAGCACAGGGU-GUAUCCCAACGGCAGUUGCACAGAGACACGAGACACGGAAGAGCUUUCAGCU-GAAAGCUCAGC ..((((((((.(((((((((............))))))).)).((-((.((.((((....))))....)).)))))).))))))..((((((((....-))))))))... ( -37.40, z-score = -2.09, R) >dp4.chr2 25764056 108 + 30794189 AGCUGUGUUCACUUGCUUGCACCAUAUUCAAAGCGAGCACAGGGU-GUAUCCCAACGGCAGUUGCACAGAGACACGAGACACGGAAGAGCUUUCAGCU-GAAAGCUCAGC ..((((((((.(((((((((............))))))).)).((-((.((.((((....))))....)).)))))).))))))..((((((((....-))))))))... ( -37.40, z-score = -2.09, R) >droAna3.scaffold_13340 6479961 96 - 23697760 AGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAGCAGCCGAUGCACACAAA----UGA---------GCUUUUCGCGCGAAAGCUCAGC ..((((((((.(((...((.....))....))).)))))))).((-(((((.........))))))).....----(((---------((((((.....))))))))).. ( -31.40, z-score = -1.29, R) >droEre2.scaffold_4770 2062974 98 + 17746568 AGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAAUGGCAGUUGCAUAUGGA----AAAUUC------GCUUUUCGCG-CAAAGCUCAGC .((((.((((((((((((((............)))))))...)))-)....(((...((....))...))).----.....(------((.....)))-...))).)))) ( -24.40, z-score = 0.65, R) >droYak2.chr3R 9963941 98 - 28832112 AGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAAUGGCAGUUGCAUAUGGA----AAAUUC------GCUUUUCGCG-AAAAGCUCAGC .((((.((((((((((((((............)))))))...)))-)....(((...((....))...))).----...(((------((.....)))-)).))).)))) ( -27.80, z-score = -0.74, R) >droSec1.super_0 5123940 98 + 21120651 AGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAAUGGCAGUUGCAUAUGGA----AAAUUC------GCUUUUCGCG-AAAAGCUCAGC .((((.((((((((((((((............)))))))...)))-)....(((...((....))...))).----...(((------((.....)))-)).))).)))) ( -27.80, z-score = -0.74, R) >droSim1.chr3R 12058116 98 + 27517382 AGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAAUGGCAGUUGCAUAUGGA----AAAUUC------GCUUUUCGCG-AAAAGCUCAGC .((((.((((((((((((((............)))))))...)))-)....(((...((....))...))).----...(((------((.....)))-)).))).)))) ( -27.80, z-score = -0.74, R) >triCas2.ChLG7 2744804 84 + 17478683 AGCUGUGUUCACUGGU-----CGAAAUACAGAGUAAACACAGGGUGGU-CCCCAGCA-CCGCCCCACCUUCAG--CAGAUUC----GGUCUUCUCUC------------- .((((((((.(((.((-----......))..))).))))).(((((((-.......)-)))))).......))--)......----...........------------- ( -21.40, z-score = -0.26, R) >consensus AGCUGUGUUCACUUGCUUGCAUUACAUUCAAAGCGAGCACAGGGU_GUAUCCCAAUGGCAGUUGCACAUAGA____AAAUUC______GCUUUUCGCG_AAAAGCUCAGC .(((((((((.(((................))).)))))).(((......)))...)))................................................... (-15.95 = -15.98 + 0.03)

| Location | 5,916,842 – 5,916,947 |
|---|---|
| Length | 105 |
| Sequences | 12 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 79.65 |
| Shannon entropy | 0.43319 |
| G+C content | 0.50913 |
| Mean single sequence MFE | -40.50 |
| Consensus MFE | -34.57 |
| Energy contribution | -33.26 |
| Covariance contribution | -1.31 |
| Combinations/Pair | 1.61 |
| Mean z-score | -2.59 |
| Structure conservation index | 0.85 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.13 |
| SVM RNA-class probability | 0.997577 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 5916842 105 + 27905053 ------UCCAUAUGCAACUGCCAUUGGGAUAC-ACCCUGUGCUCGCUUUGAAUGAAAUGCAAGCAAGUGAACACAGCUGGUGGUAUCCAGUGGCCGUUUG--GCAUCCUU-GCUG ------.........(((.(((((((((...(-((((((((.((((((.(..((.....))..))))))).)))))..))))...))))))))).))).(--(((....)-))). ( -39.10, z-score = -2.44, R) >droGri2.scaffold_14624 2253118 106 + 4233967 ------UCGCUGUGCAAUUGCCACUGGGAUAUUGCCCUGUGCUCGCUUUGAAUAUUGUGUAAGCAAGUGAACACAGCUGGUGGUAUCCAGUGGCCGUUUG--GCGCACAU-GCAA ------..((((((((((.((((((((.((((..(((((((.((((((.(..(((...)))..))))))).)))))..))..)))))))))))).)))..--..))))).-)).. ( -46.70, z-score = -3.50, R) >droMoj3.scaffold_6540 22249601 104 - 34148556 -----CUCUCUGUGCAAUUGCCGCUGGGAUAC-ACCCUGUGCUCGCUUUGAGU-UUAUGUU-GCAAGUGAACACAGCUGGUGGUAUCCAGAGGCCGUUGA--CAGCCAAU-GCAA -----....((((.((((.(((.(((((...(-((((((((.((((((...((-.......-)))))))).)))))..))))...))))).))).)))))--))).....-.... ( -38.20, z-score = -1.82, R) >droWil1.scaffold_181089 1189473 107 - 12369635 ------CUUCUGUGCAACUGCCACUGGGAUAC-ACCCUGUGCUCGCUUUGAAUAUUGUGCAAACAAGUGAACACAGCUGGUGGUAUCCAGUGGCCGUUUUUUGCAUCCAU-GCAA ------.....(((((((.(((((((((...(-((((((((.(((((........(((....)))))))).)))))..))))...))))))))).))....)))))....-.... ( -39.40, z-score = -2.83, R) >droPer1.super_19 1133926 111 - 1869541 CUCGUGUCUCUGUGCAACUGCCGUUGGGAUAC-ACCCUGUGCUCGCUUUGAAUAUGGUGCAAGCAAGUGAACACAGCUGGUGGUAUCCAAUGGCCGUUCU--GCAACAAU-GCAA ....(((.....((((((.(((((((((...(-((((((((.((((((................)))))).)))))..))))...))))))))).))..)--))).....-))). ( -38.19, z-score = -1.41, R) >dp4.chr2 25764087 111 - 30794189 CUCGUGUCUCUGUGCAACUGCCGUUGGGAUAC-ACCCUGUGCUCGCUUUGAAUAUGGUGCAAGCAAGUGAACACAGCUGGUGGUAUCCAAUGGCCGUUCU--GCAACAAU-GCAA ....(((.....((((((.(((((((((...(-((((((((.((((((................)))))).)))))..))))...))))))))).))..)--))).....-))). ( -38.19, z-score = -1.41, R) >droAna3.scaffold_13340 6479989 102 + 23697760 ---------GUGUGCAUCGGCUGCUGGGAUAC-ACCCUGUGCUCGCUUUGAAUGAAAUGCAAGCAAGUGAACACAGCUGGUGGUAUCCAGUGGCCGUUUU--GCAUCCUU-UCAA ---------(..((((.((((..(((((...(-((((((((.((((((.(..((.....))..))))))).)))))..))))...)))))..))))...)--)))..)..-.... ( -45.10, z-score = -4.21, R) >droEre2.scaffold_4770 2063001 105 - 17746568 ------UCCAUAUGCAACUGCCAUUGGGAUAC-ACCCUGUGCUCGCUUUGAAUGAAAUGCAAGCAAGUGAACACAGCUGGUGGUAUCCAGUGGCCGUUUG--GCGUCCUU-GCUG ------.....((((.((.(((((((((...(-((((((((.((((((.(..((.....))..))))))).)))))..))))...))))))))).))...--))))....-.... ( -38.50, z-score = -2.17, R) >droYak2.chr3R 9963968 105 + 28832112 ------UCCAUAUGCAACUGCCAUUGGGAUAC-ACCCUGUGCUCGCUUUGAAUGAAAUGCAAGCAAGUGAACACAGCUGGUGGUAUCCAGUGGCCGUUUG--GCGUCCUU-GCUG ------.....((((.((.(((((((((...(-((((((((.((((((.(..((.....))..))))))).)))))..))))...))))))))).))...--))))....-.... ( -38.50, z-score = -2.17, R) >droSec1.super_0 5123967 105 - 21120651 ------UCCAUAUGCAACUGCCAUUGGGAUAC-ACCCUGUGCUCGCUUUGAAUGAAAUGCAAGCAAGUGAACACAGCUGGUGGUAUCCAGUGGCCGUUUG--GCAUCCUU-GCUG ------.........(((.(((((((((...(-((((((((.((((((.(..((.....))..))))))).)))))..))))...))))))))).))).(--(((....)-))). ( -39.10, z-score = -2.44, R) >droSim1.chr3R 12058143 106 - 27517382 ------UCCAUAUGCAACUGCCAUUGGGAUAC-ACCCUGUGCUCGCUUUGAAUGAAAUGCAAGCAAGUGAACACAGCUGGUGGUAUCCAGUGGCCGUUUG--GCAUCCUUUGCUG ------.........(((.(((((((((...(-((((((((.((((((.(..((.....))..))))))).)))))..))))...))))))))).))).(--(((.....)))). ( -39.10, z-score = -2.50, R) >triCas2.ChLG7 2744821 100 - 17478683 ----GCUGAAGGUGGGGCGG-UGCUGGGG-ACCACCCUGUGUUUACUCUG-----UAUUUCGACCAGUGAACACAGCUGGUGGUAUCU--CAGUUUCUUC--CCAUCAAUGACAA ----......(((((((.((-.(((((((-(((((((((((((((((..(-----(......)).)))))))))))..)))))).)))--)))).)).))--)))))........ ( -45.90, z-score = -4.26, R) >consensus ______UCCAUGUGCAACUGCCAUUGGGAUAC_ACCCUGUGCUCGCUUUGAAUGAAAUGCAAGCAAGUGAACACAGCUGGUGGUAUCCAGUGGCCGUUUG__GCAUCCAU_GCAA ...........(((((((.((((((((.((((.((((((((.((((((................)))))).)))))..))).)))))))))))).)))....))))......... (-34.57 = -33.26 + -1.31)


| Location | 5,916,842 – 5,916,947 |
|---|---|
| Length | 105 |
| Sequences | 12 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 79.65 |
| Shannon entropy | 0.43319 |
| G+C content | 0.50913 |
| Mean single sequence MFE | -36.30 |
| Consensus MFE | -25.86 |
| Energy contribution | -26.48 |
| Covariance contribution | 0.62 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.71 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.08 |
| SVM RNA-class probability | 0.981622 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 5916842 105 - 27905053 CAGC-AAGGAUGC--CAAACGGCCACUGGAUACCACCAGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAAUGGCAGUUGCAUAUGGA------ ....-....((((--..(((.((((..((((((.(((..((((((((.(((...((.....))....))).)))))))))))-))))))...)))).))))))).....------ ( -35.80, z-score = -1.43, R) >droGri2.scaffold_14624 2253118 106 - 4233967 UUGC-AUGUGCGC--CAAACGGCCACUGGAUACCACCAGCUGUGUUCACUUGCUUACACAAUAUUCAAAGCGAGCACAGGGCAAUAUCCCAGUGGCAAUUGCACAGCGA------ ((((-.((((((.--......(((((((((((...((..((((((((.(((................))).))))))))))...))).))))))))...))))))))))------ ( -40.09, z-score = -3.29, R) >droMoj3.scaffold_6540 22249601 104 + 34148556 UUGC-AUUGGCUG--UCAACGGCCUCUGGAUACCACCAGCUGUGUUCACUUGC-AACAUAA-ACUCAAAGCGAGCACAGGGU-GUAUCCCAGCGGCAAUUGCACAGAGAG----- ....-.....(((--((((..(((.((((((((.(((..((((((((.(((..-.......-.....))).)))))))))))-)))).)))).)))..))).))))....----- ( -38.84, z-score = -2.88, R) >droWil1.scaffold_181089 1189473 107 + 12369635 UUGC-AUGGAUGCAAAAAACGGCCACUGGAUACCACCAGCUGUGUUCACUUGUUUGCACAAUAUUCAAAGCGAGCACAGGGU-GUAUCCCAGUGGCAGUUGCACAGAAG------ ((((-(....)))))..(((.((((((((((((.(((..((((((((.(((................))).)))))))))))-)))).)))))))).))).........------ ( -41.19, z-score = -3.38, R) >droPer1.super_19 1133926 111 + 1869541 UUGC-AUUGUUGC--AGAACGGCCAUUGGAUACCACCAGCUGUGUUCACUUGCUUGCACCAUAUUCAAAGCGAGCACAGGGU-GUAUCCCAACGGCAGUUGCACAGAGACACGAG .(((-.((((.((--((....(((.((((((((.(((..((((((((.(((................))).)))))))))))-)))).)))).)))..)))))))).).)).... ( -36.49, z-score = -1.88, R) >dp4.chr2 25764087 111 + 30794189 UUGC-AUUGUUGC--AGAACGGCCAUUGGAUACCACCAGCUGUGUUCACUUGCUUGCACCAUAUUCAAAGCGAGCACAGGGU-GUAUCCCAACGGCAGUUGCACAGAGACACGAG .(((-.((((.((--((....(((.((((((((.(((..((((((((.(((................))).)))))))))))-)))).)))).)))..)))))))).).)).... ( -36.49, z-score = -1.88, R) >droAna3.scaffold_13340 6479989 102 - 23697760 UUGA-AAGGAUGC--AAAACGGCCACUGGAUACCACCAGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAGCAGCCGAUGCACAC--------- ....-.....(((--(...((((..((((((((.(((..((((((((.(((...((.....))....))).)))))))))))-)))).))))..)))).))))...--------- ( -36.40, z-score = -2.78, R) >droEre2.scaffold_4770 2063001 105 + 17746568 CAGC-AAGGACGC--CAAACGGCCACUGGAUACCACCAGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAAUGGCAGUUGCAUAUGGA------ ..((-((...((.--....))((((..((((((.(((..((((((((.(((...((.....))....))).)))))))))))-))))))...))))..)))).......------ ( -34.80, z-score = -1.28, R) >droYak2.chr3R 9963968 105 - 28832112 CAGC-AAGGACGC--CAAACGGCCACUGGAUACCACCAGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAAUGGCAGUUGCAUAUGGA------ ..((-((...((.--....))((((..((((((.(((..((((((((.(((...((.....))....))).)))))))))))-))))))...))))..)))).......------ ( -34.80, z-score = -1.28, R) >droSec1.super_0 5123967 105 + 21120651 CAGC-AAGGAUGC--CAAACGGCCACUGGAUACCACCAGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAAUGGCAGUUGCAUAUGGA------ ....-....((((--..(((.((((..((((((.(((..((((((((.(((...((.....))....))).)))))))))))-))))))...)))).))))))).....------ ( -35.80, z-score = -1.43, R) >droSim1.chr3R 12058143 106 + 27517382 CAGCAAAGGAUGC--CAAACGGCCACUGGAUACCACCAGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU-GUAUCCCAAUGGCAGUUGCAUAUGGA------ .........((((--..(((.((((..((((((.(((..((((((((.(((...((.....))....))).)))))))))))-))))))...)))).))))))).....------ ( -35.80, z-score = -1.42, R) >triCas2.ChLG7 2744821 100 + 17478683 UUGUCAUUGAUGG--GAAGAAACUG--AGAUACCACCAGCUGUGUUCACUGGUCGAAAUA-----CAGAGUAAACACAGGGUGGU-CCCCAGCA-CCGCCCCACCUUCAGC---- ......(((((((--(..(...(((--.(..((((((..(((((((.(((.((......)-----)..))).)))))))))))))-..))))..-.)..))))...)))).---- ( -29.10, z-score = -0.84, R) >consensus UUGC_AAGGAUGC__CAAACGGCCACUGGAUACCACCAGCUGUGUUCACUUGCUUGCAUUUCAUUCAAAGCGAGCACAGGGU_GUAUCCCAAUGGCAGUUGCACAUGGA______ .....................(((...((((((.(((..((((((((.(((................))).))))))))))).))))))....)))................... (-25.86 = -26.48 + 0.62)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:02:13 2011