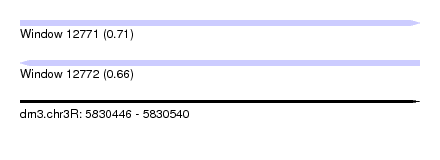
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 5,830,446 – 5,830,540 |
| Length | 94 |
| Max. P | 0.709485 |

| Location | 5,830,446 – 5,830,540 |
|---|---|
| Length | 94 |
| Sequences | 12 |
| Columns | 103 |
| Reading direction | forward |
| Mean pairwise identity | 79.68 |
| Shannon entropy | 0.42425 |
| G+C content | 0.67735 |
| Mean single sequence MFE | -44.57 |
| Consensus MFE | -27.61 |
| Energy contribution | -28.18 |
| Covariance contribution | 0.57 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.41 |
| Structure conservation index | 0.62 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.709485 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
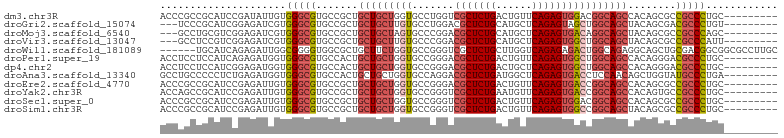
>dm3.chr3R 5830446 94 + 27905053 ACCCGCCGCAUCCGAUAUUGUGGGCGUGCCGCUGCUGCUGGUGCCUGGUCGCUCUGACUGUUCAGAGUGGACGGCAGCCACAGCGCCGCCCUGC--------- ...(((((((........))).)))).((.((.((((.(((((((.(..((((((((....))))))))..))))).))))))))).)).....--------- ( -41.80, z-score = -0.85, R) >droGri2.scaffold_15074 4089538 91 + 7742996 ---UCCCGCAUCGGAGAUCGUGGGCGUGCCGCUGCUGCUUGUGCCUGGACGCUCUGCAUGCUCAGAGUAGCUGGCAGCUACAGCGACGCCCUGU--------- ---(((((...))).))....((((((..(((((..(((...(((.((..((((((......))))))..))))))))..)))))))))))...--------- ( -38.00, z-score = 0.01, R) >droMoj3.scaffold_6540 31115014 91 + 34148556 ---GCCUGCGUCGGAGAUCGUGGGCGUGCCGCUGCUGCUAGUGCCCGGACGCUCUGCAUGCUCAGAGUGACAGGCAGCUACAGCGCCGCCCAGC--------- ---(((..((........))..)))(.((.((.((((.(((((((.(..(((((((......))))))).).)))).))))))))).)).)...--------- ( -40.50, z-score = 0.22, R) >droVir3.scaffold_13047 13064993 91 + 19223366 ---GCCUCCGUCGGAGAUCGUGGGCGUGCCGCUGCUGCUUGUGCCCGGACGCUCUGCAUGCUCAGAGUGGCUGGCAGCUACAGCGCCGCCCAUU--------- ---..((((...))))...(((((((.((.((((..(((...(((.((.(((((((......))))))).))))))))..))))))))))))).--------- ( -43.60, z-score = -1.12, R) >droWil1.scaffold_181089 6821232 97 - 12369635 ------UGCAUCAGAGAUUGGCGGGGUGGCGCUGCUUCUGGUGCCGGGUCGCUCUGCUUGGUCAGAGAGACUGGCAGAGGCAGCUGCGACGGCGGCGCCUUGC ------..............((((((((.((((((((((...(((((.((.(((((......))))).)))))))))))))))).((....))).)))))))) ( -46.10, z-score = -0.92, R) >droPer1.super_19 1026954 94 - 1869541 ACCUCCUCCAUCAGAGAUGGUGGGCGUGCCACUGCUGCUGGUGCCGGGACGCUCUGACUGUUCAGAGUGGCUGGCAGCCACAGGGACGCCCUGC--------- (((..(((.....)))..)))((((((.((.(((....(((((((((..((((((((....)))))))).)))))).))))))))))))))...--------- ( -47.50, z-score = -2.12, R) >dp4.chr2 25660063 94 - 30794189 ACCUCCUCCAUCGGAGAUGGUGGGCGUGCCACUGCUGCUGGUGCCGGGACGCUCUGACUGCUCAGAGUGGCUGGCAGCCACAGGGACGCCCUGC--------- (((..((((...))))..)))((((((.((.(((....(((((((((..((((((((....)))))))).)))))).))))))))))))))...--------- ( -49.70, z-score = -2.42, R) >droAna3.scaffold_13340 6391082 94 + 23697760 GCCUGCCCCCUCUGAGAUGGUGGGCGUGCCACUGCUGCUGGUGCCAGGACGCUCUGAUGGCUCAGAGUGACCUCCAACAGCUGGUAUGCCCUGA--------- ....(((..(.....)..)))((((((((((..((((.(((....(((.((((((((....)))))))).)))))).))))))))))))))...--------- ( -42.90, z-score = -2.58, R) >droEre2.scaffold_4770 1974374 94 - 17746568 ACCCGCCGCAUCCGAGAUUGUGGGCGUGCCGCUGCUGCUGGUGCCGGGACGCUCUGACUGUUCAGAGUGACCGGCAGCCACAGCGCCGCCCUGC--------- ...(((((((........))).)))).((.((.((((.(((((((((..((((((((....)))))))).)))))).))))))))).)).....--------- ( -46.40, z-score = -2.12, R) >droYak2.chr3R 9878903 94 + 28832112 ACCAGCCGCAUCCGAGAUUGUGGGCGUGCCGCUGCUGCUGGUGCCGGGUCGCUCUGAAUGUUCAGAGUGACCGGCAGCCACAGUGCCGCCCUGC--------- .......(((........)))(((((.((....((((.(((((((((.(((((((((....))))))))))))))).))))))))))))))...--------- ( -46.90, z-score = -2.46, R) >droSec1.super_0 5032845 94 - 21120651 ACCCGCCGCAUCCGAGAUUGUGGGCGUGCCGCUGCUGCUGGUGCCGGGUCGCUCUGACUGUUCAGAGUGGACGGCAGCCACAGCGCCGCCCUGC--------- ...(((((((........))).)))).((.((.((((.((((((((..(((((((((....))))))))).))))).))))))))).)).....--------- ( -44.40, z-score = -0.92, R) >droSim1.chr3R 11981787 94 - 27517382 ACCCGCCGCAUCCGAGAUUGUGGGCGUGCCGCUGCUGCUGGUGCCGGGUCGCUCUGACUGUUCAGAGUGGCCGGCAGCUACAGCGCCGCCCUGC--------- .......(((........)))(((((.((.((((..(((...(((((.(((((((((....)))))))))))))))))..)))))))))))...--------- ( -47.00, z-score = -1.59, R) >consensus ACCCGCCGCAUCCGAGAUUGUGGGCGUGCCGCUGCUGCUGGUGCCGGGACGCUCUGACUGUUCAGAGUGGCCGGCAGCCACAGCGCCGCCCUGC_________ .....................(((((.......(((((((((.......(((((((......))))))))))))))))........)))))............ (-27.61 = -28.18 + 0.57)


| Location | 5,830,446 – 5,830,540 |
|---|---|
| Length | 94 |
| Sequences | 12 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 79.68 |
| Shannon entropy | 0.42425 |
| G+C content | 0.67735 |
| Mean single sequence MFE | -42.02 |
| Consensus MFE | -27.40 |
| Energy contribution | -27.36 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.25 |
| Structure conservation index | 0.65 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.36 |
| SVM RNA-class probability | 0.659558 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 5830446 94 - 27905053 ---------GCAGGGCGGCGCUGUGGCUGCCGUCCACUCUGAACAGUCAGAGCGACCAGGCACCAGCAGCAGCGGCACGCCCACAAUAUCGGAUGCGGCGGGU ---------((.((((((((((((.(((((((((..((((((....)))))).)))..)))...))).))))).)).))))).......((....)))).... ( -40.50, z-score = -0.61, R) >droGri2.scaffold_15074 4089538 91 - 7742996 ---------ACAGGGCGUCGCUGUAGCUGCCAGCUACUCUGAGCAUGCAGAGCGUCCAGGCACAAGCAGCAGCGGCACGCCCACGAUCUCCGAUGCGGGA--- ---------...((((((.(((((.(((((......(((((......))))).((....))....))))).)))))))))))......((((...)))).--- ( -38.50, z-score = -0.77, R) >droMoj3.scaffold_6540 31115014 91 - 34148556 ---------GCUGGGCGGCGCUGUAGCUGCCUGUCACUCUGAGCAUGCAGAGCGUCCGGGCACUAGCAGCAGCGGCACGCCCACGAUCUCCGACGCAGGC--- ---------(((((((((((((((.((((((((.(.(((((......))))).)..)))))...))).))))).)).))))))((.....))..))....--- ( -42.30, z-score = -0.55, R) >droVir3.scaffold_13047 13064993 91 - 19223366 ---------AAUGGGCGGCGCUGUAGCUGCCAGCCACUCUGAGCAUGCAGAGCGUCCGGGCACAAGCAGCAGCGGCACGCCCACGAUCUCCGACGGAGGC--- ---------..((((((..(((((.(((((..(((.(((((......)))))......)))....))))).))))).))))))...(((((...))))).--- ( -42.10, z-score = -1.38, R) >droWil1.scaffold_181089 6821232 97 + 12369635 GCAAGGCGCCGCCGUCGCAGCUGCCUCUGCCAGUCUCUCUGACCAAGCAGAGCGACCCGGCACCAGAAGCAGCGCCACCCCGCCAAUCUCUGAUGCA------ ((..((((.....((.((.(((((.((((((.(((.(((((......))))).)))..)))...))).))))))).))..)))).(((...))))).------ ( -35.80, z-score = -1.52, R) >droPer1.super_19 1026954 94 + 1869541 ---------GCAGGGCGUCCCUGUGGCUGCCAGCCACUCUGAACAGUCAGAGCGUCCCGGCACCAGCAGCAGUGGCACGCCCACCAUCUCUGAUGGAGGAGGU ---------...((((((((((((.((((...(((.((((((....))))))......)))..)))).)))).)).))))))(((..(((.....)))..))) ( -43.10, z-score = -1.58, R) >dp4.chr2 25660063 94 + 30794189 ---------GCAGGGCGUCCCUGUGGCUGCCAGCCACUCUGAGCAGUCAGAGCGUCCCGGCACCAGCAGCAGUGGCACGCCCACCAUCUCCGAUGGAGGAGGU ---------...((((((((((((.((((...(((.((((((....))))))......)))..)))).)))).)).))))))(((..((((...))))..))) ( -44.40, z-score = -1.81, R) >droAna3.scaffold_13340 6391082 94 - 23697760 ---------UCAGGGCAUACCAGCUGUUGGAGGUCACUCUGAGCCAUCAGAGCGUCCUGGCACCAGCAGCAGUGGCACGCCCACCAUCUCAGAGGGGGCAGGC ---------...((.....)).(((((((((((.(.((((((....)))))).).)))....))))))))....((..((((.((........))))))..)) ( -40.50, z-score = -1.60, R) >droEre2.scaffold_4770 1974374 94 + 17746568 ---------GCAGGGCGGCGCUGUGGCUGCCGGUCACUCUGAACAGUCAGAGCGUCCCGGCACCAGCAGCAGCGGCACGCCCACAAUCUCGGAUGCGGCGGGU ---------...((((((((((((.((((((((.(.((((((....)))))).)..)))))...))).))))).)).))))).....((((.......)))). ( -45.70, z-score = -1.31, R) >droYak2.chr3R 9878903 94 - 28832112 ---------GCAGGGCGGCACUGUGGCUGCCGGUCACUCUGAACAUUCAGAGCGACCCGGCACCAGCAGCAGCGGCACGCCCACAAUCUCGGAUGCGGCUGGU ---------((..((((((......))))))((((.((((((....)))))).))))..))((((((.((((((...)))((........)).))).)))))) ( -44.40, z-score = -2.01, R) >droSec1.super_0 5032845 94 + 21120651 ---------GCAGGGCGGCGCUGUGGCUGCCGUCCACUCUGAACAGUCAGAGCGACCCGGCACCAGCAGCAGCGGCACGCCCACAAUCUCGGAUGCGGCGGGU ---------...((((((((((((.(((((((..(.((((((....)))))).)...))))...))).))))).)).))))).....((((.......)))). ( -41.80, z-score = -0.36, R) >droSim1.chr3R 11981787 94 + 27517382 ---------GCAGGGCGGCGCUGUAGCUGCCGGCCACUCUGAACAGUCAGAGCGACCCGGCACCAGCAGCAGCGGCACGCCCACAAUCUCGGAUGCGGCGGGU ---------...((((((((((((.((((((((.(.((((((....)))))).)..)))))...))).))))).)).))))).....((((.......)))). ( -45.20, z-score = -1.54, R) >consensus _________GCAGGGCGGCGCUGUGGCUGCCGGCCACUCUGAACAGUCAGAGCGUCCCGGCACCAGCAGCAGCGGCACGCCCACAAUCUCGGAUGCGGCAGGU ............(((((...((((.(((((..(((.(((((......)))))......)))....))))).))))..)))))..................... (-27.40 = -27.36 + -0.04)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:02:01 2011