
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 4,214,255 – 4,214,370 |
| Length | 115 |
| Max. P | 0.809845 |

| Location | 4,214,255 – 4,214,370 |
|---|---|
| Length | 115 |
| Sequences | 9 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 87.71 |
| Shannon entropy | 0.27603 |
| G+C content | 0.57834 |
| Mean single sequence MFE | -47.46 |
| Consensus MFE | -34.19 |
| Energy contribution | -35.53 |
| Covariance contribution | 1.34 |
| Combinations/Pair | 1.30 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.72 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.76 |
| SVM RNA-class probability | 0.809845 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 4214255 115 + 27905053 GCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGUGAUCCGUGUCCCGGGUACUGUUGCUGCGGAUGCUGGGAUGCGGCAUGUA (((..((((...))))..)))(((((((((...)))))))))((((......((((((....))))))..((((((((((.((((((....)))).))))))))))))))))... ( -49.20, z-score = -2.09, R) >droSim1.chr3R 10466564 115 - 27517382 GCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGUGAUCCGUGUCCCGGGUACUGUUGCUGCGGAUGCUGGGAUGCGGCAUGUA (((..((((...))))..)))(((((((((...)))))))))((((......((((((....))))))..((((((((((.((((((....)))).))))))))))))))))... ( -49.20, z-score = -2.09, R) >droSec1.super_0 3505271 115 - 21120651 GCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGUGAUCCGUGUCCCGGGUACUGUUGCUGCGGAUGCUGGGAUGCGGCAUGUA (((..((((...))))..)))(((((((((...)))))))))((((......((((((....))))))..((((((((((.((((((....)))).))))))))))))))))... ( -49.20, z-score = -2.09, R) >droYak2.chr3R 8292759 115 + 28832112 GCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCGUGUCCCGGGUACUGUUGCUGCGGAUGCUGAGAUGCGGCAUGUA (((..((((...))))..)))(((((((((...)))))))))(((((......)..(((.((((((.((((((((..(((...)))..)).)))))))))).)).)))))))... ( -45.00, z-score = -1.27, R) >droEre2.scaffold_4770 390981 115 - 17746568 GCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCGUGUCCCGGGUACUGUUGCUGCGGAUGCUGGGAUGCGGCAUGUA (((..((((...))))..)))(((((((((...)))))))))((((........((((((...)))))).((((((((((.((((((....)))).))))))))))))))))... ( -50.70, z-score = -2.26, R) >droAna3.scaffold_13340 19517760 115 - 23697760 GCCUGCUAGACUCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCGUGGCCCGGAUACUGCUGCUGCGGAUGCUGGGACGCGGCGUGGA (((..((((...))))..)))(((((((((...))))))))).........(((((.(((((((((.(((((..((.(((...)))..))..)))))))))))....)))))))) ( -51.10, z-score = -1.87, R) >droWil1.scaffold_181089 779771 115 + 12369635 GCCUGCUAGACUCUAGUGGGCGGUUGUAAUAUCAUAGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCAUGGCCCGGAUACUGCUGUUGCGGAUGCUGUGAGGCAGCAUGUA (((..((((...))))..)))((((((.........))))))(((((......)..(((((((((((((((.......)))).((((....)))).))))).))))))))))... ( -47.70, z-score = -1.84, R) >droMoj3.scaffold_6540 3255097 115 + 34148556 GCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCAUGUCCCGGAUAUUGUUGCUGUGGGUGCUGCGAGGCGGCGUGCA (((..((((...))))..)))(((((((((...))))))))).((((..((......(((((((((.((((((((..((.....))..)).)))))))))).)))))))..)))) ( -49.40, z-score = -1.90, R) >anoGam1.chr2R 46321772 95 + 62725911 GUCUGCUAAACUCUUGUCGGUGGCUGCAAUAUCAUCGCAGCCAUGCGUACCUUCAUUGCUUCCGGCGAGCCGGGCGUUCCGUUCUGGUGC---GGAUG----------------- ((((((.......((((((((((((((.........))))))).(((.........)))..)))))))((((((((...)).))))))))---)))).----------------- ( -35.60, z-score = -1.90, R) >consensus GCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCGUGUCCCGGGUACUGUUGCUGCGGAUGCUGGGAUGCGGCAUGUA (((((((((...)))))))))(((((((((...)))))))))(((((......)..(((.((((((.((((((((..(((...)))..)).)))))))))).)).)))))))... (-34.19 = -35.53 + 1.34)

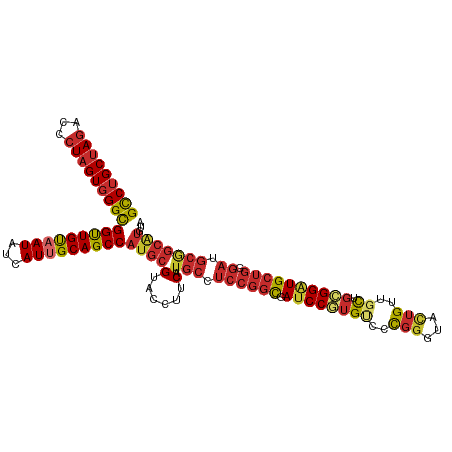
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:58:15 2011