
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 3,358,052 – 3,358,169 |
| Length | 117 |
| Max. P | 0.565026 |

| Location | 3,358,052 – 3,358,169 |
|---|---|
| Length | 117 |
| Sequences | 15 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 79.19 |
| Shannon entropy | 0.48149 |
| G+C content | 0.60399 |
| Mean single sequence MFE | -46.31 |
| Consensus MFE | -26.83 |
| Energy contribution | -26.75 |
| Covariance contribution | -0.09 |
| Combinations/Pair | 1.69 |
| Mean z-score | -1.10 |
| Structure conservation index | 0.58 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.565026 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
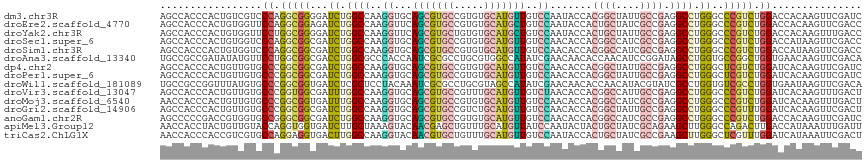
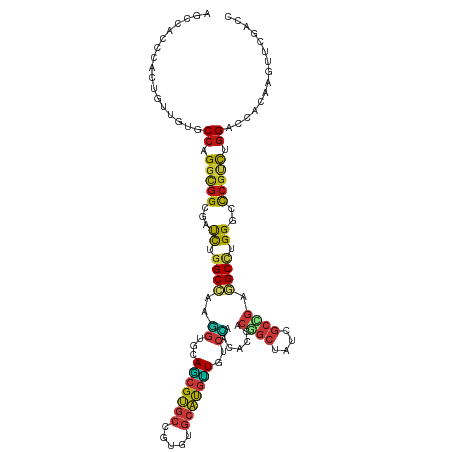
>dm3.chr3R 3358052 117 + 27905053 AGCCACCCACUGUCGUCCCAGGCGGGGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAAUACCACGGCUAUUGCCGAGGCCUGGGCCCGUCUGGACCACAAGUUCGAUC ..........(((.((((.(((((((..((.((((..((.(((((((((.....))))))))))).......((((....)))).)))).)).))))))))))).)))......... ( -52.90, z-score = -2.04, R) >droEre2.scaffold_4770 3618649 117 + 17746568 AGCCACCCACUGUGGUUCCAGGCGGAGAUCUGGCCAAGGUUCAGCGUGCCGUGUGCAUGCUGUCCAAUACCACUGCUAUCGCCGAGGCCUGGGCCCGUCUGGACCACAAGUUCGACC ..........((((((.((((((((.(..(.((((..((..((((((((.....)))))))).)).......(.((....)).).)))).)..)))))))))))))))......... ( -50.90, z-score = -2.18, R) >droYak2.chr3R 21873692 117 - 28832112 AGCCACCCACUGUGGUUCCUGGCGGGGAUCUGGCCAAGGUUCAGCGUGCUGUGUGCAUGCUGUCCAAUACCACUGCUAUUGCCGAGGCCUGGGCCCGUCUGGACCACAAGUUUGACC ..........((((((((..((((((..((.((((..((..((((((((.....)))))))).)).......(.((....)).).)))).)).)))))).))))))))......... ( -50.90, z-score = -1.71, R) >droSec1.super_6 3453687 117 + 4358794 AGCCACCCACUGUGGUCCCAGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAACACCACGGCCAUCGCCGAGGCCUGGGCCCGUCUGGACCAUAAGUUCGACC ..........((((((((.(((((((((..(((((..((((((((((((.....)))))))).....))))..))))))))))(.(((....))))))))))))))))......... ( -55.90, z-score = -2.52, R) >droSim1.chr3R 3409846 117 + 27517382 AGCCACCCACUGUGGUCCCAGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAACACCACGGCCAUCGCCGAGGCCUGGGCCCGUCUGGACCAUAAGUUCGACC ..........((((((((.(((((((((..(((((..((((((((((((.....)))))))).....))))..))))))))))(.(((....))))))))))))))))......... ( -55.90, z-score = -2.52, R) >droAna3.scaffold_13340 11836443 117 - 23697760 UGCCGCCGAUAUAUGUUCCUGGCGGCGACCUGGCGCCCACCAAUCGCGCCUGCGUGGCCAUAUCGAACAACACCAACAUCCGGAUAGCCUGGUGCCGGCUGGUGAACAAGUUCGACA .(((((((.....(((((.((((.(((....(((((.........)))))..))).))))....))))).(((((......((....))))))).)))).)))(((....))).... ( -41.00, z-score = -0.33, R) >dp4.chr2 26328248 117 + 30794189 AGCCACCCACUGUUGUGCCCGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAACACCACGGCUAUUGCCGAGGCCUGGGCUCGUCUGGAUCACAAGUUCGAUC .(((...(((....)))...)))((((((((((((..((((((((((((.....)))))))).....)))).((((....)))).)))).))).)))))..((((........)))) ( -46.80, z-score = -0.43, R) >droPer1.super_6 1658104 117 + 6141320 AGCCACCCACUGUUGUGCCCGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAACACCACGGCUAUUGCCGAGGCCUGGGCUCGUCUGGAUCACAAGUUCGAUC .(((...(((....)))...)))((((((((((((..((((((((((((.....)))))))).....)))).((((....)))).)))).))).)))))..((((........)))) ( -46.80, z-score = -0.43, R) >droWil1.scaffold_181089 7532724 117 - 12369635 UGCCGCCGGUUUAUGUGCCCGGCGGUGAUCUCGCUCCUACAAAUCGCGCCUGCGUAGCCAUAUCGAACAACACCAACAUACGUAUCGCCUGGUGUCGCCUGGUGAAUAAGUUCGACA .((((((((.........))))))))......(((.........(((....))).)))....((((((.......((....)).(((((.((.....)).)))))....)))))).. ( -34.20, z-score = 0.14, R) >droVir3.scaffold_13047 16763579 117 + 19223366 AGCCACCCACUGUUGUGCCCGGUGGCGAUUUGGCCAAGGUGCAGCGUGCCGUUUGCAUGUUGUCUAACACCACGGCCAUUGCCGAGGCCUGGGCCCGUCUGGAUCACAAGUUUGACU .(((((((((....)))...))))))(((((((((.(((((((((((((.....))))))))).........((((....))))..)))).)))).....)))))............ ( -46.20, z-score = -0.77, R) >droMoj3.scaffold_6540 20773237 117 - 34148556 AACCACCCACUGUUGUGCCCGGCGGUGAUUUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAACACCACGGCCAUCGCCGAGGCCUGGGCCCGUCUGGAUCACAAGUUUGACU ............(((((.((((((((((..(((((..((((((((((((.....)))))))).....))))..))))))))))).(((....)))...))))..)))))........ ( -45.60, z-score = -0.17, R) >droGri2.scaffold_14906 3192320 117 + 14172833 AGCCACCCACUGUUGUGCCCGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUCUGCAUGUUGUCCAACACCACGGCUAUUGCCGAGGCCUGGGCCCGUUUGGAUCACAAGUUCGACU .(((.(((((....)))...)).)))(((((((((.(((((((((((((.....))))))))).........((((....))))..)))).)))).....)))))............ ( -46.20, z-score = -0.40, R) >anoGam1.chr2R 1623845 117 + 62725911 AGCCCCCGACCGUGGUGCCGGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAACACCACGGCCAUCGCCGAGGCCUGGGCCCGUCUGGACCACAAGUUCGAUC ......(((..(((((.(((((((((((..(((((..((((((((((((.....)))))))).....))))..))))))))))(.(((....)))))))))))))))....)))... ( -57.10, z-score = -1.80, R) >apiMel3.Group12 2438101 117 - 9182753 AACCACCUACUGUUGUACCAGGUGGUGAUCUUGCUAAAGUACAACGAGCUGUUUGCAUGUUAUCCAAUACUACUGCUAUCGCAGAAGCUUGGGCCAGACUUGACCAUAAAUUUGAUU .(((((((((....))...)))))))..(((.(((.((((..((((.((.....)).))))...........((((....))))..)))).))).)))................... ( -27.70, z-score = -0.50, R) >triCas2.ChLG1X 5601001 117 + 8109244 AACCACCCACCGUCGUGCCAGGAGGUGACUUGGCCAAGGUACAACGUGCUGUUUGCAUGUUGUCCAAUACCACUGCUAUCGCCGAAGCUUGGGCUCGUUUGGAUCAUAAAUUCGACU .........(((.((.((((((.(((((..((((...((((((((((((.....))))))))).....)))...)))))))))....))).))).))..)))............... ( -36.50, z-score = -0.87, R) >consensus AGCCACCCACUGUUGUGCCAGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAACACCACGGCUAUCGCCGAGGCCUGGGCCCGUCUGGACCACAAGUUCGACC .................((.(((((...((.((((..((...(((((((.....)))))))..)).......((((....)))).)))).))..))))).))............... (-26.83 = -26.75 + -0.09)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:56:23 2011