
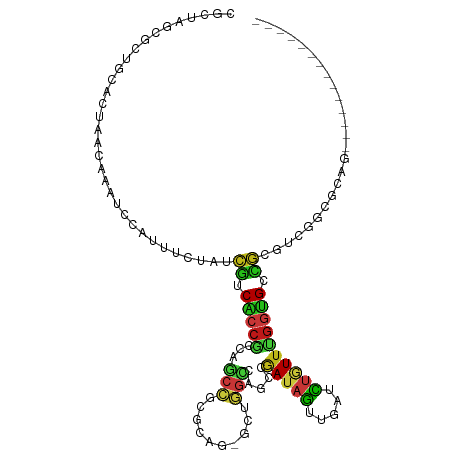
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 3,018,480 – 3,018,577 |
| Length | 97 |
| Max. P | 0.965198 |

| Location | 3,018,480 – 3,018,577 |
|---|---|
| Length | 97 |
| Sequences | 15 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 73.31 |
| Shannon entropy | 0.58524 |
| G+C content | 0.56840 |
| Mean single sequence MFE | -37.69 |
| Consensus MFE | -7.00 |
| Energy contribution | -6.85 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.93 |
| Mean z-score | -3.58 |
| Structure conservation index | 0.19 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.75 |
| SVM RNA-class probability | 0.965198 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 3018480 97 + 27905053 CGCUAGCGCUGCACUAACAAAUCCAUUUCUAUCGUCGCCGCCAGCCGCGCAG-GCUGGCCAGCCGAUAGUUGAUCUGUUCGGUGCCGCGUCGGCGCAG-------------- (((..((((.(((((((((.(((.....((((((.(...(((((((.....)-))))))..).))))))..))).)))).))))).))))..)))...-------------- ( -48.10, z-score = -4.33, R) >droSim1.chr3R 3060768 97 + 27517382 CGCUAGCGCUGCACUAACAAAUCCAUUUCUAUCGUCGCCGCCAGCCGCGCAG-GCUGGCCAGCCGAUAGUUGAUCUGUUCGGUGCCGCGUCGGCGCAG-------------- (((..((((.(((((((((.(((.....((((((.(...(((((((.....)-))))))..).))))))..))).)))).))))).))))..)))...-------------- ( -48.10, z-score = -4.33, R) >droSec1.super_6 3110533 97 + 4358794 CGCUAGCGCUGCACUAACAAAUCCAUUUCUAUCGUCGCCGCCAGCCGCGCAG-GCUGGCCAGCCGAUAGUUGAUCUGUUCGGUGCCGCGUCGGCGCAG-------------- (((..((((.(((((((((.(((.....((((((.(...(((((((.....)-))))))..).))))))..))).)))).))))).))))..)))...-------------- ( -48.10, z-score = -4.33, R) >droYak2.chr3R_random 783062 97 + 3277457 CGCUAACGCUGCACUAACAAAUCCAUUUCUAUCGUCGCCGCCAGCCGCGCAG-GCUGGCCAGCCGAUAGUUGAUCUGUUCGGUGCCGCGUCUGCGCAG-------------- (((..((((.(((((((((.(((.....((((((.(...(((((((.....)-))))))..).))))))..))).)))).))))).))))..)))...-------------- ( -46.00, z-score = -4.79, R) >droEre2.scaffold_4770 3281267 97 + 17746568 CGCUAGCGCUGCACUAACAAAUCCAUUUCUAUCGUCGCCGCCUGCCGCGCAG-GCUGGCCAGCCGAUAGUUGAUCUGUUCGGUGCCGCGUCGGCGCAG-------------- (((..((((.(((((((((.(((.....((((((..(((((((((...))))-)).)))....))))))..))).)))).))))).))))..)))...-------------- ( -46.40, z-score = -3.70, R) >droAna3.scaffold_13340 2791815 91 + 23697760 ------CGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCGCCCAG-GCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCGCAG-------------- ------(((.(((((((((.(((.....((((((.(...(((((((.....)-))))))..).))))))..))).)).))))))).))).........-------------- ( -37.10, z-score = -3.26, R) >dp4.chr2 26430133 97 + 30794189 CUCAAGCGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCUCAUCG-GCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAG-------------- .....((((.(((((((((.(((.....((((((.(...(((((((.....)-))))))..).))))))..))).)).))))))).))))........-------------- ( -38.10, z-score = -4.10, R) >droPer1.super_6 1758888 97 + 6141320 CUCAAGCGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCUCAUCG-GCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAG-------------- .....((((.(((((((((.(((.....((((((.(...(((((((.....)-))))))..).))))))..))).)).))))))).))))........-------------- ( -38.10, z-score = -4.10, R) >droWil1.scaffold_181130 16627016 97 + 16660200 CUCUAGCGCUGCACUAACAAAUCCUUUUCUGUCGUCACCGCCAGCCUCGCAG-GCUGGUCAGCCGAUAGUUGAUCUGUUUGGUGCUGCGUCUGGACAG-------------- .((((((((.(((((((((.(((.....((((((.(...(((((((.....)-))))))..).))))))..))).)).))))))).))).)))))...-------------- ( -42.40, z-score = -4.61, R) >droVir3.scaffold_12855 5816295 97 + 10161210 CGCUAGCGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCUCACAG-GCUGGUCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAG-------------- .((..((((.(((((((((.(((.....((((((.(...(((((((.....)-))))))..).))))))..))).)).))))))).))))..))....-------------- ( -43.40, z-score = -5.32, R) >droMoj3.scaffold_6540 6146674 97 + 34148556 UGCUAGCGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCGCCCAG-GCUGGUCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAG-------------- (((..((((.(((((((((.(((.....((((((.(...(((((((.....)-))))))..).))))))..))).)).))))))).))))..)))...-------------- ( -44.00, z-score = -5.00, R) >droGri2.scaffold_14906 5976064 97 - 14172833 CGCUAGCGCUGCACUAACAAAUCCAUUUCUAUCGUCACCACCAGCCUCGCAG-GCUGGUCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAG-------------- .((..((((.(((((((((.(((.....((((((.(...(((((((.....)-))))))..).))))))..))).)).))))))).))))..))....-------------- ( -43.80, z-score = -5.52, R) >anoGam1.chr3R 49381806 87 + 53272125 ----------GCUAUAACGAAUCCGUUCCUAUCGUCGCCUACUGCUAGU-CA----AAAUAAC--AUCGUCGAUCUUUUCGAAGCACCAAAACCGGCACAGGAU-------- ----------......(((....)))((((...((((.....((((.((-..----.....))--....((((.....)))))))).......))))..)))).-------- ( -12.60, z-score = 0.32, R) >apiMel3.GroupUn 25303909 99 + 399230636 ---------CCCCAGUACGAAUCCAUUUCUUAGUUCACCGACAAACAAUGCU----AAUCAACCAAUUGUAGAUUUAUUUGGUGCACCAUCAACGAUAACGAAUACUGAAAG ---------...(((((.(((.....)))...((.((((((.......(((.----............))).......)))))).))................))))).... ( -13.06, z-score = -0.56, R) >triCas2.ChLG3 17312145 88 - 32080666 UCCCAACCCAUUUUUGAACAAUUCGUCUUCGACGUCGAGCAAAACCACCUCGCAGUCGGCUUCCGA-AAUUGAUUUGUUUGCCGCCGAG----------------------- ...............((((((.(((..((((..(((((((...........))..)))))...)))-)..))).)))))).........----------------------- ( -16.10, z-score = -0.07, R) >consensus CGCUAGCGCUGCACUAACAAAUCCAUUUCUAUCGUCACCGCCAGCCGCGCAG_GCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCGCAG______________ ................................((.(((((...(((..........))).....(((((.....)))))))))).))......................... ( -7.00 = -6.85 + -0.14)

| Location | 3,018,480 – 3,018,577 |
|---|---|
| Length | 97 |
| Sequences | 15 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 73.31 |
| Shannon entropy | 0.58524 |
| G+C content | 0.56840 |
| Mean single sequence MFE | -39.40 |
| Consensus MFE | -10.88 |
| Energy contribution | -11.89 |
| Covariance contribution | 1.01 |
| Combinations/Pair | 1.42 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.28 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.30 |
| SVM RNA-class probability | 0.923553 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 3018480 97 - 27905053 --------------CUGCGCCGACGCGGCACCGAACAGAUCAACUAUCGGCUGGCCAGC-CUGCGCGGCUGGCGGCGACGAUAGAAAUGGAUUUGUUAGUGCAGCGCUAGCG --------------...(((...(((.((((..((((((((..((((((((((.(((((-(.....))))))))))..)))))).....)))))))).)))).)))...))) ( -45.70, z-score = -2.85, R) >droSim1.chr3R 3060768 97 - 27517382 --------------CUGCGCCGACGCGGCACCGAACAGAUCAACUAUCGGCUGGCCAGC-CUGCGCGGCUGGCGGCGACGAUAGAAAUGGAUUUGUUAGUGCAGCGCUAGCG --------------...(((...(((.((((..((((((((..((((((((((.(((((-(.....))))))))))..)))))).....)))))))).)))).)))...))) ( -45.70, z-score = -2.85, R) >droSec1.super_6 3110533 97 - 4358794 --------------CUGCGCCGACGCGGCACCGAACAGAUCAACUAUCGGCUGGCCAGC-CUGCGCGGCUGGCGGCGACGAUAGAAAUGGAUUUGUUAGUGCAGCGCUAGCG --------------...(((...(((.((((..((((((((..((((((((((.(((((-(.....))))))))))..)))))).....)))))))).)))).)))...))) ( -45.70, z-score = -2.85, R) >droYak2.chr3R_random 783062 97 - 3277457 --------------CUGCGCAGACGCGGCACCGAACAGAUCAACUAUCGGCUGGCCAGC-CUGCGCGGCUGGCGGCGACGAUAGAAAUGGAUUUGUUAGUGCAGCGUUAGCG --------------...(((.(((((.((((..((((((((..((((((((((.(((((-(.....))))))))))..)))))).....)))))))).)))).))))).))) ( -50.10, z-score = -4.80, R) >droEre2.scaffold_4770 3281267 97 - 17746568 --------------CUGCGCCGACGCGGCACCGAACAGAUCAACUAUCGGCUGGCCAGC-CUGCGCGGCAGGCGGCGACGAUAGAAAUGGAUUUGUUAGUGCAGCGCUAGCG --------------...(((...(((.((((..((((((((..((((((....(((.((-((((...)))))))))..)))))).....)))))))).)))).)))...))) ( -44.00, z-score = -2.42, R) >droAna3.scaffold_13340 2791815 91 - 23697760 --------------CUGCGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGC-CUGGGCGGCUGGCGGUGACGAUAGGAAUGGAUUUGUUAGUGCAGCG------ --------------((((((((...))))....((((((((..((((((..(.((((((-(.....))))))).)...)))))).....))))))))...))))..------ ( -41.90, z-score = -3.21, R) >dp4.chr2 26430133 97 - 30794189 --------------CUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGC-CGAUGAGGCUGGCGGUGACGAUAGGAAUGGAUUUGUUAGUGCAGCGCUUGAG --------------......((((((.((((..((((((((..((((((..(.((((((-(.....))))))).)...)))))).....)))))))).)))).))).))).. ( -44.40, z-score = -4.24, R) >droPer1.super_6 1758888 97 - 6141320 --------------CUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGC-CGAUGAGGCUGGCGGUGACGAUAGGAAUGGAUUUGUUAGUGCAGCGCUUGAG --------------......((((((.((((..((((((((..((((((..(.((((((-(.....))))))).)...)))))).....)))))))).)))).))).))).. ( -44.40, z-score = -4.24, R) >droWil1.scaffold_181130 16627016 97 - 16660200 --------------CUGUCCAGACGCAGCACCAAACAGAUCAACUAUCGGCUGACCAGC-CUGCGAGGCUGGCGGUGACGACAGAAAAGGAUUUGUUAGUGCAGCGCUAGAG --------------...((.((.(((.((((..((((((((..((.(((((((.(((((-(.....))))))))))..))).)).....)))))))).)))).))))).)). ( -38.80, z-score = -3.34, R) >droVir3.scaffold_12855 5816295 97 - 10161210 --------------CUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGACCAGC-CUGUGAGGCUGGCGGUGACGAUAGGAAUGGAUUUGUUAGUGCAGCGCUAGCG --------------....((...(((.((((..((((((((..((((((((((.(((((-(.....))))))))))..)))))).....)))))))).)))).)))...)). ( -44.30, z-score = -4.05, R) >droMoj3.scaffold_6540 6146674 97 - 34148556 --------------CUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGACCAGC-CUGGGCGGCUGGCGGUGACGAUAGGAAUGGAUUUGUUAGUGCAGCGCUAGCA --------------...(((...(((.((((..((((((((..((((((((((.(((((-(.....))))))))))..)))))).....)))))))).)))).)))...))) ( -44.10, z-score = -3.41, R) >droGri2.scaffold_14906 5976064 97 + 14172833 --------------CUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGACCAGC-CUGCGAGGCUGGUGGUGACGAUAGAAAUGGAUUUGUUAGUGCAGCGCUAGCG --------------....((...(((.((((..((((((((..(((((((((.((((((-(.....))))))))))..)))))).....)))))))).)))).)))...)). ( -41.60, z-score = -3.54, R) >anoGam1.chr3R 49381806 87 - 53272125 --------AUCCUGUGCCGGUUUUGGUGCUUCGAAAAGAUCGACGAU--GUUAUUU----UG-ACUAGCAGUAGGCGACGAUAGGAACGGAUUCGUUAUAGC---------- --------..(((((((..(((..(((((.((((.....))))....--).)))).----.)-))..))).)))).(((((...........))))).....---------- ( -17.40, z-score = 1.22, R) >apiMel3.GroupUn 25303909 99 - 399230636 CUUUCAGUAUUCGUUAUCGUUGAUGGUGCACCAAAUAAAUCUACAAUUGGUUGAUU----AGCAUUGUUUGUCGGUGAACUAAGAAAUGGAUUCGUACUGGGG--------- .(..((((((((((((....))))))...........((((((...((((((.((.----.(((.....)))..)).))))))....)))))).))))))..)--------- ( -23.80, z-score = -1.41, R) >triCas2.ChLG3 17312145 88 + 32080666 -----------------------CUCGGCGGCAAACAAAUCAAUU-UCGGAAGCCGACUGCGAGGUGGUUUUGCUCGACGUCGAAGACGAAUUGUUCAAAAAUGGGUUGGGA -----------------------((((((...........(((((-.((.....((((..((((.........))))..))))....)))))))...........)))))). ( -19.05, z-score = 1.51, R) >consensus ______________CUGCGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGC_CUGCGCGGCUGGCGGUGACGAUAGAAAUGGAUUUGUUAGUGCAGCGCUAGCG .......................(((.(((...(((.((((......(((((..............)))))..(....)..........)))).)))..))).)))...... (-10.88 = -11.89 + 1.01)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:55:33 2011