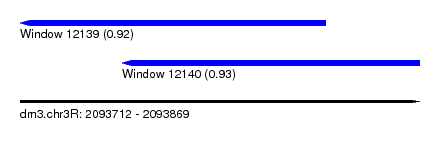
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 2,093,712 – 2,093,869 |
| Length | 157 |
| Max. P | 0.931199 |

| Location | 2,093,712 – 2,093,832 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 121 |
| Reading direction | reverse |
| Mean pairwise identity | 76.98 |
| Shannon entropy | 0.53129 |
| G+C content | 0.53108 |
| Mean single sequence MFE | -44.39 |
| Consensus MFE | -20.60 |
| Energy contribution | -20.29 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.87 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.46 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.28 |
| SVM RNA-class probability | 0.920421 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 2093712 120 - 27905053 GAUGGCAUACCACUCCAUUGCCAUUGUAGCGGGAAAGGCGCUUAUUGUGGGCAAGAUUGCGCUGAUCAUCAGUGCUAUAAUCGGUCUGAAAAAACUUGUGGGUCACGACGGCGGCGAGAA- (((((((...........)))))))...........(.((((..((((((.(..((((((((((.....))))))...))))(((.(....).)))....).)))))).)))).).....- ( -32.90, z-score = 0.76, R) >droWil1.scaffold_181130 5444365 120 + 16660200 GAUGGCCUAUCAUUCGAUUGCCAUUGUGGCGGGCAAGGCUCUGAUUGUGGGCAAAAUUGCUCUUAUCAUUAGCGCCAUCAUUGGCCUCAAGAAGCUUGUUAGCCACGAUGGCGGCGAAAA- ............((((.(((((((((((((..((((((((.((((...(((((....)))))..))))..)))((((....)))).........)))))..)))))))))))))))))..- ( -51.70, z-score = -3.90, R) >droSim1.chr3R 2107598 120 - 27517382 GAUGGCAUACCACUCCAUCGCCAUUGUAGCGGGAAAGGCGCUGAUUGUGGGCAAGAUUGCGCUGAUCAUCAGUGCUAUAAUCGGGCUGAAAAAACUGGUGGGUCACGACGGCGGCGAGAA- ..(((....)))(((..(((((.((((..(......(((((((((.((.(((........))).)).)))))))))...(((((..(....)..)))))..)..)))).))))).)))..- ( -40.10, z-score = -0.76, R) >droSec1.super_6 2179608 120 - 4358794 GAUGGCAUACCACUCCAUCGCCAUUGUAGCGGGAAAGGCGCUGAUUGUGGGCAAGAUUGCGCUGAUCAUCAGUGCUAUAAUCGGGCUAAAAAAACUGGUGGGUCACGACGGCGGCGAGAA- ..(((....)))(((..(((((.((((..(......(((((((((.((.(((........))).)).)))))))))...(((((..........)))))..)..)))).))))).)))..- ( -38.70, z-score = -0.58, R) >droYak2.chr3R 18020252 120 - 28832112 GAUGGCAUACCACUCGAUCGCCAUUGUAGCGGGAAAGGCGUUGAUUGUGGGCAAGAUAGCGCUGAUCAUCAGUGCUAUAAUCGGGCUGAAAAAACUGGUGGGUCACGACGGCGGCGAGAA- ..(((....)))((((.(((((....(((((.......))))).((((((.(...(((((((((.....))))))))).(((((..(....)..))))).).)))))).)))))))))..- ( -43.10, z-score = -2.19, R) >droEre2.scaffold_4770 2336292 120 - 17746568 GAUGGCAUACCACUCGAUCGCCAUUGUGGCGGGAAAGGCGCUGAUUGUGGGCAAGAUAGCGUUGAUCAUUAGUGCUAUAAUCGGGCUGAAAAAACUGGUGGGUCACGACGGCGGCGAGAA- ..(((....)))((((.(((((.(((((((......(((((((((.((.(((........))).)).)))))))))...(((((..(....)..)))))..))))))).)))))))))..- ( -44.60, z-score = -2.79, R) >droAna3.scaffold_13340 5631835 120 + 23697760 AAUGGCCUACCAUUCGAUUGCCAUUGUGGCCGGCAAGGCGUUGAUAGUGGGAAAAAUAGCGCUAAUCAUUAGCGCCAUUAUCGGUUUAAAGAAACUUGUGGGCCACGACGGCGGUGAGAA- ..(((....)))(((.((((((.((((((((.(((((.(.(((((((((.........((((((.....)))))))))))))))......)...))))).)))))))).)))))))))..- ( -46.20, z-score = -3.19, R) >droGri2.scaffold_14624 1449846 120 + 4233967 GAUGGCCUAUCAUUCGAUUGCCAUUGUCGCUGGCAAGGCGCUGAUUGUGGGCAAAAUAGCGUUGAUCAUCAGCGCCAUCAUUGGAUUGAAGAAGCUCGUCAGUCACGAUGGCGGCGAGAA- ((((((..(((....))).)))))).((((((....(((((((((.((.(((........))).)).))))))))).((((((((((((.(.....).)))))).))))))))))))...- ( -50.40, z-score = -3.17, R) >droMoj3.scaffold_6540 11364707 120 - 34148556 GAUGGCCUAUCACUCGAUAGCCAUUGUGGCCGGCAAGGCGCUGAUCGUGGGCAAGAUCGCGCUGAUCAUCAGCGCCAUCAUCGGGCUGAAGAAGCUGGUCAGCCACGACGGCGGCGAGAA- ((((((.((((....))))))))))((.((((....(((((((((.((.(((........))).)).)))))))))....(((((((((.........)))))).))))))).)).....- ( -57.30, z-score = -3.17, R) >droVir3.scaffold_12822 2325433 120 + 4096053 AAUGGCCUACCAUUCGAUUGCCAUUGUGGCUGGCAAGGCGCUGAUUGUGGGCAAAAUCGCGCUGAUCAUCAGCGCCAUCAUUGGGCUAAAGAAGCUGGUCGGUCACGAUGGCGGCGAGAA- ..(((....)))((((.(((((((((((((((((..(((((((((.((.(((........))).)).))))))))).......((((.....)))).)))))))))))))))))))))..- ( -60.10, z-score = -5.06, R) >dp4.chr2 15058984 120 + 30794189 GAUGGCUUAUCACUCGAUCGCCAUUGUGGCUGGCAAGGCUCUGAUUGUGGGCAAAAUAGCGCUCAUCAUCAGCGCCAUCAUCGGACUGAAGAAGCUUGUUGGUCACGACGGGGGCGAGAA- ............((((.((.((.(((((((..(((((((.(((((.((((((........)))))).))))).))).(((......)))......))))..))))))).)).))))))..- ( -49.40, z-score = -3.07, R) >droPer1.super_0 4678503 120 - 11822988 GAUGGCUUAUCAUUCGAUCGCCAUUGUGGCUGGCAAGGCUUUGAUUGUGGGCAAAAUAGCUCUCAUCAUCAGCGCCAUCAUCGGUCUGAAGAAGCUUGUUGGCCACGACGGGGGCGAGAA- ............((((.((.((.(((((((..(((((.(((((((.(.((((......)))).)))).((((.(((......))))))).)))))))))..))))))).)).))))))..- ( -48.40, z-score = -2.82, R) >anoGam1.chr2R 10335268 120 + 62725911 CAUGGUCUACCACUCGAUCGCCAUCGUCGCCGGUAAGGCGCUCAUCAUCGGCAAGAUCGCCCUGGUCAUCUCCGCCAUCAUCGGUCUGAAGAAGCUCGUCACCCCG-GAGGGACACGAGAA ..(((....)))((((....((.....((((.....))))(((......(((.((((.((....)).))))..))).....(((..(((.(.....).)))..)))-)))))...)))).. ( -34.90, z-score = 1.01, R) >apiMel3.Group15 5438285 117 - 7856270 GUUGGCGUUGGGCGCGAUCGCCGCCAUCGCCGGGAAAGCGCUCCUCGUGGGGAAAAUCGCUCUGGUCAUCUCGGCCAUCAUCGGGUUGAAGAAGUUGCUUAGCUCGAGCGGGAAACA---- .((((((...(((((....).))))..)))))).......((((....)))).....((((((((((.....))))).....(((((((.(......)))))))))))))(....).---- ( -41.50, z-score = 0.88, R) >triCas2.ChLG9 903425 117 + 15222296 GAUGGCUUAUCAGGGAAUUGCUGUGAUGUCUGGAACUGCAAUUAUUGUCGGAAAAAUGGCUCUAAUUUUGAGUGCAAUUUUGGGUUUGAAAAAACUAGUUAGU---AGUGGCCAAGAAAA- ..((((((((((.((.....)).))))).(..(((.((((.(((..((..((........))..))..))).)))).)))..)((((....))))........---...)))))......- ( -26.50, z-score = -0.80, R) >consensus GAUGGCCUACCACUCGAUCGCCAUUGUGGCGGGCAAGGCGCUGAUUGUGGGCAAAAUAGCGCUGAUCAUCAGCGCCAUCAUCGGGCUGAAGAAGCUGGUGGGUCACGACGGCGGCGAGAA_ ((((((.............))))))...........(((((((((.((.(((........))).)).))))))))).........................(((.....)))......... (-20.60 = -20.29 + -0.30)

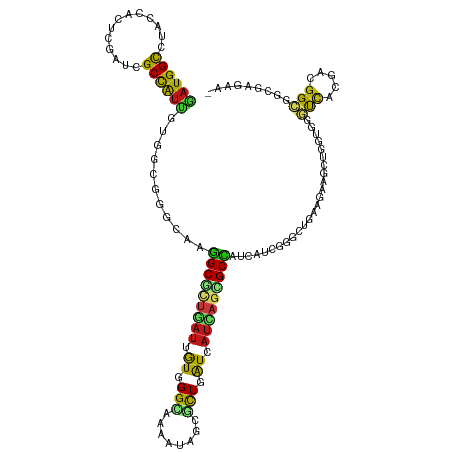
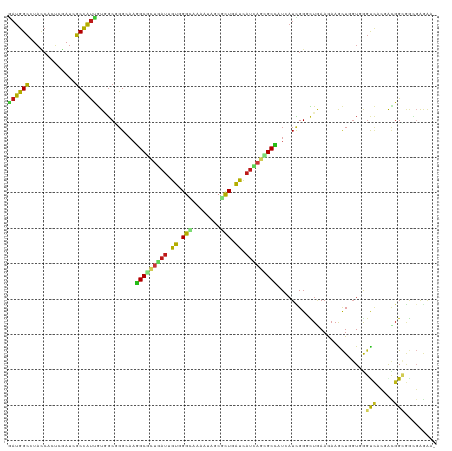
| Location | 2,093,752 – 2,093,869 |
|---|---|
| Length | 117 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.21 |
| Shannon entropy | 0.55304 |
| G+C content | 0.51171 |
| Mean single sequence MFE | -39.76 |
| Consensus MFE | -24.68 |
| Energy contribution | -24.97 |
| Covariance contribution | 0.30 |
| Combinations/Pair | 1.81 |
| Mean z-score | -1.22 |
| Structure conservation index | 0.62 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.36 |
| SVM RNA-class probability | 0.931199 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 2093752 117 - 27905053 UCAUUGCCGCCGUUCUGUUAAAG---ACCGCUAUUCUAAAGAUGGCAUACCACUCCAUUGCCAUUGUAGCGGGAAAGGCGCUUAUUGUGGGCAAGAUUGCGCUGAUCAUCAGUGCUAUAA ((.((((((((..(((.....))---)(((((((......(((((((...........))))))))))))))....)))((.....)).)))))))..((((((.....))))))..... ( -40.80, z-score = -2.59, R) >droWil1.scaffold_181130 5444405 117 + 16660200 UCAUUGCGGCUGUGCUGCUAAAA---ACAGCCAUUUUAAAGAUGGCCUAUCAUUCGAUUGCCAUUGUGGCGGGCAAGGCUCUGAUUGUGGGCAAAAUUGCUCUUAUCAUUAGCGCCAUCA .......((((((..........---))))))........(((((((((..........(((.....)))((((((.((((.......))))....)))))).......))).)))))). ( -40.20, z-score = -1.79, R) >droSim1.chr3R 2107638 117 - 27517382 UCAUUGCCGCCGUUCUGCUAAAG---ACCGCUAUUCUAAAGAUGGCAUACCACUCCAUCGCCAUUGUAGCGGGAAAGGCGCUGAUUGUGGGCAAGAUUGCGCUGAUCAUCAGUGCUAUAA ((.((((((((..(((.....))---)(((((((......(((((.........)))))......)))))))....)))((.....)).)))))))..((((((.....))))))..... ( -39.80, z-score = -1.73, R) >droSec1.super_6 2179648 117 - 4358794 UCAUUGCCGCCGUUCUGCUAAAG---ACCGCUAUUCUAAAGAUGGCAUACCACUCCAUCGCCAUUGUAGCGGGAAAGGCGCUGAUUGUGGGCAAGAUUGCGCUGAUCAUCAGUGCUAUAA ((.((((((((..(((.....))---)(((((((......(((((.........)))))......)))))))....)))((.....)).)))))))..((((((.....))))))..... ( -39.80, z-score = -1.73, R) >droYak2.chr3R 18020292 117 - 28832112 UCAUUGCCGCCGUUCUGCUUAAG---ACCGCAAUACUGAAGAUGGCAUACCACUCGAUCGCCAUUGUAGCGGGAAAGGCGUUGAUUGUGGGCAAGAUAGCGCUGAUCAUCAGUGCUAUAA ...(((((((((((.(((.....---...)))........))))))....(((((((.((((.((.(....).)).))))))))..)))))))).(((((((((.....))))))))).. ( -38.20, z-score = -1.18, R) >droEre2.scaffold_4770 2336332 117 - 17746568 UCAUUGCCGCCGUUCUGCUGAAG---ACCGCUAUACUGAAGAUGGCAUACCACUCGAUCGCCAUUGUGGCGGGAAAGGCGCUGAUUGUGGGCAAGAUAGCGUUGAUCAUUAGUGCUAUAA ...((((((((..(((.....))---)(((((((......((((((.............)))))))))))))....)))((.....)).))))).((((((((((...)))))))))).. ( -34.52, z-score = 0.13, R) >droAna3.scaffold_13340 5631875 117 + 23697760 UCAUUGCUGCCGUUCUUCUAAAA---ACAGCUAUCCUGAAAAUGGCCUACCAUUCGAUUGCCAUUGUGGCCGGCAAGGCGUUGAUAGUGGGAAAAAUAGCGCUAAUCAUUAGCGCCAUUA ...(((((((((...........---.(((.....)))..((((((.............)))))).))).))))))(((((((((((((..........)))))....)))))))).... ( -30.72, z-score = -0.20, R) >droGri2.scaffold_14624 1449886 117 + 4233967 UUAUUGCCGCUGUGCUCCUGAAG---ACUGCCAUACUUAAGAUGGCCUAUCAUUCGAUUGCCAUUGUCGCUGGCAAGGCGCUGAUUGUGGGCAAAAUAGCGUUGAUCAUCAGCGCCAUCA ...(((((((((.((........---...)))).......((((((..(((....))).))))))...)).)))))(((((((((.((.(((........))).)).))))))))).... ( -39.90, z-score = -1.38, R) >droMoj3.scaffold_6540 11364747 117 - 34148556 UCAUAGCCGCCGUGCUGCUCAAG---ACGGCCAUACUGAAGAUGGCCUAUCACUCGAUAGCCAUUGUGGCCGGCAAGGCGCUGAUCGUGGGCAAGAUCGCGCUGAUCAUCAGCGCCAUCA ........(((((.((.....))---))((((((......((((((.((((....)))))))))))))))))))..(((((((((.((.(((........))).)).))))))))).... ( -50.70, z-score = -2.48, R) >droVir3.scaffold_12822 2325473 117 + 4096053 UCAUAGCCGCCGUGCUGCUAAAG---ACGGCCAUACUGAAAAUGGCCUACCAUUCGAUUGCCAUUGUGGCUGGCAAGGCGCUGAUUGUGGGCAAAAUCGCGCUGAUCAUCAGCGCCAUCA ...((((((((((.((.....))---))))).........((((((.............))))))..)))))....(((((((((.((.(((........))).)).))))))))).... ( -45.32, z-score = -1.92, R) >dp4.chr2 15059024 117 + 30794189 UCAUCGCCGCCGUUCUGCUGAAG---ACGGCCAUUCUGAAGAUGGCUUAUCACUCGAUCGCCAUUGUGGCUGGCAAGGCUCUGAUUGUGGGCAAAAUAGCGCUCAUCAUCAGCGCCAUCA .....((((((((.((.....))---))))).........((((((..(((....))).))))))......)))..(((.(((((.((((((........)))))).))))).))).... ( -43.80, z-score = -1.79, R) >droPer1.super_0 4678543 117 - 11822988 UCAUCGCCGCCGUUCUGCUGAAG---ACGGCCAUUCUGAAGAUGGCUUAUCAUUCGAUCGCCAUUGUGGCUGGCAAGGCUUUGAUUGUGGGCAAAAUAGCUCUCAUCAUCAGCGCCAUCA .....((((((((.((.....))---))))).........((((((..(((....))).))))))......)))..(((.(((((((.((((......)))).))..))))).))).... ( -38.20, z-score = -0.67, R) >anoGam1.chr2R 10335308 117 + 62725911 UCCUCGCUGCAAUGGCUAUCAAG---GGAGGUAUCCUGACCAUGGUCUACCACUCGAUCGCCAUCGUCGCCGGUAAGGCGCUCAUCAUCGGCAAGAUCGCCCUGGUCAUCUCCGCCAUCA .....((.((.(((((.(((..(---((.((((....(((....))))))).)))))).))))).)).(((.....)))))........(((.((((.((....)).))))..))).... ( -35.50, z-score = -0.17, R) >apiMel3.Group15 5438322 120 - 7856270 UGAUGAUGGCCCUGGCCCUCAAGGCUGCAGCCCUGUUGCCGUUGGCGUUGGGCGCGAUCGCCGCCAUCGCCGGGAAAGCGCUCCUCGUGGGGAAAAUCGCUCUGGUCAUCUCGGCCAUCA ...((((((((.((((((((..(((.(((....))).)))...((((...(((((....).))))..)))))))..((((.((((....))))....))))..)))))....)))))))) ( -52.30, z-score = -0.53, R) >triCas2.ChLG9 903462 117 + 15222296 UAUUGGGAGCACUAGCAAUUAAG---GGGACUUUUUUGGCGAUGGCUUAUCAGGGAAUUGCUGUGAUGUCUGGAACUGCAAUUAUUGUCGGAAAAAUGGCUCUAAUUUUGAGUGCAAUUU ........(((((...(((((.(---((...((((((((((((((..(((((.((.....)).)))))..((......)).))))))))))))))....))))))))...)))))..... ( -26.60, z-score = -0.22, R) >consensus UCAUUGCCGCCGUUCUGCUAAAG___ACCGCCAUUCUGAAGAUGGCCUACCACUCGAUCGCCAUUGUGGCGGGCAAGGCGCUGAUUGUGGGCAAAAUAGCGCUGAUCAUCAGCGCCAUCA ...........................(((((((......((((((.............)))))))))))))....(((((((((.((.(((........))).)).))))))))).... (-24.68 = -24.97 + 0.30)
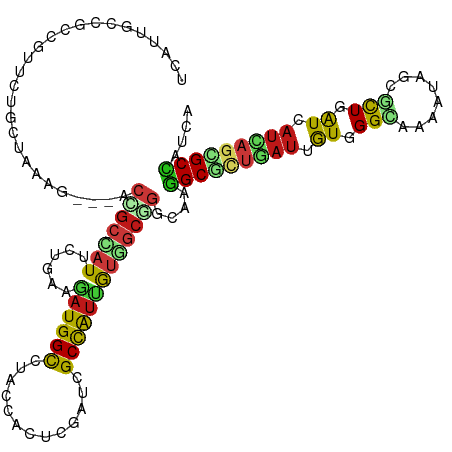
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:53:14 2011