
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 1,119,384 – 1,119,491 |
| Length | 107 |
| Max. P | 0.950665 |

| Location | 1,119,384 – 1,119,491 |
|---|---|
| Length | 107 |
| Sequences | 14 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 83.37 |
| Shannon entropy | 0.39374 |
| G+C content | 0.56136 |
| Mean single sequence MFE | -40.81 |
| Consensus MFE | -25.34 |
| Energy contribution | -25.06 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.46 |
| Mean z-score | -2.14 |
| Structure conservation index | 0.62 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.950665 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
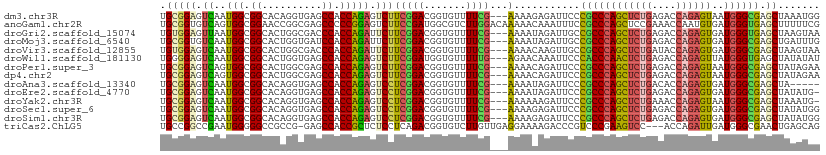
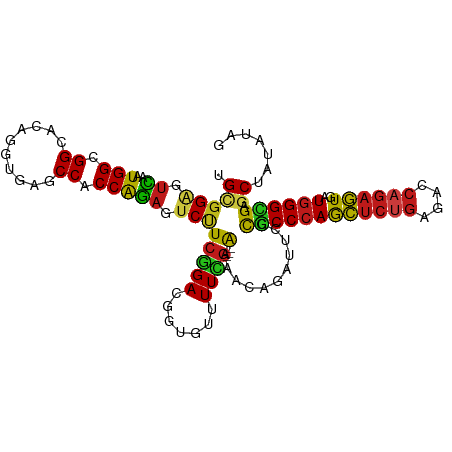
>dm3.chr3R 1119384 107 + 27905053 UGCGGAGUCAAUGGCGGCACAGGUGAGCCACCAGAGUCUUCGGACGGUGUUUUCG---AAAAGAGAUUCCCGCCCAGCUCUGAGACCAGAGUAAUGGGCGAGCUAAAUGG .((((((((..(((.(((........))).)))..(((....)))..........---......))))))((((((((((((....))))))..)))))).))....... ( -44.30, z-score = -3.35, R) >anoGam1.chr2R 53352545 110 - 62725911 UGCGGUGUCAGUGGCGGAACCGGCGAGCCCCCGGAGUCUUCCGAUGGCGUCUUGGACAAAAACAAAUUUCCGCCCAGCUCCGAAACCAAUGUGAUGGGUGAGCUUUUUCG ..........(.(((((((((((.(((((..(((......)))..))).))))))............))))))))(((((.....(((......)))..)))))...... ( -31.30, z-score = 1.25, R) >droGri2.scaffold_15074 3395644 107 - 7742996 UGUGGAGUUAAUGGCGGCACUGGCGACCCACCAGAUUCUUCGGACGGUGUUUUCG---AAAAUAGAUUGCCGCCCAGCUCUGAGACCAGAGUGAUGGGUGAGCUAAGUAA ...........(((((((((((.(((..((((....((....)).))))...)))---....)))..))))(((((((((((....))))))..)))))..))))..... ( -35.20, z-score = -1.26, R) >droMoj3.scaffold_6540 11134741 107 - 34148556 UGCGGUGUCAAUGGCGGCACUGGUGAUCCACCAGAUUCUUCGGACGGUGUUUUCG---AAAAUAGAUUGCCGCCCAGCUCUGAGACCAGAGUGAUGGGCGAGCUGAUUUG ..(((((((......))))))).(((..((((....((....)).))))...)))---....((((((((((((((((((((....))))))..)))))).)).)))))) ( -40.00, z-score = -1.92, R) >droVir3.scaffold_12855 2367823 107 - 10161210 UGUGGAGUCAAUGGCGGCACUGGCGACCCACCAGAUUCUUCGGACGGUGUUUUCG---AAAACAAGUUGCCGCCCAGCUCUGAUACCAGAGUGAUGGGCGAGCUAAGUAA ...........(((((((((((.(((..((((....((....)).))))...)))---....).)).))))(((((((((((....))))))..)))))..))))..... ( -37.30, z-score = -1.31, R) >droWil1.scaffold_181130 3851162 107 - 16660200 UGGGGAGUCAAUGGCGGCACUGGUGAGCCACCAGAGUCUUCGGAUGGUGUUUUUG---AGAACAAAUUCCCACCCAACUCUGAGACCAGAGUUAUGGGUGAGCUAUAUAU .(((((.((..(((.(((.(....).))).))))).)))))(((...(((((...---.)))))...)))((((((((((((....))))))..)))))).......... ( -40.30, z-score = -2.73, R) >droPer1.super_3 2924750 107 - 7375914 UGCGGAGUCAGUGGCGGCACUGGCGAGCCACCAGAGUCUUCGGACGGUGUUUUCG---AAAACAGAUUCCCGCCCAGCUCUGAGACCAGAGUAAUGGGCGAGCUAUAGAA .((((((((.(((((.((....))..)))))....(((....)))..(((((...---.)))))))))))((((((((((((....))))))..)))))).))....... ( -46.60, z-score = -3.52, R) >dp4.chr2 9127851 107 - 30794189 UGCGGAGUCAGUGGCGGCACUGGCGAGCCACCAGAGUCUUCGGACGGUGUUUUCG---AAAACAGAUUCCCGCCCAGCUCUGAGACCAGAGUAAUGGGCGAGCUAUAGAA .((((((((.(((((.((....))..)))))....(((....)))..(((((...---.)))))))))))((((((((((((....))))))..)))))).))....... ( -46.60, z-score = -3.52, R) >droAna3.scaffold_13340 23301759 102 + 23697760 UGCGGAGUCAAUGGCGGCACAGGUGAGCCACCAGAGUCCUCGGACGGUGUUUUCG---AAAAUAGAUUCCCGCCCAGCUCUGACACCAGAGUGAUGGGCGAGCUA----- .((((((((..(((.(((........))).)))..(((....)))..........---......))))))((((((((((((....))))))..)))))).))..----- ( -42.40, z-score = -2.80, R) >droEre2.scaffold_4770 1394109 106 + 17746568 UGCGGAGUCAAUGGCGGCACAGGUGAGCCACCAGAGUCCUCGGACGGUGUUUUCG---AAAAUAGAUUCCCGCCCAGCUCUGAGACCAGAGUGAUGGGCGAGCUAUAUG- .((((((((..(((.(((........))).)))..(((....)))..........---......))))))((((((((((((....))))))..)))))).))......- ( -42.40, z-score = -2.83, R) >droYak2.chr3R 1499295 106 + 28832112 UGCGGAGUCAAUGGCGGCACAGGUGAGCCACCAGAGUCCUCGGACGGUGUUUUCG---AAAAAAGAUUCCCGCCCAGCUCUGAAACCAGAGUAAUGGGCGAGCUAAAUG- .((((((((..(((.(((........))).)))..(((....)))..........---......))))))((((((((((((....))))))..)))))).))......- ( -42.40, z-score = -3.40, R) >droSec1.super_6 1240730 107 + 4358794 UGCGGAGUCAAUGGCGGCACAGGUGAGCCACCAGAGUCCUCGGACGGUGUUUUCG---AAAAGAGAUUCCCGCCCAGCUCUGAGACCAGAGUGAUGGGCGAGCUAUAUGG .((((((((..(((.(((........))).)))..(((....)))..........---......))))))((((((((((((....))))))..)))))).))....... ( -42.40, z-score = -2.34, R) >droSim1.chr3R 1143699 107 + 27517382 UGCGGAGUCAAUGGCGGCACAGGUGAGCCACCAGAGUCCUCGGACGGUGUUUUCG---AAAAGAGAUUCCCGCCCAGCUCUGAGACCAGAGUGAUGGGCGAGCUAUAUGG .((((((((..(((.(((........))).)))..(((....)))..........---......))))))((((((((((((....))))))..)))))).))....... ( -42.40, z-score = -2.34, R) >triCas2.ChLG5 18069821 106 + 18847211 UGCCGGCCGAAUGGGGGCCGCCG-GAGCCACCGCUCUCCUCAGACGGUGUCUUGUUGAGGAAAAGACCCGUCCCGAAGUCC---ACCAGAUUGAUGGGCGAACUGAGCAG .(.(((((.......))))).)(-(.....))((((...((.(((((.(((((.........)))))))))).....((((---(.(.....).)))))))...)))).. ( -37.70, z-score = 0.07, R) >consensus UGCGGAGUCAAUGGCGGCACAGGUGAGCCACCAGAGUCUUCGGACGGUGUUUUCG___AAAACAGAUUCCCGCCCAGCUCUGAGACCAGAGUGAUGGGCGAGCUAUAUAG .(((((.((..(((.((..........)).))))).)))...............................((((((((((((....))))))..)))))).))....... (-25.34 = -25.06 + -0.28)
| Location | 1,119,384 – 1,119,491 |
|---|---|
| Length | 107 |
| Sequences | 14 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 83.37 |
| Shannon entropy | 0.39374 |
| G+C content | 0.56136 |
| Mean single sequence MFE | -38.67 |
| Consensus MFE | -21.45 |
| Energy contribution | -21.86 |
| Covariance contribution | 0.41 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.55 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.30 |
| SVM RNA-class probability | 0.923336 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
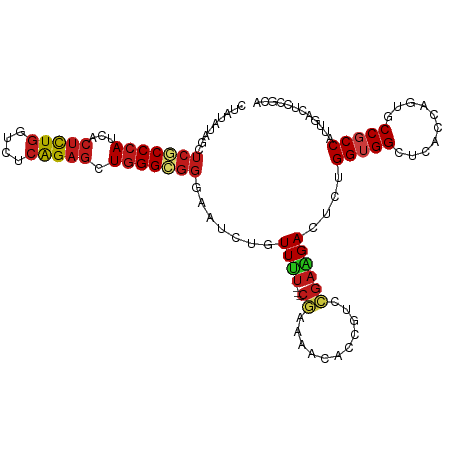
>dm3.chr3R 1119384 107 - 27905053 CCAUUUAGCUCGCCCAUUACUCUGGUCUCAGAGCUGGGCGGGAAUCUCUUUU---CGAAAACACCGUCCGAAGACUCUGGUGGCUCACCUGUGCCGCCAUUGACUCCGCA .......((.((((((...(((((....))))).))))))(((.((..((((---((...........))))))...(((((((.(....).)))))))..)).))))). ( -39.00, z-score = -2.90, R) >anoGam1.chr2R 53352545 110 + 62725911 CGAAAAAGCUCACCCAUCACAUUGGUUUCGGAGCUGGGCGGAAAUUUGUUUUUGUCCAAGACGCCAUCGGAAGACUCCGGGGGCUCGCCGGUUCCGCCACUGACACCGCA .......((....(((......)))...((((((((((((((((....)))))))))..((.(((..((((....))))..)))))...)))))))...........)). ( -35.80, z-score = -0.08, R) >droGri2.scaffold_15074 3395644 107 + 7742996 UUACUUAGCUCACCCAUCACUCUGGUCUCAGAGCUGGGCGGCAAUCUAUUUU---CGAAAACACCGUCCGAAGAAUCUGGUGGGUCGCCAGUGCCGCCAUUAACUCCACA .....((((((..(((......))).....))))))(((((((.....((((---((...........))))))..((((((...)))))))))))))............ ( -33.10, z-score = -2.13, R) >droMoj3.scaffold_6540 11134741 107 + 34148556 CAAAUCAGCUCGCCCAUCACUCUGGUCUCAGAGCUGGGCGGCAAUCUAUUUU---CGAAAACACCGUCCGAAGAAUCUGGUGGAUCACCAGUGCCGCCAUUGACACCGCA (((..((((((..(((......))).....))))))(((((((.....((((---((...........))))))..((((((...))))))))))))).)))........ ( -36.70, z-score = -2.59, R) >droVir3.scaffold_12855 2367823 107 + 10161210 UUACUUAGCUCGCCCAUCACUCUGGUAUCAGAGCUGGGCGGCAACUUGUUUU---CGAAAACACCGUCCGAAGAAUCUGGUGGGUCGCCAGUGCCGCCAUUGACUCCACA .....((((((..(((......))).....))))))(((((((.....((((---((...........))))))..((((((...)))))))))))))............ ( -34.10, z-score = -1.20, R) >droWil1.scaffold_181130 3851162 107 + 16660200 AUAUAUAGCUCACCCAUAACUCUGGUCUCAGAGUUGGGUGGGAAUUUGUUCU---CAAAAACACCAUCCGAAGACUCUGGUGGCUCACCAGUGCCGCCAUUGACUCCCCA ..........((((((..((((((....))))))))))))(((...((((..---....))))...)))...((.(((((((((........)))))))..)).)).... ( -34.30, z-score = -2.28, R) >droPer1.super_3 2924750 107 + 7375914 UUCUAUAGCUCGCCCAUUACUCUGGUCUCAGAGCUGGGCGGGAAUCUGUUUU---CGAAAACACCGUCCGAAGACUCUGGUGGCUCGCCAGUGCCGCCACUGACUCCGCA .......((.((((((...(((((....))))).))))))(((.((.(((((---((...........)))))))..(((((((.(....).)))))))..)).))))). ( -42.60, z-score = -3.14, R) >dp4.chr2 9127851 107 + 30794189 UUCUAUAGCUCGCCCAUUACUCUGGUCUCAGAGCUGGGCGGGAAUCUGUUUU---CGAAAACACCGUCCGAAGACUCUGGUGGCUCGCCAGUGCCGCCACUGACUCCGCA .......((.((((((...(((((....))))).))))))(((.((.(((((---((...........)))))))..(((((((.(....).)))))))..)).))))). ( -42.60, z-score = -3.14, R) >droAna3.scaffold_13340 23301759 102 - 23697760 -----UAGCUCGCCCAUCACUCUGGUGUCAGAGCUGGGCGGGAAUCUAUUUU---CGAAAACACCGUCCGAGGACUCUGGUGGCUCACCUGUGCCGCCAUUGACUCCGCA -----..((.((((((...(((((....))))).))))))(((.((......---..........(((....)))..(((((((.(....).)))))))..)).))))). ( -39.80, z-score = -2.37, R) >droEre2.scaffold_4770 1394109 106 - 17746568 -CAUAUAGCUCGCCCAUCACUCUGGUCUCAGAGCUGGGCGGGAAUCUAUUUU---CGAAAACACCGUCCGAGGACUCUGGUGGCUCACCUGUGCCGCCAUUGACUCCGCA -......((.((((((...(((((....))))).))))))(((.((......---..........(((....)))..(((((((.(....).)))))))..)).))))). ( -39.80, z-score = -2.67, R) >droYak2.chr3R 1499295 106 - 28832112 -CAUUUAGCUCGCCCAUUACUCUGGUUUCAGAGCUGGGCGGGAAUCUUUUUU---CGAAAACACCGUCCGAGGACUCUGGUGGCUCACCUGUGCCGCCAUUGACUCCGCA -......((.((((((...(((((....))))).))))))(((.((......---..........(((....)))..(((((((.(....).)))))))..)).))))). ( -39.80, z-score = -2.92, R) >droSec1.super_6 1240730 107 - 4358794 CCAUAUAGCUCGCCCAUCACUCUGGUCUCAGAGCUGGGCGGGAAUCUCUUUU---CGAAAACACCGUCCGAGGACUCUGGUGGCUCACCUGUGCCGCCAUUGACUCCGCA .......((.((((((...(((((....))))).))))))(((.((......---..........(((....)))..(((((((.(....).)))))))..)).))))). ( -39.80, z-score = -2.49, R) >droSim1.chr3R 1143699 107 - 27517382 CCAUAUAGCUCGCCCAUCACUCUGGUCUCAGAGCUGGGCGGGAAUCUCUUUU---CGAAAACACCGUCCGAGGACUCUGGUGGCUCACCUGUGCCGCCAUUGACUCCGCA .......((.((((((...(((((....))))).))))))(((.((......---..........(((....)))..(((((((.(....).)))))))..)).))))). ( -39.80, z-score = -2.49, R) >triCas2.ChLG5 18069821 106 - 18847211 CUGCUCAGUUCGCCCAUCAAUCUGGU---GGACUUCGGGACGGGUCUUUUCCUCAACAAGACACCGUCUGAGGAGAGCGGUGGCUC-CGGCGGCCCCCAUUCGGCCGGCA ((((((.......(((((.....)))---)).((((((.(((((((((.........))))).)))))))))).))))))......-.(.(((((.......))))).). ( -44.20, z-score = -1.15, R) >consensus CUAUAUAGCUCGCCCAUCACUCUGGUCUCAGAGCUGGGCGGGAAUCUGUUUU___CGAAAACACCGUCCGAAGACUCUGGUGGCUCACCAGUGCCGCCAUUGACUCCGCA ......(((((..(((......))).....)))))((((((......................)))))).........(((((..........)))))............ (-21.45 = -21.86 + 0.41)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:51:11 2011