
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 1,118,482 – 1,118,587 |
| Length | 105 |
| Max. P | 0.875111 |

| Location | 1,118,482 – 1,118,585 |
|---|---|
| Length | 103 |
| Sequences | 15 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 73.22 |
| Shannon entropy | 0.63118 |
| G+C content | 0.48797 |
| Mean single sequence MFE | -30.51 |
| Consensus MFE | -13.33 |
| Energy contribution | -14.48 |
| Covariance contribution | 1.15 |
| Combinations/Pair | 1.52 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.44 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.02 |
| SVM RNA-class probability | 0.875111 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
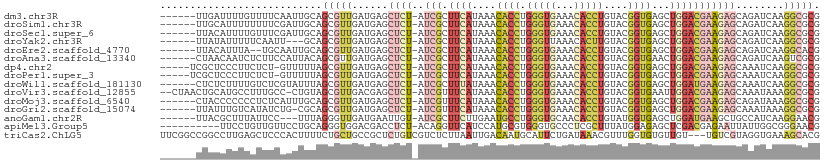
>dm3.chr3R 1118482 103 - 27905053 ------UUGAUUUUGUUUUCAAUUGCAGCGUUGAUGAGCUCU-AUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAUCAAGGCGCG ------((((........))))..((.((.(((((..(((((-.(((.((((....((((((((((...))))))...))))...))))))))))))..))))).)))). ( -33.60, z-score = -2.13, R) >droSim1.chr3R 1142782 103 - 27517382 ------UUGCAUUUUUUUUCGAUUGCAGCGUUGAUGAGCUCU-AUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAUCAAGGCGCG ------.((((.((......)).))))((.(((((..(((((-.(((.((((....((((((((((...))))))...))))...))))))))))))..))))).))... ( -33.70, z-score = -2.00, R) >droSec1.super_6 1239789 103 - 4358794 ------UUACAUUUUGUUUCGAUUGCAGCGUUGAUGAGCUCU-AUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAUCAAGGCGCG ------..................((.((.(((((..(((((-.(((.((((....((((((((((...))))))...))))...))))))))))))..))))).)))). ( -32.60, z-score = -1.82, R) >droYak2.chr3R 1498189 100 - 28832112 ------UUAUAUUUUUCAAUU---GCAGCGUUGAUGAGCUCU-AUCGCUUCAUAAACACCUGGGUUAAACACUUGUACGGUGAGCUGGACGAAGAGCAGAUCAAGGCGCG ------...............---((.((.(((((..(((((-.(((.((((....(((((((((.....)))))...))))...))))))))))))..))))).)))). ( -27.90, z-score = -1.45, R) >droEre2.scaffold_4770 1393271 101 - 17746568 ------UUACAUUUA--UGCAAUUGCAGCGUUGAUGAGCUCU-AUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAUCAAGGCACG ------.........--(((....)))((.(((((..(((((-.(((.((((....((((((((((...))))))...))))...))))))))))))..))))).))... ( -33.10, z-score = -2.78, R) >droAna3.scaffold_13340 23300945 103 - 23697760 ------CUAACAAUCUCUUCCAUUACAGCGUUGAUGAGCUCU-AUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAACUGGACGAAGAGCAGAUCAAGUCGCG ------.....................((((((((..(((((-.(((.((((....((((((((((...))))))...))))...))))))))))))..)))))..))). ( -28.90, z-score = -1.95, R) >dp4.chr2 9126979 103 + 30794189 -----UCGCUCCCUUCUCU-GUUUUUAGCGUUGAUGAGCUCU-AUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAAAUCAAGGCGCG -----.(((...(......-)......((.(((((..(((((-.(((.((((....((((((((((...))))))...))))...))))))))))))..))))).))))) ( -31.50, z-score = -1.39, R) >droPer1.super_3 2923879 103 + 7375914 -----UCGCUCCCUUCUCU-GUUUUUAGCGUUGAUGAGCUCU-AUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAAAUCAAGGCGCG -----.(((...(......-)......((.(((((..(((((-.(((.((((....((((((((((...))))))...))))...))))))))))))..))))).))))) ( -31.50, z-score = -1.39, R) >droWil1.scaffold_181130 3850376 103 + 16660200 ------CUCUCUUUUGUCUCGUAUUUAGCGUUGAUGAGCUCU-AUCGCUUUAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGAUGAAGAGCAAAUCAAGGCGCG ------.....................((.(((((..(((((-((((((.......(((....)))...((((.....)))))))..)))..)))))..))))).))... ( -27.80, z-score = -0.82, R) >droVir3.scaffold_12855 2367028 106 + 10161210 --CUAACUGCAUGCCUUUGCC-CUGUAGCGUUGACGAGCUCU-AUCGUUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAAUUGGACGAAGAGCAAAUAAAGGCGCG --......((..(((((((..-......((....)).(((((-.(((((.((....((((((((((...))))))...))))...))))))))))))...))))))))). ( -31.60, z-score = -1.88, R) >droMoj3.scaffold_6540 11133941 103 + 34148556 ------CUACCCCCCCUCUCAUUUGCAGCGUUGAUGAGCUCU-AUCGUUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAUAAAGGCGCG ------.....................(((((.((..(((((-.(((((.((....((((((((((...))))))...))))...))))))))))))..))...))))). ( -27.90, z-score = -0.61, R) >droGri2.scaffold_15074 3394772 102 + 7742996 ------UUAUUUGUCAUAUCUG-CGCAGCGUUGAUGAGCUCU-AUCGUUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAAAUAAAGGCGCG ------................-....(((((.((..(((((-.(((((.((....((((((((((...))))))...))))...))))))))))))..))...))))). ( -28.30, z-score = -0.83, R) >anoGam1.chr2R 53349214 100 + 62725911 ------UUACGCUUUAUUCC---UUUAGGGUUGAUGAAUUGU-AUCGCUUCUUGAAUGCCUGGGUGCAACACCUGUAUGGUGAGCUGGAUGAAGCUGCCAUCAAGGAACG ------.((((.((((((((---.....))..)))))).)))-).((.(((((((((((..(((((...)))))))))(((.((((......))))))).))))))).)) ( -27.10, z-score = -0.80, R) >apiMel3.Group5 6096509 99 - 13386189 ----------UUCCUGUUGUUCCUGCAGGGUGGACGACCUCU-ACAGGUUCAUCCAUGCGUGGGUGCCCUCGCUUUAUGGAGAGCUCGACGAGAAUUAUUGGCGGGAACG ----------........((((((((...(((((.....)))-)).(((((.((((((((.((....)).)))...))))))))))...............)))))))). ( -36.20, z-score = -1.39, R) >triCas2.ChLG5 18067420 107 - 18847211 UUCGGCCGGCCUUGAGCUCCCACUUUUCUGCUGCCGCUCUGUCGUCUCUUAAUUGACAAUGCAUUCUGAUAAACGUUUGGUGUGUUGU---UGUCGUAGGUGAAAGCACG ..((((.(((...(((......)))....)))))))...(((..((.((((..((((((((((((..(((....))).))))))))))---))...)))).))..))).. ( -25.90, z-score = -0.00, R) >consensus ______CUACCUUUUUUUUCGAUUGCAGCGUUGAUGAGCUCU_AUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAUCAAGGCGCG ...........................(((((......((((..(((.((((....((((.(((((...)))))....))))...)))))))))))........))))). (-13.33 = -14.48 + 1.15)


| Location | 1,118,491 – 1,118,587 |
|---|---|
| Length | 96 |
| Sequences | 14 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 76.68 |
| Shannon entropy | 0.54961 |
| G+C content | 0.45807 |
| Mean single sequence MFE | -23.08 |
| Consensus MFE | -12.39 |
| Energy contribution | -13.82 |
| Covariance contribution | 1.43 |
| Combinations/Pair | 1.16 |
| Mean z-score | -0.94 |
| Structure conservation index | 0.54 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.591502 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
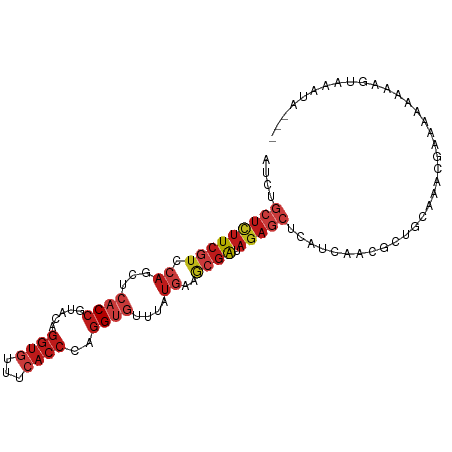
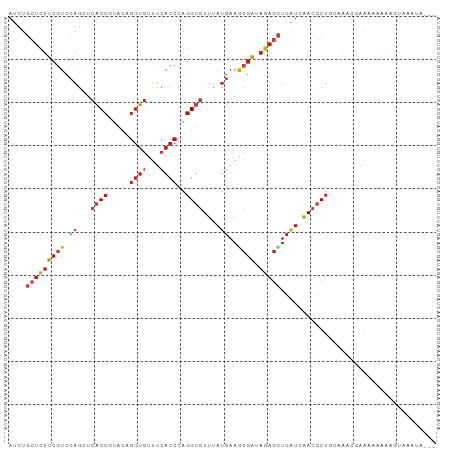
>dm3.chr3R 1118491 96 + 27905053 AUCUGCUCUUCGUCCAGCUCACCGUACAGGUGUUUCACCCAGGUGUUUAUGAAGCGAUAGAGCUCAUCAACGCUGCAAUUGAAAACAAAAUCAAUA--- ....(((((((((.((...((((.....((((...))))..))))....))..)))).)))))..............(((((........))))).--- ( -21.60, z-score = -1.13, R) >droSim1.chr3R 1142791 96 + 27517382 AUCUGCUCUUCGUCCAGCUCACCGUACAGGUGUUUCACCCAGGUGUUUAUGAAGCGAUAGAGCUCAUCAACGCUGCAAUCGAAAAAAAAUGCAAUA--- ...(((..((((....((.((((.....)))).........((((((.((((.((......)))))).))))))))...)))).......)))...--- ( -21.60, z-score = -0.82, R) >droSec1.super_6 1239798 96 + 4358794 AUCUGCUCUUCGUCCAGCUCACCGUACAGGUGUUUCACCCAGGUGUUUAUGAAGCGAUAGAGCUCAUCAACGCUGCAAUCGAAACAAAAUGUAAUA--- ....(((........)))......((((..((((((.....((((((.((((.((......)))))).))))))......))))))...))))...--- ( -23.10, z-score = -1.37, R) >droYak2.chr3R 1498198 93 + 28832112 AUCUGCUCUUCGUCCAGCUCACCGUACAAGUGUUUAACCCAGGUGUUUAUGAAGCGAUAGAGCUCAUCAACGCUGC--AAUUGAAAAAUAUAAUA---- ....(((((((((.((...((((......((.....))...))))....))..)))).)))))...((((......--..))))...........---- ( -16.30, z-score = 0.01, R) >droEre2.scaffold_4770 1393280 94 + 17746568 AUCUGCUCUUCGUCCAGCUCACCGUACAGGUGUUUCACCCAGGUGUUUAUGAAGCGAUAGAGCUCAUCAACGCUGCAAUUGCA--UAAAUGUAAUA--- ....(((((((((.((...((((.....((((...))))..))))....))..)))).)))))..............((((((--....)))))).--- ( -22.70, z-score = -0.99, R) >droAna3.scaffold_13340 23300954 96 + 23697760 AUCUGCUCUUCGUCCAGUUCACCGUACAGGUGUUUCACCCAGGUGUUUAUGAAGCGAUAGAGCUCAUCAACGCUGUAAUGGAAGAGAUUGUUAGUU--- .....((((((........((((.....))))...((....((((((.((((.((......)))))).))))))....))))))))..........--- ( -25.60, z-score = -1.23, R) >dp4.chr2 9126988 96 - 30794189 AUUUGCUCUUCGUCCAGCUCACCGUACAGGUGUUUCACCCAGGUGUUUAUGAAGCGAUAGAGCUCAUCAACGCUAAAAAC-AGAGAAGGGAGCGAUA-- ..((((((((..(((....((((.....)))).........((((((.((((.((......)))))).))))))......-.).)).))))))))..-- ( -26.50, z-score = -1.36, R) >droPer1.super_3 2923888 96 - 7375914 AUUUGCUCUUCGUCCAGCUCACCGUACAGGUGUUUCACCCAGGUGUUUAUGAAGCGAUAGAGCUCAUCAACGCUAAAAAC-AGAGAAGGGAGCGAUA-- ..((((((((..(((....((((.....)))).........((((((.((((.((......)))))).))))))......-.).)).))))))))..-- ( -26.50, z-score = -1.36, R) >droWil1.scaffold_181130 3850385 96 - 16660200 AUUUGCUCUUCAUCCAGCUCACCGUACAGGUGUUUCACCCAGGUGUUUAUAAAGCGAUAGAGCUCAUCAACGCUAAAUACGAGACAAAAGAGAGAG--- .....(((((.......(((........((((...))))..((((((.....(((......)))....))))))......)))......)))))..--- ( -18.72, z-score = -0.32, R) >droVir3.scaffold_12855 2367037 99 - 10161210 AUUUGCUCUUCGUCCAAUUCACCGUACAGGUGUUUCACCCAGGUGUUUAUGAAACGAUAGAGCUCGUCAACGCUACAGGGCAAAGGCAUGCAGUUAGAU ...(((.(((.((((....((((.....)))).........((((((.((((...........)))).))))))...)))).))))))........... ( -23.70, z-score = -0.41, R) >droMoj3.scaffold_6540 11133950 96 - 34148556 AUCUGCUCUUCGUCCAGCUCACCGUACAGGUGUUUCACCCAGGUGUUUAUGAAACGAUAGAGCUCAUCAACGCUGCAAAUGAGAGGGGGGGUAGUC--- ..(((((((((.((((((.((((.....)))).........((((((.((((...........)))).))))))))...)).)).)))))))))..--- ( -27.60, z-score = -0.47, R) >droGri2.scaffold_15074 3394781 95 - 7742996 AUUUGCUCUUCGUCCAGCUCACCGUACAGGUGUUUCACCCAGGUGUUUAUGAAACGAUAGAGCUCAUCAACGCUGCGCAGAUAUGACAAAUAAAU---- ((((((.(..(((..((((((((.....)))((((((............))))))....))))).....)))..).)))))).............---- ( -21.40, z-score = -0.70, R) >anoGam1.chr2R 53349223 93 - 62725911 AUGGCAGCUUCAUCCAGCUCACCAUACAGGUGUUGCACCCAGGCAUUCAAGAAGCGAUACAAUUCAUCAACCCUAAAGGAAUAAAGCGUAACG------ ...((.(((((........((((.....)))).(((......))).....)))))................((....))......))......------ ( -17.20, z-score = -0.42, R) >apiMel3.Group5 6096518 92 + 13386189 CAAUAAUUCUCGUCGAGCUCUCCAUAAAGCGAGGGCACCCACGCAUGGAUGAACCUGUAGAGGUCGUCCACCCUGCAGGAACAACAGGAAUA------- .....((((..((.(...(((((((...(((.((....)).)))))))).)).(((((((.(((.....))))))))))..).))..)))).------- ( -30.60, z-score = -2.61, R) >consensus AUCUGCUCUUCGUCCAGCUCACCGUACAGGUGUUUCACCCAGGUGUUUAUGAAGCGAUAGAGCUCAUCAACGCUGCAAACGAAAAAAAAGUAAAUA___ ....(((((((((.((...((((.....((((...))))..))))....))..)))).))))).................................... (-12.39 = -13.82 + 1.43)

| Location | 1,118,491 – 1,118,587 |
|---|---|
| Length | 96 |
| Sequences | 14 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 76.68 |
| Shannon entropy | 0.54961 |
| G+C content | 0.45807 |
| Mean single sequence MFE | -24.66 |
| Consensus MFE | -16.09 |
| Energy contribution | -16.67 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.33 |
| Mean z-score | -0.87 |
| Structure conservation index | 0.65 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.831332 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
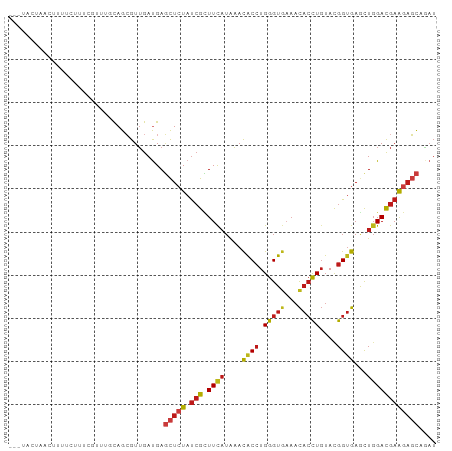
>dm3.chr3R 1118491 96 - 27905053 ---UAUUGAUUUUGUUUUCAAUUGCAGCGUUGAUGAGCUCUAUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAU ---(((..((.((((........)))).))..))).(((((.(((.((((....((((((((((...))))))...))))...)))))))))))).... ( -26.70, z-score = -1.45, R) >droSim1.chr3R 1142791 96 - 27517382 ---UAUUGCAUUUUUUUUCGAUUGCAGCGUUGAUGAGCUCUAUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAU ---..((((.....((((((..(.((((....((((((......)).))))...((((((((((...))))))...)))).)))).))))))))))).. ( -25.50, z-score = -0.95, R) >droSec1.super_6 1239798 96 - 4358794 ---UAUUACAUUUUGUUUCGAUUGCAGCGUUGAUGAGCUCUAUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAU ---........(((((((((..(.((((....((((((......)).))))...((((((((((...))))))...)))).)))).)))))..))))). ( -24.40, z-score = -0.70, R) >droYak2.chr3R 1498198 93 - 28832112 ----UAUUAUAUUUUUCAAUU--GCAGCGUUGAUGAGCUCUAUCGCUUCAUAAACACCUGGGUUAAACACUUGUACGGUGAGCUGGACGAAGAGCAGAU ----...........(((((.--.....)))))...(((((.(((.((((....(((((((((.....)))))...))))...)))))))))))).... ( -18.40, z-score = 0.30, R) >droEre2.scaffold_4770 1393280 94 - 17746568 ---UAUUACAUUUA--UGCAAUUGCAGCGUUGAUGAGCUCUAUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAU ---.....((((.(--(((.......)))).)))).(((((.(((.((((....((((((((((...))))))...))))...)))))))))))).... ( -27.50, z-score = -2.08, R) >droAna3.scaffold_13340 23300954 96 - 23697760 ---AACUAACAAUCUCUUCCAUUACAGCGUUGAUGAGCUCUAUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAACUGGACGAAGAGCAGAU ---........((((....(((((......))))).(((((.(((.((((....((((((((((...))))))...))))...)))))))))))))))) ( -23.70, z-score = -1.49, R) >dp4.chr2 9126988 96 + 30794189 --UAUCGCUCCCUUCUCU-GUUUUUAGCGUUGAUGAGCUCUAUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAAAU --............((((-...((((((....((((((......)).))))...((((((((((...))))))...)))).))))))...))))..... ( -23.50, z-score = -0.46, R) >droPer1.super_3 2923888 96 + 7375914 --UAUCGCUCCCUUCUCU-GUUUUUAGCGUUGAUGAGCUCUAUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAAAU --............((((-...((((((....((((((......)).))))...((((((((((...))))))...)))).))))))...))))..... ( -23.50, z-score = -0.46, R) >droWil1.scaffold_181130 3850385 96 + 16660200 ---CUCUCUCUUUUGUCUCGUAUUUAGCGUUGAUGAGCUCUAUCGCUUUAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGAUGAAGAGCAAAU ---........((((.(((.((((((((......((((......))))......((((((((((...))))))...)))).))))))))..))))))). ( -25.30, z-score = -1.33, R) >droVir3.scaffold_12855 2367037 99 + 10161210 AUCUAACUGCAUGCCUUUGCCCUGUAGCGUUGACGAGCUCUAUCGUUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAAUUGGACGAAGAGCAAAU ......(((((.((....))..))))).........(((((.(((((.((....((((((((((...))))))...))))...)))))))))))).... ( -26.20, z-score = -1.39, R) >droMoj3.scaffold_6540 11133950 96 + 34148556 ---GACUACCCCCCCUCUCAUUUGCAGCGUUGAUGAGCUCUAUCGUUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAU ---..............(((((.((...)).)))))(((((.(((((.((....((((((((((...))))))...))))...)))))))))))).... ( -23.70, z-score = -0.37, R) >droGri2.scaffold_15074 3394781 95 + 7742996 ----AUUUAUUUGUCAUAUCUGCGCAGCGUUGAUGAGCUCUAUCGUUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAAAU ----.....((((((.((((.(((...))).))))(((((....(((((((...(....).)))))))(((.....)))))))).))))))........ ( -25.00, z-score = -0.83, R) >anoGam1.chr2R 53349223 93 + 62725911 ------CGUUACGCUUUAUUCCUUUAGGGUUGAUGAAUUGUAUCGCUUCUUGAAUGCCUGGGUGCAACACCUGUAUGGUGAGCUGGAUGAAGCUGCCAU ------......(((((((((.(((((((.(((((.....)))))..))))))).(((.(((((...)))))....))).....)))))))))...... ( -22.60, z-score = 0.07, R) >apiMel3.Group5 6096518 92 - 13386189 -------UAUUCCUGUUGUUCCUGCAGGGUGGACGACCUCUACAGGUUCAUCCAUGCGUGGGUGCCCUCGCUUUAUGGAGAGCUCGACGAGAAUUAUUG -------.((((.(((((...(((.(((((.....))).)).)))((((.((((((((.((....)).)))...))))))))).))))).))))..... ( -29.30, z-score = -1.00, R) >consensus ___UACUAACUUUUCUUUCGUUUGCAGCGUUGAUGAGCUCUAUCGCUUCAUAAACACCUGGGUGAAACACCUGUACGGUGAGCUGGACGAAGAGCAGAU ....................................(((((.(((.((((....((((.(((((...)))))....))))...)))))))))))).... (-16.09 = -16.67 + 0.59)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:51:09 2011