
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 852,522 – 852,652 |
| Length | 130 |
| Max. P | 0.998388 |

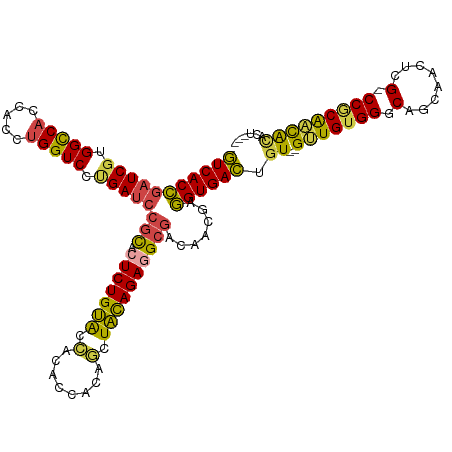
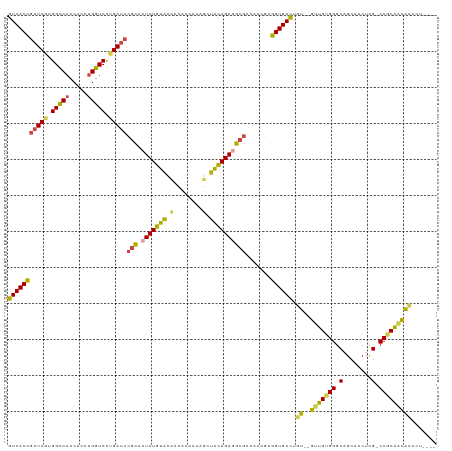
| Location | 852,522 – 852,634 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 78.77 |
| Shannon entropy | 0.40160 |
| G+C content | 0.60085 |
| Mean single sequence MFE | -47.92 |
| Consensus MFE | -36.16 |
| Energy contribution | -35.67 |
| Covariance contribution | -0.49 |
| Combinations/Pair | 1.37 |
| Mean z-score | -3.00 |
| Structure conservation index | 0.75 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.34 |
| SVM RNA-class probability | 0.998388 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 852522 112 - 27905053 GUCACCGAUCGUGGCCACCACCUGGUCCUGAUCCACACUCUGUACCACACCACAGCUACAGAGGCGACAACGAGGUGACUGU--GUUGUGGGCAGCAACUCG-CCGCAACACACU---- (((((((((((.(((((.....))))).)))))....(((((((.(........).)))))))..........))))))(((--(((((((.(........)-))))))))))..---- ( -45.20, z-score = -2.97, R) >droYak2.chrU 2863715 108 - 28119190 GUCACCGUUCGUGGCCACCACCUGGUCCCGAUCCGCACUCUGUGUGGUA-CAGAGCUACAGAAGCGACAACGAGGUGACUGU--GCUGGGGGCAGCAACGUG-CCGCGGUAU------- ((((((..(((.(((((.....))))).)))..(((..(((((((....-....)).))))).))).......)))))).((--((((((.((......)).-)).))))))------- ( -38.80, z-score = 0.73, R) >droEre2.scaffold_4845 1960403 113 - 22589142 GUCACCUAUCCUGGUCACCACCAGGUCCCGAUCCGCGAUCUGUACCACACCACAGCUACAGAGGCGACAUCGAGGUGACUGU--GCUGUGGGCAGCAACGUG-CCGCAACACCCGU--- (((((((..((((((....))))))...((((.(((..((((((.(........).)))))).)))..))))))))))).((--(.(((((.((......))-))))).)))....--- ( -42.90, z-score = -1.81, R) >droSec1.super_6 4341027 113 + 4358794 GUCACCGAUCGUGGCCACCACCUGGUCCUGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCGACAACGAGGUGACUGU--GUUGUGGACAGCAACUCGCCCGCAACACACU---- (((((((((((.(((((.....))))).)))))(((.(((((((.(........).)))))))))).......))))))(((--(((((((...((.....))))))))))))..---- ( -50.70, z-score = -4.86, R) >droSim1.chr2L 950824 112 + 22036055 GUCACCGAUCGGGGCCACCACCUGGUCCUGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCGUCAACGAGGUGACUGU--GUUGUGGGCAGCAACUCG-CCGCAACACACU---- (((((((((((((((((.....)))))))))))(((.(((((((.(........).)))))))))).......))))))(((--(((((((.(........)-))))))))))..---- ( -56.10, z-score = -5.37, R) >droAna3.scaffold_13021 90687 118 + 236608 AUCACUGAUCGUGGCCACCACCUGGUCUUGAUCGGUGCUCUGCAGUGCUACACGACUGUAGAGGCACCUGCUGAGUGAUCGUCCGUUGUGGGCAGCCACUUG-CCACAACGGUUCCCCU (((((((((((.(((((.....))))).)))))(((((((((((((........)))))))).))))).....))))))...(((((((((.(((....)))-)))))))))....... ( -53.80, z-score = -3.73, R) >consensus GUCACCGAUCGUGGCCACCACCUGGUCCUGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCGACAACGAGGUGACUGU__GUUGUGGGCAGCAACUCG_CCGCAACACACU____ (((((((((((.(((((.....))))).)))))(((.(((((((.(........).)))))))))).......)))))).....(((((((.(........).)))))))......... (-36.16 = -35.67 + -0.49)

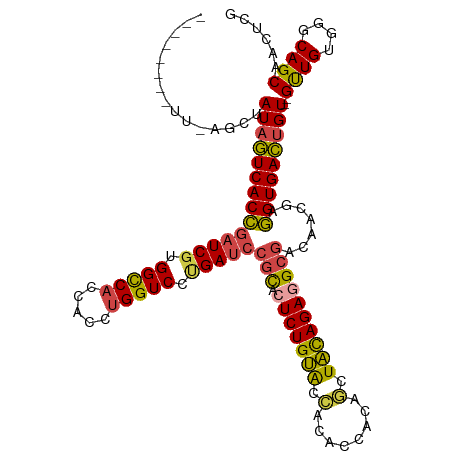
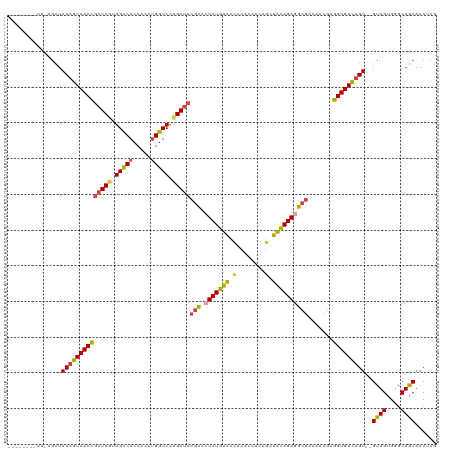
| Location | 852,534 – 852,652 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.28 |
| Shannon entropy | 0.41561 |
| G+C content | 0.57055 |
| Mean single sequence MFE | -44.08 |
| Consensus MFE | -34.74 |
| Energy contribution | -34.47 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.79 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.51 |
| SVM RNA-class probability | 0.991893 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 852534 118 - 27905053 UUGGUCACUUCAGCUAUAGUCACCGAUCGUGGCCACCACCUGGUCCUGAUCCACACUCUGUACCACACCACAGCUACAGAGGCGACAACGAGGUGACUGU--GUUGUGGGCAGCAACUCG ...(((....((((.((((((((((((((.(((((.....))))).)))))....(((((((.(........).)))))))..........)))))))))--))))..)))......... ( -42.10, z-score = -1.54, R) >droYak2.chrU 2863724 109 - 28119190 --------UUGAGCUAUAGUCACCGUUCGUGGCCACCACCUGGUCCCGAUCCGCACUCUGUGUGGUA-CAGAGCUACAGAAGCGACAACGAGGUGACUGU--GCUGGGGGCAGCAACGUG --------.......(((((((((..(((.(((((.....))))).)))..(((..(((((((....-....)).))))).))).......)))))))))--((((....))))...... ( -37.10, z-score = 0.30, R) >droEre2.scaffold_4845 1960416 106 - 22589142 ------------GCUAUAGUCACCUAUCCUGGUCACCACCAGGUCCCGAUCCGCGAUCUGUACCACACCACAGCUACAGAGGCGACAUCGAGGUGACUGU--GCUGUGGGCAGCAACGUG ------------...((((((((((..((((((....))))))...((((.(((..((((((.(........).)))))).)))..))))))))))))))--((((....))))...... ( -42.70, z-score = -2.73, R) >droSec1.super_6 4341040 118 + 4358794 UUGGUCACUUCAGCUAUAGUCACCGAUCGUGGCCACCACCUGGUCCUGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCGACAACGAGGUGACUGU--GUUGUGGACAGCAACUCG ...(((....((((.((((((((((((((.(((((.....))))).)))))(((.(((((((.(........).)))))))))).......)))))))))--))))..)))......... ( -47.60, z-score = -3.27, R) >droSim1.chr2L 950836 118 + 22036055 UUGGUCACUUUAGCUAUAGUCACCGAUCGGGGCCACCACCUGGUCCUGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCGUCAACGAGGUGACUGU--GUUGUGGGCAGCAACUCG (((.((((...(((.((((((((((((((((((((.....)))))))))))(((.(((((((.(........).)))))))))).......)))))))))--))))))).)))....... ( -52.20, z-score = -4.01, R) >droAna3.scaffold_13021 90703 108 + 236608 ------------AUCAUCAUCACUGAUCGUGGCCACCACCUGGUCUUGAUCGGUGCUCUGCAGUGCUACACGACUGUAGAGGCACCUGCUGAGUGAUCGUCCGUUGUGGGCAGCCACUUG ------------......(((((((((((.(((((.....))))).)))))(((((((((((((........)))))))).))))).....)))))).((((.....))))......... ( -42.80, z-score = -1.81, R) >consensus ________UU_AGCUAUAGUCACCGAUCGUGGCCACCACCUGGUCCUGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCGACAACGAGGUGACUGU__GUUGUGGGCAGCAACUCG ...............((((((((((((((.(((((.....))))).)))))(((.(((((((.(........).)))))))))).......)))))))))..((((....))))...... (-34.74 = -34.47 + -0.27)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:50:33 2011