
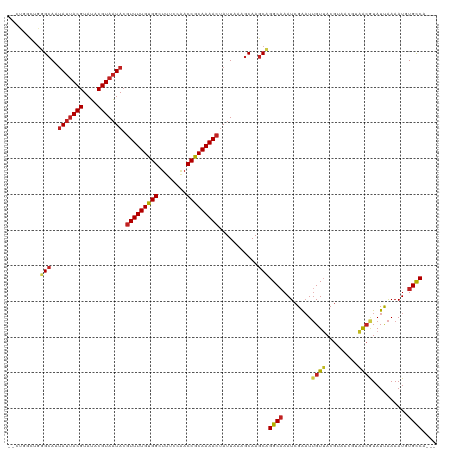
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 6,302,292 – 6,302,428 |
| Length | 136 |
| Max. P | 0.998293 |

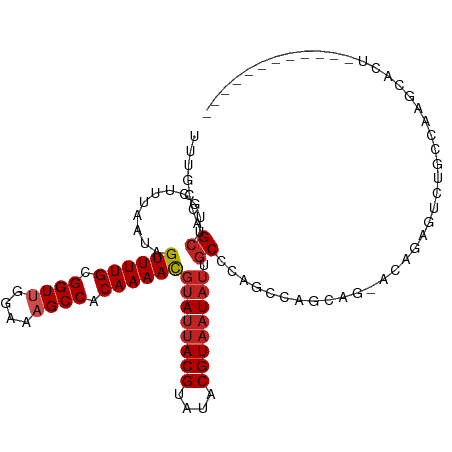
| Location | 6,302,292 – 6,302,407 |
|---|---|
| Length | 115 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.88 |
| Shannon entropy | 0.30814 |
| G+C content | 0.38302 |
| Mean single sequence MFE | -31.88 |
| Consensus MFE | -21.31 |
| Energy contribution | -23.11 |
| Covariance contribution | 1.80 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.83 |
| Structure conservation index | 0.67 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.27 |
| SVM RNA-class probability | 0.987159 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 6302292 115 + 23011544 ---UGGCACAUUUUAAUACGUUACGUUACGUAACAAUUCGAUUUGCCAGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGCCAG-- ---(((((.(((.......((((((...)))))).....))).)))))(((.((.......((((((.((((....)))).))))))((((((((....)))))))).))..)))...-- ( -34.10, z-score = -3.34, R) >droSim1.chr2L 6107179 115 + 22036055 ---UGGCACAUUUUAAUACGUUACGUUACGUAACAAUUCGAUUUGCCAGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGCCAG-- ---(((((.(((.......((((((...)))))).....))).)))))(((.((.......((((((.((((....)))).))))))((((((((....)))))))).))..)))...-- ( -34.10, z-score = -3.34, R) >droSec1.super_5 4373267 115 + 5866729 ---UGGCACAUUUUAAUACGUUACGUUACGUAACAAUUCGAUUUGCCAGCUUGCUUUAAUAGUUUUGUGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGCCAG-- ---(((((.(((.......((((((...)))))).....))).)))))(((.((.......(((((((((((....)))))))))))((((((((....)))))))).))..)))...-- ( -38.00, z-score = -4.65, R) >droYak2.chr2L 15721421 115 - 22324452 ---UGGCACAUUUUAAUACGUUACGUUACGUAACAAUUCGAUUUGCCAGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGCCAG-- ---(((((.(((.......((((((...)))))).....))).)))))(((.((.......((((((.((((....)))).))))))((((((((....)))))))).))..)))...-- ( -34.10, z-score = -3.34, R) >droEre2.scaffold_4929 15229861 115 + 26641161 ---UGGCACAUUUUAAUACGUUACGUUACGUAACAAUUCGAUUUGCCAGCUUGCUUUAAUAGUUUUGCGGUCGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGCCAG-- ---(((((.(((.......((((((...)))))).....))).)))))(((.((.......((((((.(((......))).))))))((((((((....)))))))).))..)))...-- ( -31.00, z-score = -2.40, R) >droAna3.scaffold_12916 7930414 111 + 16180835 ---UGACACAUUUUAAUACGCUGUUUCACGUAACAAACCCAUU-GCCUGCUUGUUUUAAUUGUUUUGCGGUAAGAAAGCCACAAAAUCUAAUACGGUUACGUAAUAUUCCCCUGG----- ---.((.(((...........))).))(((((((.........-(....).(((.(((...((((((.(((......))).)))))).))).)))))))))).............----- ( -15.40, z-score = 1.07, R) >dp4.chr4_group5 250195 116 - 2436548 ---UGGCACAUUUUAAUAUGUUACUCAACGUAACAAUUCGAUUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCACCAGCAGA- ---.((((.(((..(((.((((((.....))))))))).))).))))(((((((.......((((((.((((....)))).))))))((((((((....)))))))).)))..))))..- ( -36.70, z-score = -4.23, R) >droPer1.super_5 248621 116 - 6813705 ---UGGCACAUUUUAAUAUGUUACUCAACGUAACAAUUCGAUUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCACCAGCAGA- ---.((((.(((..(((.((((((.....))))))))).))).))))(((((((.......((((((.((((....)))).))))))((((((((....)))))))).)))..))))..- ( -36.70, z-score = -4.23, R) >droWil1.scaffold_180708 5277098 109 - 12563649 UGUGGACA-AUUUUGAUAACUAAA----CGUUACAAUUCGAUUUGCCGGCUUGCUUUAAUAGUUUUGCGGUUAGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCAGAG------ ..(((.((-(..((((((((....----.))))....)))).))))))..((((.......((((((.((((....)))).))))))((((((((....)))))))).))))..------ ( -26.30, z-score = -1.73, R) >droVir3.scaffold_12963 2868302 111 + 20206255 ---UGGCA-AUUUUGAUACGUUU-----CGUAACAAUUCGAUUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUAGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCACUGCAGUGG ---.((((-(..((((...(((.-----...)))...)))).)))))(((.(((.......((((((.((((....)))).))))))((((((((....)))))))).)))..))).... ( -32.90, z-score = -2.83, R) >droMoj3.scaffold_6500 13135897 111 + 32352404 ---UGGCA-AUUUUGAUACGUUU-----CGUAACAAUUCGAUUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUAGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCACUGCAGUGG ---.((((-(..((((...(((.-----...)))...)))).)))))(((.(((.......((((((.((((....)))).))))))((((((((....)))))))).)))..))).... ( -32.90, z-score = -2.83, R) >droGri2.scaffold_15252 2485932 111 + 17193109 ---UGGCA-AUUUCGAUACGUUC-----CGUUACCAUUCGAUUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUAGAAAGCCACAAAACGUAUUACGUAUACGUUAUUUUGCACUGCAGUGG ---.((((-(..((((...((..-----....))...)))).)))))(((.(((...((((((((((.((((....)))).))))))((((.....))))..))))..)))..))).... ( -30.30, z-score = -2.13, R) >consensus ___UGGCACAUUUUAAUACGUUACGUUACGUAACAAUUCGAUUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCACAGCCAG__ ....((((.(((....((((........)))).......))).)))).((...........((((((.((((....)))).))))))((((((((....)))))))).)).......... (-21.31 = -23.11 + 1.80)

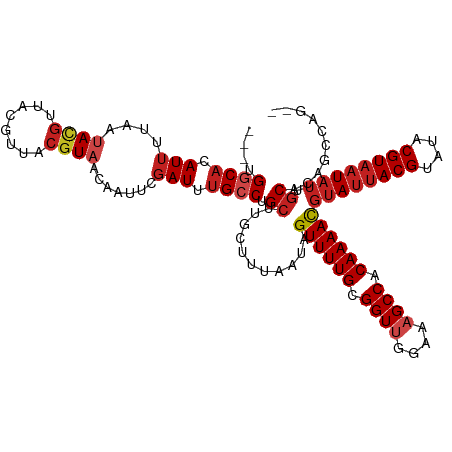
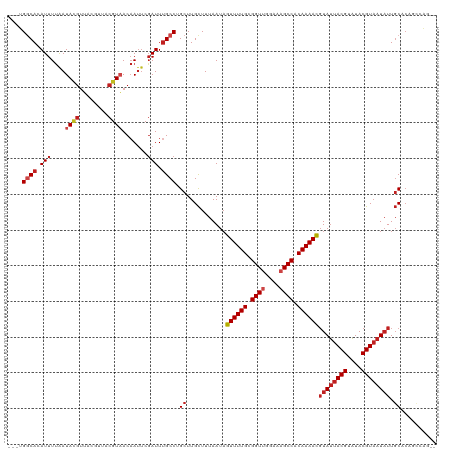
| Location | 6,302,292 – 6,302,407 |
|---|---|
| Length | 115 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.88 |
| Shannon entropy | 0.30814 |
| G+C content | 0.38302 |
| Mean single sequence MFE | -33.08 |
| Consensus MFE | -22.44 |
| Energy contribution | -22.55 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.22 |
| Mean z-score | -3.05 |
| Structure conservation index | 0.68 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.63 |
| SVM RNA-class probability | 0.993631 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 6302292 115 - 23011544 --CUGGCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCUGGCAAAUCGAAUUGUUACGUAACGUAACGUAUUAAAAUGUGCCA--- --..((((..((..(((((((....))))))).(((((((((........))))))))).......)).))))((((.....((((((((((...))))))).)))......)))).--- ( -36.90, z-score = -3.53, R) >droSim1.chr2L 6107179 115 - 22036055 --CUGGCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCUGGCAAAUCGAAUUGUUACGUAACGUAACGUAUUAAAAUGUGCCA--- --..((((..((..(((((((....))))))).(((((((((........))))))))).......)).))))((((.....((((((((((...))))))).)))......)))).--- ( -36.90, z-score = -3.53, R) >droSec1.super_5 4373267 115 - 5866729 --CUGGCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCACAAAACUAUUAAAGCAAGCUGGCAAAUCGAAUUGUUACGUAACGUAACGUAUUAAAAUGUGCCA--- --..((((..((..(((((((....))))))).(((((((((........))))))))).......)).))))((((.....((((((((((...))))))).)))......)))).--- ( -37.30, z-score = -4.07, R) >droYak2.chr2L 15721421 115 + 22324452 --CUGGCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCUGGCAAAUCGAAUUGUUACGUAACGUAACGUAUUAAAAUGUGCCA--- --..((((..((..(((((((....))))))).(((((((((........))))))))).......)).))))((((.....((((((((((...))))))).)))......)))).--- ( -36.90, z-score = -3.53, R) >droEre2.scaffold_4929 15229861 115 - 26641161 --CUGGCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCGACCGCAAAACUAUUAAAGCAAGCUGGCAAAUCGAAUUGUUACGUAACGUAACGUAUUAAAAUGUGCCA--- --..((((..((..(((((((....))))))).(((((((((........))))))))).......)).))))((((.....((((((((((...))))))).)))......)))).--- ( -36.90, z-score = -3.23, R) >droAna3.scaffold_12916 7930414 111 - 16180835 -----CCAGGGGAAUAUUACGUAACCGUAUUAGAUUUUGUGGCUUUCUUACCGCAAAACAAUUAAAACAAGCAGGC-AAUGGGUUUGUUACGUGAAACAGCGUAUUAAAAUGUGUCA--- -----...........(((((((...((.((((.((((((((........))))))))...)))).))..((((((-.....)))))))))))))....((((......))))....--- ( -20.40, z-score = 0.86, R) >dp4.chr4_group5 250195 116 + 2436548 -UCUGCUGGUGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCAGGCAAAUCGAAUUGUUACGUUGAGUAACAUAUUAAAAUGUGCCA--- -..((((..(((..(((((((....))))))).(((((((((........))))))))).......)))))))((((.....(((((((((.....)))))).)))......)))).--- ( -36.80, z-score = -3.95, R) >droPer1.super_5 248621 116 + 6813705 -UCUGCUGGUGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCAGGCAAAUCGAAUUGUUACGUUGAGUAACAUAUUAAAAUGUGCCA--- -..((((..(((..(((((((....))))))).(((((((((........))))))))).......)))))))((((.....(((((((((.....)))))).)))......)))).--- ( -36.80, z-score = -3.95, R) >droWil1.scaffold_180708 5277098 109 + 12563649 ------CUCUGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCUAACCGCAAAACUAUUAAAGCAAGCCGGCAAAUCGAAUUGUAACG----UUUAGUUAUCAAAAU-UGUCCACA ------...(((..(((((((....))))))).(((((((((........))))))))).......)))....(((((........(((((.----....))))).....)-)))).... ( -25.02, z-score = -2.41, R) >droVir3.scaffold_12963 2868302 111 - 20206255 CCACUGCAGUGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCUAACCGCAAAACUAUUAAAGCAAGCAGGCAAAUCGAAUUGUUACG-----AAACGUAUCAAAAU-UGCCA--- ....(((..(((..(((((((....))))))).(((((((((........))))))))).......))).)))(((((......(((.((((-----...)))).)))..)-)))).--- ( -31.40, z-score = -3.45, R) >droMoj3.scaffold_6500 13135897 111 - 32352404 CCACUGCAGUGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCUAACCGCAAAACUAUUAAAGCAAGCAGGCAAAUCGAAUUGUUACG-----AAACGUAUCAAAAU-UGCCA--- ....(((..(((..(((((((....))))))).(((((((((........))))))))).......))).)))(((((......(((.((((-----...)))).)))..)-)))).--- ( -31.40, z-score = -3.45, R) >droGri2.scaffold_15252 2485932 111 - 17193109 CCACUGCAGUGCAAAAUAACGUAUACGUAAUACGUUUUGUGGCUUUCUAACCGCAAAACUAUUAAAGCAAGCAGGCAAAUCGAAUGGUAACG-----GAACGUAUCGAAAU-UGCCA--- ....(((..(((......(((....)))((((.(((((((((........)))))))))))))...))).)))(((((.(((((((......-----...))).))))..)-)))).--- ( -30.20, z-score = -2.31, R) >consensus __CUGGCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCAGGCAAAUCGAAUUGUUACGUAACGUAACGUAUUAAAAUGUGCCA___ .........(((..(((((((....))))))).(((((((((........)))))))))...........)))((((........((((.........))))..........)))).... (-22.44 = -22.55 + 0.11)
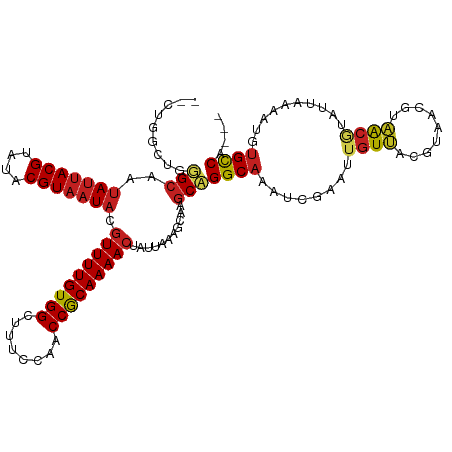
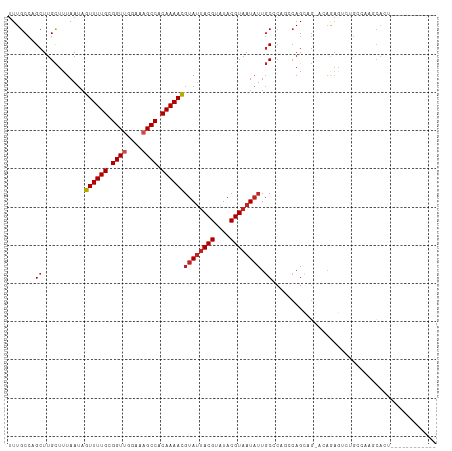
| Location | 6,302,330 – 6,302,428 |
|---|---|
| Length | 98 |
| Sequences | 11 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 77.79 |
| Shannon entropy | 0.46190 |
| G+C content | 0.43925 |
| Mean single sequence MFE | -32.43 |
| Consensus MFE | -16.89 |
| Energy contribution | -18.26 |
| Covariance contribution | 1.37 |
| Combinations/Pair | 1.05 |
| Mean z-score | -3.07 |
| Structure conservation index | 0.52 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.08 |
| SVM RNA-class probability | 0.997310 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 6302330 98 + 23011544 UUUGCCAGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGCCAGCAG-GCAAAGU-UGCCCAGCACU------------ ((((((.(((.(((......((((((.((((....)))).))))))((((((((....)))))))).....))).))).)-)))))((-(....)))...------------ ( -35.40, z-score = -3.83, R) >droSim1.chr2L 6107217 99 + 22036055 UUUGCCAGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGCCAGCAG-GCAAAGUCUGCCCAGCACU------------ ((((((.(((.(((......((((((.((((....)))).))))))((((((((....)))))))).....))).))).)-)))))..............------------ ( -34.80, z-score = -3.55, R) >droSec1.super_5 4373305 99 + 5866729 UUUGCCAGCUUGCUUUAAUAGUUUUGUGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGCCAGCAG-GCAAAGUCUGCCCAGCACU------------ ((((((.(((.(((......(((((((((((....)))))))))))((((((((....)))))))).....))).))).)-)))))..............------------ ( -38.70, z-score = -4.88, R) >droYak2.chr2L 15721459 98 - 22324452 UUUGCCAGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGCCAGCAG-GCAGAGU-UGCCCAGCACU------------ ((((((.(((.(((......((((((.((((....)))).))))))((((((((....)))))))).....))).))).)-)))))((-(....)))...------------ ( -35.50, z-score = -3.85, R) >droEre2.scaffold_4929 15229899 97 + 26641161 UUUGCCAGCUUGCUUUAAUAGUUUUGCGGUCGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGCCAGCAG-GCAGAGU-UGGCCACCAC------------- ...((((((((((.......((((((.(((......))).))))))((((((((....)))))))).))...(((....)-)).))))-))))......------------- ( -34.00, z-score = -3.57, R) >droAna3.scaffold_12916 7930452 98 + 16180835 -UUGCCUGCUUGUUUUAAUUGUUUUGCGGUAAGAAAGCCACAAAAUCUAAUACGGUUACGUAAUAUUCCCCUGGCAGGCUCUGUGCCAUCGCCAGGACG------------- -..(((((((.(........((((((.(((......))).))))))....((((....))))........).)))))))((((.((....))))))...------------- ( -27.70, z-score = -1.58, R) >dp4.chr4_group5 250233 112 - 2436548 UUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCACCAGCAGAUGCACAGAGGACGCCACACAAGAGACAGAGAGAG ..(((((((((((.......((((((.((((....)))).))))))((((((((....)))))))).)))..)))))..))).............................. ( -30.70, z-score = -2.66, R) >droPer1.super_5 248659 110 - 6813705 UUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCACCAGCAGAUGCACAGAGGACGCCACACAAGAGACAGAGAG-- ..(((((((((((.......((((((.((((....)))).))))))((((((((....)))))))).)))..)))))..)))............................-- ( -30.70, z-score = -2.63, R) >droWil1.scaffold_180708 5277134 94 - 12563649 UUUGCCGGCUUGCUUUAAUAGUUUUGCGGUUAGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCAG--AGAGACGAACUGCAGCUGCAAUG---------------- .((((.(((...........((((((.((((....)))).))))))((((((((....))))))))(((((--.........))))))))))))..---------------- ( -28.50, z-score = -2.52, R) >droVir3.scaffold_12963 2868334 98 + 20206255 UUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUAGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCACU-GCAGUGGCAGAAAAAGCAACA-GCAGC------------ .....(((((((((((....((((((.((((....)))).))))))((((((((....))))))))....((-((....))))..))))))..)-)))).------------ ( -33.90, z-score = -3.34, R) >droGri2.scaffold_15252 2485964 101 + 17193109 UUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUAGAAAGCCACAAAACGUAUUACGUAUACGUUAUUUUGCACU-GCAGUGGUAGAAAAACUAGAA-GCAAAAGC--------- (((((((((.(((...((((((((((.((((....)))).))))))((((.....))))..))))..)))..-))))((((......))))...-)))))...--------- ( -26.80, z-score = -1.39, R) >consensus UUUGCCAGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGCCAGCAG_ACAGAGUCUGCCAAGCACU____________ .......((...........((((((.((((....)))).))))))((((((((....)))))))).))........................................... (-16.89 = -18.26 + 1.37)

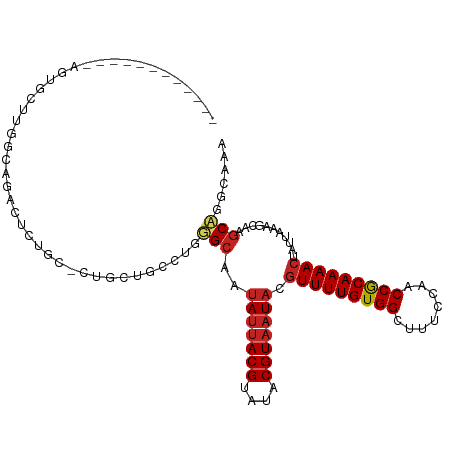
| Location | 6,302,330 – 6,302,428 |
|---|---|
| Length | 98 |
| Sequences | 11 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 77.79 |
| Shannon entropy | 0.46190 |
| G+C content | 0.43925 |
| Mean single sequence MFE | -33.64 |
| Consensus MFE | -18.06 |
| Energy contribution | -18.28 |
| Covariance contribution | 0.22 |
| Combinations/Pair | 1.11 |
| Mean z-score | -3.16 |
| Structure conservation index | 0.54 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.31 |
| SVM RNA-class probability | 0.998293 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 6302330 98 - 23011544 ------------AGUGCUGGGCA-ACUUUGC-CUGCUGGCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCUGGCAAA ------------......(((((-....)))-))((..(((..((..(((((((....))))))).(((((((((........))))))))).......)).)))..))... ( -37.30, z-score = -3.40, R) >droSim1.chr2L 6107217 99 - 22036055 ------------AGUGCUGGGCAGACUUUGC-CUGCUGGCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCUGGCAAA ------------......((((((...))))-))((..(((..((..(((((((....))))))).(((((((((........))))))))).......)).)))..))... ( -37.00, z-score = -3.03, R) >droSec1.super_5 4373305 99 - 5866729 ------------AGUGCUGGGCAGACUUUGC-CUGCUGGCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCACAAAACUAUUAAAGCAAGCUGGCAAA ------------......((((((...))))-))((..(((..((..(((((((....))))))).(((((((((........))))))))).......)).)))..))... ( -37.40, z-score = -3.53, R) >droYak2.chr2L 15721459 98 + 22324452 ------------AGUGCUGGGCA-ACUCUGC-CUGCUGGCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCUGGCAAA ------------......(((((-....)))-))((..(((..((..(((((((....))))))).(((((((((........))))))))).......)).)))..))... ( -37.50, z-score = -3.57, R) >droEre2.scaffold_4929 15229899 97 - 26641161 -------------GUGGUGGCCA-ACUCUGC-CUGCUGGCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCGACCGCAAAACUAUUAAAGCAAGCUGGCAAA -------------..((..(...-...)..)-)(((..(((..((..(((((((....))))))).(((((((((........))))))))).......)).)))..))).. ( -36.10, z-score = -3.20, R) >droAna3.scaffold_12916 7930452 98 - 16180835 -------------CGUCCUGGCGAUGGCACAGAGCCUGCCAGGGGAAUAUUACGUAACCGUAUUAGAUUUUGUGGCUUUCUUACCGCAAAACAAUUAAAACAAGCAGGCAA- -------------....(((((....)).))).((((((........((.((((....)))).))..((((((((........))))))))............))))))..- ( -26.90, z-score = -1.12, R) >dp4.chr4_group5 250233 112 + 2436548 CUCUCUCUGUCUCUUGUGUGGCGUCCUCUGUGCAUCUGCUGGUGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCAGGCAAA .......(((((((((((((((((......((((((....)))))).....)))).))).(((((.(((((((((........))))))))))))))..))))).))))).. ( -36.00, z-score = -4.26, R) >droPer1.super_5 248659 110 + 6813705 --CUCUCUGUCUCUUGUGUGGCGUCCUCUGUGCAUCUGCUGGUGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCAGGCAAA --.....(((((((((((((((((......((((((....)))))).....)))).))).(((((.(((((((((........))))))))))))))..))))).))))).. ( -36.00, z-score = -4.05, R) >droWil1.scaffold_180708 5277134 94 + 12563649 ----------------CAUUGCAGCUGCAGUUCGUCUCU--CUGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCUAACCGCAAAACUAUUAAAGCAAGCCGGCAAA ----------------..((((.(((((((.........--))))..(((((((....))))))).(((((((((........)))))))))..........)))..)))). ( -27.80, z-score = -3.27, R) >droVir3.scaffold_12963 2868334 98 - 20206255 ------------GCUGC-UGUUGCUUUUUCUGCCACUGC-AGUGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCUAACCGCAAAACUAUUAAAGCAAGCAGGCAAA ------------.((((-(..((((((..((((....))-)).....(((((((....))))))).(((((((((........)))))))))....)))))))))))..... ( -32.10, z-score = -3.07, R) >droGri2.scaffold_15252 2485964 101 - 17193109 ---------GCUUUUGC-UUCUAGUUUUUCUACCACUGC-AGUGCAAAAUAACGUAUACGUAAUACGUUUUGUGGCUUUCUAACCGCAAAACUAUUAAAGCAAGCAGGCAAA ---------...(((((-...(((.....)))...((((-..(((......(((....)))((((.(((((((((........)))))))))))))...))).))))))))) ( -25.90, z-score = -2.22, R) >consensus ____________AGUGCUUGGCAGACUCUGC_CUGCUGCCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCAGGCAAA ..........................................(((..(((((((....))))))).(((((((((........)))))))))...........)))...... (-18.06 = -18.28 + 0.22)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:21:01 2011