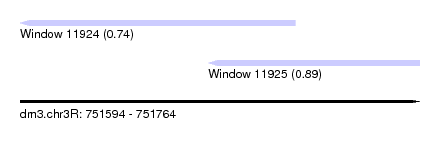
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 751,594 – 751,764 |
| Length | 170 |
| Max. P | 0.891689 |

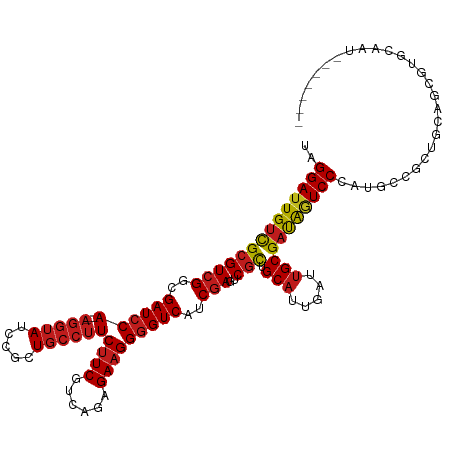
| Location | 751,594 – 751,711 |
|---|---|
| Length | 117 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.20 |
| Shannon entropy | 0.28146 |
| G+C content | 0.54906 |
| Mean single sequence MFE | -40.39 |
| Consensus MFE | -31.77 |
| Energy contribution | -32.07 |
| Covariance contribution | 0.30 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.79 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.740465 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 751594 117 - 27905053 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGCUGCAUUGAUUGCGAUAGUCCCAUGCCGCUGCAGCGUGCAAUAGAGAGG ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))..))).(((.....)))))))))))..((((((....))).)))......... ( -44.20, z-score = -1.42, R) >droSec1.super_6 847822 117 - 4358794 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGCUGCAUUGAUUGCGAUAGUCCCAUGCCGCUGCAGCGUGCAAUAGAGAGG ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))..))).(((.....)))))))))))..((((((....))).)))......... ( -44.20, z-score = -1.42, R) >droSim1.chr3R 792618 117 - 27517382 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGCUGCAUUGAUUGCGAUAGUCCCAUGCCGCUGCAGCGUGCAAUAGAGAGG ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))..))).(((.....)))))))))))..((((((....))).)))......... ( -44.20, z-score = -1.42, R) >droYak2.chr3R 1073062 117 - 28832112 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGCUGCAUUGAUUGCGAUAGUCCCAUGCCGCUGCAGCGUGCAAUUAAAAGG ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))..))).(((.....)))))))))))..((((((....))).)))......... ( -44.20, z-score = -2.04, R) >droEre2.scaffold_4770 1049863 116 - 17746568 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGCUGCAUUGAUUGCGAUAGUCCCAUGCCGCUGCAGCGUGCAAAAGAAGG- ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))..))).(((.....)))))))))))..((((((....))).)))........- ( -44.20, z-score = -1.81, R) >droAna3.scaffold_13340 22963268 106 - 23697760 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGCUGCAUUGAUUGCGAUAGUCCCAUGCCGCUGUAGCAGG----------- ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))..))).(((.....)))))))))))....((((....)).))----------- ( -38.30, z-score = -1.11, R) >dp4.chr2 8752238 104 + 30794189 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGCUGCAUUGAUUGCGAUAGUCCCAUGUCGCUGUAGAG------------- ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))..))).(((.....)))))))))))...............------------- ( -36.70, z-score = -1.28, R) >droPer1.super_3 2552528 104 + 7375914 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGCUGCAUUGAUUGCGAUAGUCCCAUGUCGCUGUAGAG------------- ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))..))).(((.....)))))))))))...............------------- ( -36.70, z-score = -1.28, R) >droWil1.scaffold_181130 8211494 104 + 16660200 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGUUGCAUUGAUUGCGAUAAUCCCAUGCCGCUAAAUUG------------- ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))((((......)))))))))))))))...............------------- ( -36.80, z-score = -2.11, R) >droMoj3.scaffold_6540 23724885 111 - 34148556 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGCUGCAUUGAUUGCGAUAAUCCCAUGCCGCUGCAGCGUGAAAUU------ ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))..))).(((.....)))))))))))(((((.......))))).....------ ( -41.50, z-score = -1.65, R) >droVir3.scaffold_12855 8525464 111 - 10161210 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGUUGCAUUGAUUGCGAUAAUCCCAUGCCGCUGCAGCGUGCAAUU------ ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))((((......)))))))))))))))..((((((....))).)))...------ ( -44.30, z-score = -2.62, R) >droGri2.scaffold_15074 5107383 112 - 7742996 UAGGAUUGUCGCGUCGGCGAUCCA---AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGCUGCAUUGAUUGCGAUAGUCCCAUGCCGCUGCAGUUUGCAAUAC----- ..((((((((((((((..((((((---(((((.....))))))((((......)))))))))..)))..))).(((.....)))))))))))........(((.....)))....----- ( -38.80, z-score = -1.08, R) >anoGam1.chr2R 60230997 113 + 62725911 UAGGACUGCUGGGUAGGCGAUCCACCUCCAUAUCCACUUGUCUCCUCGUCUGAGAAGGCGUCCUCAAUACGUUGCUCUGACUGCGACGUUCCCAUUCCGCUGCAAGUUGAAAA------- .(((((...((((((((.(......).)).))))))...((((.(((....))).)))))))))....(((((((.......))))))).....(((.((.....)).)))..------- ( -31.00, z-score = -0.56, R) >consensus UAGGAUUGUCGCGUCGGCGAUCCA___AGGUAUCCGCUGCCUUCUUCGUCAGAGAAGGGGUCAUCGAGUCGCUGCAUUGAUUGCGAUAGUCCCAUGCCGCUGCAGCGUGCAAU_______ ..((((((((((((((..(((((....(((((.....))))).((((......)))))))))..)))..))).(((.....)))))))))))............................ (-31.77 = -32.07 + 0.30)

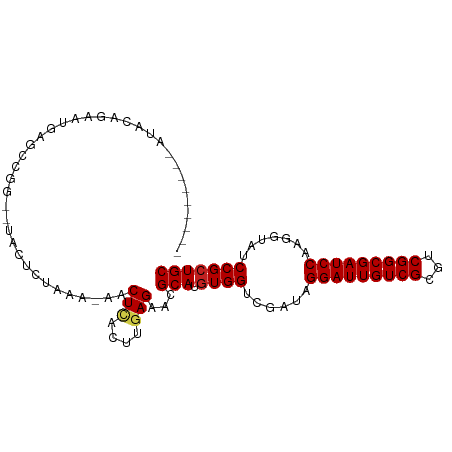
| Location | 751,674 – 751,764 |
|---|---|
| Length | 90 |
| Sequences | 12 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 75.03 |
| Shannon entropy | 0.53302 |
| G+C content | 0.49587 |
| Mean single sequence MFE | -26.38 |
| Consensus MFE | -19.00 |
| Energy contribution | -19.08 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.05 |
| Mean z-score | -0.91 |
| Structure conservation index | 0.72 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.10 |
| SVM RNA-class probability | 0.891689 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 751674 90 - 27905053 ---------AUACAGAAUGUGACG--AAGCCCACAAGGACUUACUUGAGAACGCAUGUGGCCGAUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC ---------.......(((((..(--.....).((((......))))....)))))(..((.(((((((((((((...)))))))))....)))).))..) ( -24.50, z-score = -0.04, R) >droSec1.super_6 847902 92 - 4358794 ---------AUACAGAAUGUGCCGGGGAACGCAAAAGGACUUACUUGAGAACGCAUUUGGUCGAUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC ---------...(((...(((((..(((.(((..(((......)))..((.(((...(((((.....)))))..))))).))).)))..)))))...))). ( -26.60, z-score = -0.34, R) >droSim1.chr3R 792698 92 - 27517382 ---------AUACAGAAUGUGCCGGGGAACGCAAAAGGACUUACUUGAGAACGCAUGUGGUCGAUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC ---------...(((...(((((..(((.(((..(((......)))..((.(((..(..(((.....)))..).))))).))).)))..)))))...))). ( -28.20, z-score = -0.62, R) >droYak2.chr3R 1073142 86 - 28832112 ---------AUGCAGAAUGGUCCG------CCUAAGGAACUUACUUGAGAACGCAUGUGGUCGAUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC ---------..((((...((....------))...(((((((..((((..((....))..))))..(((((((((...))))))))).)))).))).)))) ( -25.00, z-score = 0.00, R) >droEre2.scaffold_4770 1049942 86 - 17746568 ---------AUGCAGAAUGUGCCG------UCACAAGAACUUACUUGAGAACGCAUGUGGUCGAUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC ---------..((((...((((((------((.((((......)))).((.((....)).))))).(((((((((...)))))))))..)))))...)))) ( -28.20, z-score = -1.18, R) >droAna3.scaffold_13340 22963337 101 - 23697760 GUAAUUAAUGUGGCUCUCAUACUAAUUGAGAAUGGAUAACUCACUUGAGAACGCAUGUGGUCGGUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC .........(..(((((((.......)))))..(((((.(((((.((......)).))).......(((((((((...))))))))).)).)))))))..) ( -30.10, z-score = -1.31, R) >dp4.chr2 8752305 86 + 30794189 --------------GAACUAGAGGGUUUAUUGUAGA-GACUAACUUGAGAACGCAUGUGGUCGAUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC --------------....(((.((((...((.((..-(((((...((......))..)))))..))(((((((((...)))))))))))...)))).))). ( -22.60, z-score = -0.04, R) >droPer1.super_3 2552595 86 + 7375914 --------------GAACUAGAGGGUUUAUUGUAGA-GACUAACUUGAGAACGCAUGUGGUCGAUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC --------------....(((.((((...((.((..-(((((...((......))..)))))..))(((((((((...)))))))))))...)))).))). ( -22.60, z-score = -0.04, R) >droWil1.scaffold_181130 8211561 93 + 16660200 -----AUAGAUAAUUUUUAAAGCGA--UGUCCUUUC-AACUCACUUGAGAACGCAUGUGGUCUAUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC -----...............(((((--((.((((..-...((((.((......)).))))......(((((((((...)))))))))))))))).)))).. ( -24.40, z-score = -0.91, R) >droMoj3.scaffold_6540 23724959 91 - 34148556 ---------AUAUCUUACGCUUUAAAGAUAAAUUUA-CACUCACUUGAGAACGCAUGUGGUCUGUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC ---------.((((((........))))))......-..(((....)))...(((.((((.((...(((((((((...)))))))))..))...))))))) ( -26.00, z-score = -1.76, R) >droVir3.scaffold_12855 8525538 86 - 10161210 --------------AAAUAAUUUUGUUUUCUGUGAA-AACUCACUUGAGAACGCAUGUGGUCUGUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC --------------..........((((((.((((.-...))))..))))))(((.((((.((...(((((((((...)))))))))..))...))))))) ( -29.90, z-score = -3.52, R) >droGri2.scaffold_15074 5107458 95 - 7742996 ---AAUCAAAUGCCUUAUAAUAUGA--UUUGGAUGG-CACUCACUUGAGAACGCAUGUGGUCUGUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC ---.......((((...(((.....--.)))...))-))(((....)))...(((.((((.((...(((((((((...)))))))))..))...))))))) ( -28.50, z-score = -1.20, R) >consensus _________AUACAGAAUGAGCCGG__UACUCUAAA_AACUCACUUGAGAACGCAUGUGGUCGAUAGGAUUGUCGCGUCGGCGAUCCAAGGUAUCCGCUGC .......................................(((....)))...(((.((((......(((((((((...))))))))).......))))))) (-19.00 = -19.08 + 0.08)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:50:14 2011