
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 482,142 – 482,254 |
| Length | 112 |
| Max. P | 0.999818 |

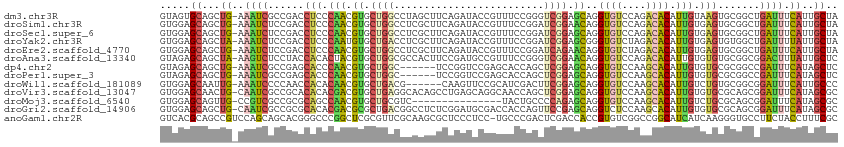
| Location | 482,142 – 482,254 |
|---|---|
| Length | 112 |
| Sequences | 13 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 69.80 |
| Shannon entropy | 0.69873 |
| G+C content | 0.58247 |
| Mean single sequence MFE | -48.00 |
| Consensus MFE | -8.22 |
| Energy contribution | -7.95 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.64 |
| Mean z-score | -3.62 |
| Structure conservation index | 0.17 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.36 |
| SVM RNA-class probability | 0.989304 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
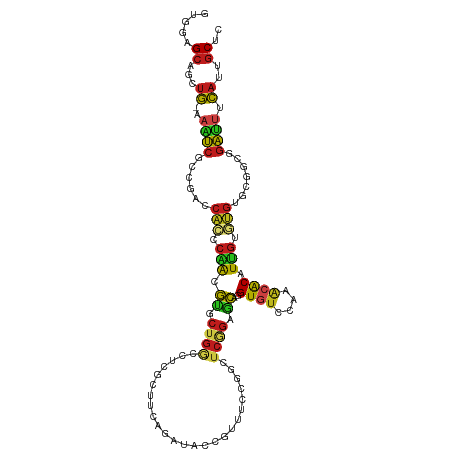
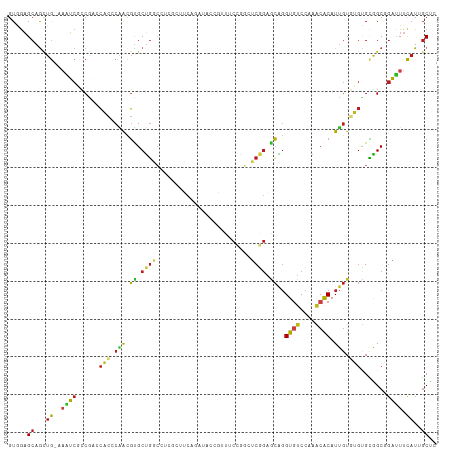
>dm3.chr3R 482142 112 + 27905053 GUAGUGCAGCUG-AAAUCGCCGACCUCCCAACGUGCUGGCCUAGCUUCAGAUACCGUUUCCGGGUCGGAGCAGGUGUCCAGACACAUUGUAAGUGCGGCUGAUUUCAUUGCUA .....((((.((-(((((((((..((..(((.((((((((((.(((((.(((.(((....))))))))))))))...)))).))).)))..))..)))).))))))))))).. ( -47.50, z-score = -3.85, R) >droSim1.chr3R 534768 112 + 27517382 GUGGAGCAGCUG-AAAUCUCCGACCUCCCAACGUGCUGGCCUCGCUUCAGAUACCGUUUCCGGAUCGGAACAGGUGUCCAGACACAUUGUGAGUGCGGCUGAUUUCAUUGCUA ....(((((.((-(((((.(((..(((.(((.((((((((...))....((((((..(((((...)))))..))))))))).))).))).)))..)))..)))))))))))). ( -43.30, z-score = -2.77, R) >droSec1.super_6 595950 112 + 4358794 GUGGAGCAGCUG-AAAUCUCCGACCUCCCAACGUGCUGGCCUCGCUUCAGAUACCGUUUCCGGAUCGGAGCAGGUGUCCAGACACAUUGUGAGUGCGGCUGAUUUCAUUGCUA ....(((((.((-(((((.(((..(((.(((.((((((((((.(((((.(((.(((....))))))))))))))...)))).))).))).)))..)))..)))))))))))). ( -48.40, z-score = -3.82, R) >droYak2.chr3R 784311 112 + 28832112 GUGGAGCAGCUA-AAAUCUCCGACCUCCCAAUGUGCUGACCUCGCUUCAGAUACCGUUUCCGGAUCGGAGCGGGUGUCUAGACACAUUGUGAGUGUGGCUGAUUUUAUUGCUA ....(((((.((-(((((.(((..(((.((((((((((((((((((((.(((.(((....)))))))))))))).))).)).))))))).)))..)))..)))))))))))). ( -51.10, z-score = -5.86, R) >droEre2.scaffold_4770 791125 112 + 17746568 GUGGAGCAGCUG-AAAUCUCCGACCUCCCAACGUGCUGGCCUCGCUUCAGAUACCGUUUCCGGAUCAGAACAGGUGUCUAGACACAUUGUGAGUGCGGCUGAUUUCAUUGCUA ....(((((.((-(((((.(((..(((.(((.((((((((((.(.(((.(((.(((....)))))).)))))))...)))).))).))).)))..)))..)))))))))))). ( -39.80, z-score = -2.17, R) >droAna3.scaffold_13340 22692659 112 + 23697760 GUAGAGCAGCUA-AAGUCUCCUACCACACUACGUGCUGGCGCCACUUCCGAUGCCGUUUCCGGGUCGGAACAGGUGUCCAGACACAUUGUGUGUGCGGCGGACUUUAUUGCUC ...((((((.((-((((((.(..((((((...(((((((((((..(((((((.(((....))))))))))..)))).)))).)))...))))).)..).)))))))))))))) ( -55.10, z-score = -5.69, R) >dp4.chr2 9357449 106 - 30794189 GUAGAGCAGCUG-AAAUCGCCGAGCACCCAACGUGCUGGC------UCCGGUCCGAGCACCAGCUCGGAGCAGGUGUCCAAGCACAUUGUGUGCGCGGCCGAUUUCAUAGCUC ...((((...((-(((((((((.((((.(((.((((((((------.(((.(((((((....))))))).).)).).)).))))).))).)))).))).))))))))..)))) ( -57.90, z-score = -6.27, R) >droPer1.super_3 3155462 106 - 7375914 GUAGAGCAGCUG-AAAUCGCCGAGCACCCAACGUGCUGGC------UCCGGUCCGAGCACCAGCUCGGAGCAGGUGUCCAAGCACAUUGUGUGCGCGGCCGAUUUCAUAGCUC ...((((...((-(((((((((.((((.(((.((((((((------.(((.(((((((....))))))).).)).).)).))))).))).)))).))).))))))))..)))) ( -57.90, z-score = -6.27, R) >droWil1.scaffold_181089 11415948 106 - 12369635 GUGGAGCAAUUG-AAAUCCCCAACCACACAACGUGCUGACG------CAAGUUCCGCAUCGACUUCGGAGCAGGUGUCCAAGCACAUUGUCUGUGCGGCGGAUUUCAUUGCCC ..((.(((((.(-((((((((...((.((((.(((((((((------(..((((((.(......)))))))..)))))..))))).)))).))...)).)))))))))))))) ( -45.10, z-score = -4.26, R) >droVir3.scaffold_13047 6409786 112 + 19223366 GUGGAGCAACUG-CAAUCGCCGCACACACGACGUGCUGAGGCACAGCCUGAGCAGGCAACCAGCUCGGAGCAGGUGUCCAAGCACAUUGUGUGCGCAGCGGAUUUCAUAGCGC (((((((....)-)..((((.((.(((((((.(((((..(((((.((((((((.(....)..)))))).))..)))))..))))).))))))).)).))))..)))))..... ( -52.50, z-score = -3.27, R) >droMoj3.scaffold_6540 32475028 97 - 34148556 GUGGAGCAGUUG-CCGUCGCCGCGCAGCCAACGUGCUGCGUC---------------UACUGCCCCAGAGCAGGUGUCCAAGCACAUUGUCUGCGCAGCGGAUUUCAUAGCGC (((((((....)-)..((((.((((((.(((.(((((.....---------------..((((......)))).......))))).))).)))))).))))..)))))..... ( -36.94, z-score = -0.32, R) >droGri2.scaffold_14906 11718816 112 + 14172833 GUGGAGCAGCUG-CAAUCGCCGCGCACACGACGCGCUGACGGCCUCUCGGAUGCGACCACCAGUUCCGAGCAGGUCUCCAAGCACAUUGUGUGCGCAGCGGAUUUCAUAGCGC (((((((....)-)..((((.((((((((((.(.(((...(((((((((((.((........)))))))).)))))....))).).)))))))))).))))..)))))..... ( -50.90, z-score = -2.51, R) >anoGam1.chr2R 50003634 112 - 62725911 GUCACGCAGCCGUCCAGCAGCACGGGCCCGGCUCGCGUUCGCAAGCGCUCCCUCC-UGCCCGACUCGACCACCGUGUCGGCCGGCAUCAUCAAGGGUGCCUUCUACCUUUCGC ((.((((((((((((........))))..)))).))))..))..(((........-.(.(((((.((.....)).))))).)((((((......))))))..........))) ( -37.60, z-score = 0.05, R) >consensus GUGGAGCAGCUG_AAAUCGCCGACCACCCAACGUGCUGGCCUCGCUUCAGAUACCGUUUCCGGCUCGGAGCAGGUGUCCAAACACAUUGUGUGUGCGGCGGAUUUCAUUGCUC ..................................((((.....................((.....)).((((((((....)))).)))).....)))).............. ( -8.22 = -7.95 + -0.28)
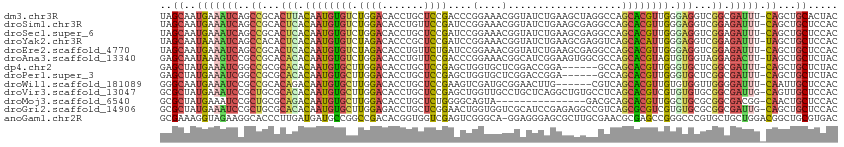
| Location | 482,142 – 482,254 |
|---|---|
| Length | 112 |
| Sequences | 13 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 69.80 |
| Shannon entropy | 0.69873 |
| G+C content | 0.58247 |
| Mean single sequence MFE | -51.92 |
| Consensus MFE | -14.01 |
| Energy contribution | -13.95 |
| Covariance contribution | -0.06 |
| Combinations/Pair | 1.70 |
| Mean z-score | -4.47 |
| Structure conservation index | 0.27 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 4.47 |
| SVM RNA-class probability | 0.999818 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 482142 112 - 27905053 UAGCAAUGAAAUCAGCCGCACUUACAAUGUGUCUGGACACCUGCUCCGACCCGGAAACGGUAUCUGAAGCUAGGCCAGCACGUUGGGAGGUCGGCGAUUU-CAGCUGCACUAC ..(((.(((((((.((((..((..(((((((.((((...(((((((.((.(((....)))..)).).))).))))))))))))))..))..)))))))))-))..)))..... ( -51.50, z-score = -5.46, R) >droSim1.chr3R 534768 112 - 27517382 UAGCAAUGAAAUCAGCCGCACUCACAAUGUGUCUGGACACCUGUUCCGAUCCGGAAACGGUAUCUGAAGCGAGGCCAGCACGUUGGGAGGUCGGAGAUUU-CAGCUGCUCCAC .((((.(((((((..(((..(((.(((((((.((((...(((((((.((((((....))).))).).))).)))))))))))))).)))..))).)))))-))..)))).... ( -49.10, z-score = -4.41, R) >droSec1.super_6 595950 112 - 4358794 UAGCAAUGAAAUCAGCCGCACUCACAAUGUGUCUGGACACCUGCUCCGAUCCGGAAACGGUAUCUGAAGCGAGGCCAGCACGUUGGGAGGUCGGAGAUUU-CAGCUGCUCCAC .((((.(((((((..(((..(((.(((((((.((((...(((((((.((((((....))).))).).))).)))))))))))))).)))..))).)))))-))..)))).... ( -51.50, z-score = -4.93, R) >droYak2.chr3R 784311 112 - 28832112 UAGCAAUAAAAUCAGCCACACUCACAAUGUGUCUAGACACCCGCUCCGAUCCGGAAACGGUAUCUGAAGCGAGGUCAGCACAUUGGGAGGUCGGAGAUUU-UAGCUGCUCCAC .((((.(((((((..((((.(((.(((((((.((.(((.(.(((((.((((((....))).))).).)))).))))))))))))).))))).)).)))))-))..)))).... ( -46.10, z-score = -5.40, R) >droEre2.scaffold_4770 791125 112 - 17746568 UAGCAAUGAAAUCAGCCGCACUCACAAUGUGUCUAGACACCUGUUCUGAUCCGGAAACGGUAUCUGAAGCGAGGCCAGCACGUUGGGAGGUCGGAGAUUU-CAGCUGCUCCAC .((((.(((((((..(((..(((.(((((((.((.(...(((((((.((((((....))).))).).))).)))).))))))))).)))..))).)))))-))..)))).... ( -42.60, z-score = -2.96, R) >droAna3.scaffold_13340 22692659 112 - 23697760 GAGCAAUAAAGUCCGCCGCACACACAAUGUGUCUGGACACCUGUUCCGACCCGGAAACGGCAUCGGAAGUGGCGCCAGCACGUAGUGUGGUAGGAGACUU-UAGCUGCUCUAC (((((.(((((((..((...(((((.(((((.((((.(.((..((((((.(((....)))..))))))..)).)))))))))).)))))...)).)))))-))..)))))... ( -58.20, z-score = -6.79, R) >dp4.chr2 9357449 106 + 30794189 GAGCUAUGAAAUCGGCCGCGCACACAAUGUGCUUGGACACCUGCUCCGAGCUGGUGCUCGGACCGGA------GCCAGCACGUUGGGUGCUCGGCGAUUU-CAGCUGCUCUAC ((((..((((((((.(((.((((.(((((((((.((.(.((.(.(((((((....)))))))).)).------)))))))))))).)))).)))))))))-))...))))... ( -62.80, z-score = -7.24, R) >droPer1.super_3 3155462 106 + 7375914 GAGCUAUGAAAUCGGCCGCGCACACAAUGUGCUUGGACACCUGCUCCGAGCUGGUGCUCGGACCGGA------GCCAGCACGUUGGGUGCUCGGCGAUUU-CAGCUGCUCUAC ((((..((((((((.(((.((((.(((((((((.((.(.((.(.(((((((....)))))))).)).------)))))))))))).)))).)))))))))-))...))))... ( -62.80, z-score = -7.24, R) >droWil1.scaffold_181089 11415948 106 + 12369635 GGGCAAUGAAAUCCGCCGCACAGACAAUGUGCUUGGACACCUGCUCCGAAGUCGAUGCGGAACUUG------CGUCAGCACGUUGUGUGGUUGGGGAUUU-CAAUUGCUCCAC ((((((((((((((.(((..((.(((((((((((((((....).)))))....((((((.....))------)))).))))))))).))..)))))))))-).))))).)).. ( -49.50, z-score = -4.61, R) >droVir3.scaffold_13047 6409786 112 - 19223366 GCGCUAUGAAAUCCGCUGCGCACACAAUGUGCUUGGACACCUGCUCCGAGCUGGUUGCCUGCUCAGGCUGUGCCUCAGCACGUCGUGUGUGCGGCGAUUG-CAGUUGCUCCAC ..((..((.((((.(((((((((((.(((((((.((.(((..(((..((((.((...)).)))).))).)))))..))))))).))))))))))))))).-))...))..... ( -53.50, z-score = -3.42, R) >droMoj3.scaffold_6540 32475028 97 + 34148556 GCGCUAUGAAAUCCGCUGCGCAGACAAUGUGCUUGGACACCUGCUCUGGGGCAGUA---------------GACGCAGCACGUUGGCUGCGCGGCGACGG-CAACUGCUCCAC ((...........((((((((((.(((((((((((..(..(((((....)))))..---------------).)).))))))))).))))))))))..(.-...).))..... ( -44.80, z-score = -2.60, R) >droGri2.scaffold_14906 11718816 112 - 14172833 GCGCUAUGAAAUCCGCUGCGCACACAAUGUGCUUGGAGACCUGCUCGGAACUGGUGGUCGCAUCCGAGAGGCCGUCAGCGCGUCGUGUGCGCGGCGAUUG-CAGCUGCUCCAC (((((....((((.(((((((((((.(((((((.(..(.(((.((((((....((....)).))))))))).)..)))))))).))))))))))))))).-.))).))..... ( -55.20, z-score = -2.81, R) >anoGam1.chr2R 50003634 112 + 62725911 GCGAAAGGUAGAAGGCACCCUUGAUGAUGCCGGCCGACACGGUGGUCGAGUCGGGCA-GGAGGGAGCGCUUGCGAACGCGAGCCGGGCCCGUGCUGCUGGACGGCUGCGUGAC ......(((....((((.(......).)))).)))..(((.(..(((..(((.((((-(.(.((.((((((((....))))))...)))).).))))).))))))..)))).. ( -47.40, z-score = -0.28, R) >consensus GAGCAAUGAAAUCAGCCGCACACACAAUGUGCCUGGACACCUGCUCCGAGCCGGAAACGGUAUCUGAAGCGACGCCAGCACGUUGGGUGGUCGGCGAUUU_CAGCUGCUCCAC ..((..(((((((..((...(((.((((((((.((((.......))))....(....)...................)))))))).)))...)).))))).))...))..... (-14.01 = -13.95 + -0.06)
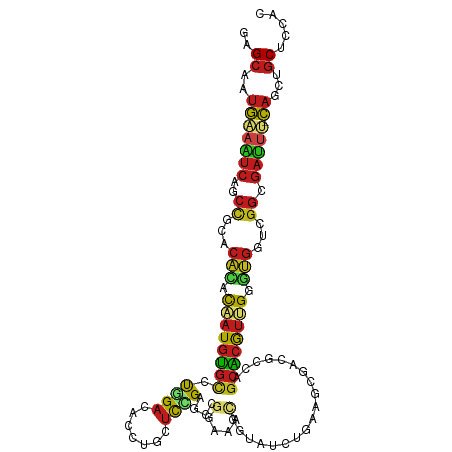
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:49:43 2011