
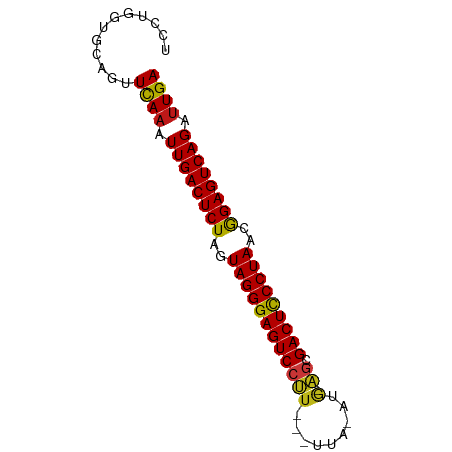
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 121,092 – 121,163 |
| Length | 71 |
| Max. P | 0.999191 |

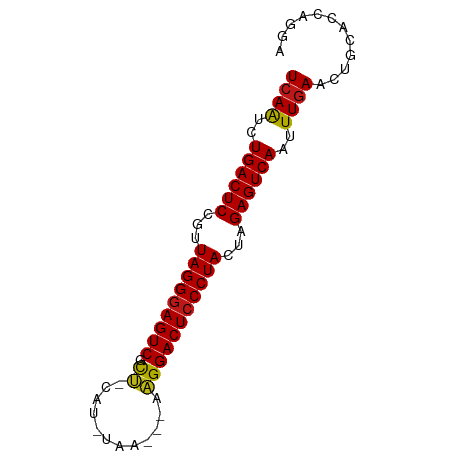
| Location | 121,092 – 121,163 |
|---|---|
| Length | 71 |
| Sequences | 15 |
| Columns | 77 |
| Reading direction | forward |
| Mean pairwise identity | 85.56 |
| Shannon entropy | 0.33302 |
| G+C content | 0.46315 |
| Mean single sequence MFE | -32.10 |
| Consensus MFE | -28.73 |
| Energy contribution | -28.69 |
| Covariance contribution | -0.03 |
| Combinations/Pair | 1.17 |
| Mean z-score | -5.17 |
| Structure conservation index | 0.89 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.69 |
| SVM RNA-class probability | 0.999170 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
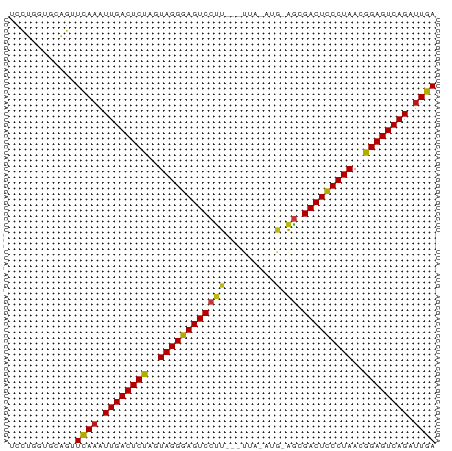
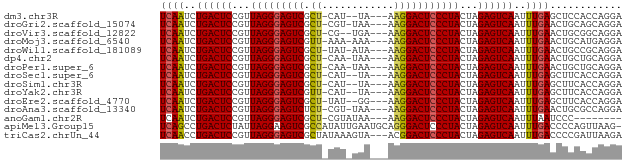
>dm3.chr3R 121092 71 + 27905053 UCCUGGUGGAGCUCAAAUUGACUCUAGUAGGGAGUCCUU---UA--AUG-AGCGACUCCCUAACGGAGUCAGAUUGA (((....)))..((((.((((((((..((((((((((((---..--..)-)).)))))))))..)))))))).)))) ( -35.30, z-score = -5.59, R) >droGri2.scaffold_15074 5274722 72 - 7742996 UCCUGCUGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUU---UUA-ACG-AGCGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..((((((((((((---...-..)-)).)))))))))..)))))))).)))) ( -32.30, z-score = -5.27, R) >droVir3.scaffold_12822 1418816 71 + 4096053 UCCUGCCGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUU---UCA--CG-AGCGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..((((((((((((---...--.)-)).)))))))))..)))))))).)))) ( -32.30, z-score = -5.10, R) >droMoj3.scaffold_6540 34020290 72 + 34148556 UCCUCAUGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUU---UUU-UUU-AACGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..(((((((((.((---...-...-)).)))))))))..)))))))).)))) ( -30.10, z-score = -5.66, R) >droWil1.scaffold_181089 2543965 72 - 12369635 UCCUGCGGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUU---UAU-AUA-AGCGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..((((((((((((---...-..)-)).)))))))))..)))))))).)))) ( -32.60, z-score = -5.58, R) >dp4.chr2 5983258 72 - 30794189 UCCUGCAGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUU---UUA-UUG-AGCGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..((((((((((((---...-..)-)).)))))))))..)))))))).)))) ( -33.00, z-score = -5.62, R) >droPer1.super_6 6029783 72 - 6141320 UCCUGCAGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUU---UUA-UUG-AGCGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..((((((((((((---...-..)-)).)))))))))..)))))))).)))) ( -33.00, z-score = -5.62, R) >droSec1.super_6 240435 71 + 4358794 UCCUGGUGAAGCUCAAAUUGACUCUAGUAGGGAGUCCUU---UA--AUG-AGCGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..((((((((((((---..--..)-)).)))))))))..)))))))).)))) ( -32.90, z-score = -5.31, R) >droSim1.chr3R 194563 71 + 27517382 UCCUGGUGAAGCUCAAAUUGACUCUAGUAGGGAGUCCUU---UA--AUG-AGCGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..((((((((((((---..--..)-)).)))))))))..)))))))).)))) ( -32.90, z-score = -5.31, R) >droYak2.chr3R 481105 71 + 28832112 UCCUGGUGAAGCUCAAAUUGACUCUAGUAGGGAGUCCUU---UA--AUG-AACGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..(((((((((.((---(.--..)-)).)))))))))..)))))))).)))) ( -29.70, z-score = -4.55, R) >droEre2.scaffold_4770 471047 71 + 17746568 UCCUGGUGAAGCUCAAAUUGACUCUAGUAGGGAGUCCUU---CC--AUA-AGCGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..((((((((((((---..--..)-)).)))))))))..)))))))).)))) ( -31.70, z-score = -4.84, R) >droAna3.scaffold_13340 11127735 72 + 23697760 UCCUGGCGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUU---UUA-ACG-AGAGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..((((((((((((---...-..)-)).)))))))))..)))))))).)))) ( -32.30, z-score = -4.86, R) >anoGam1.chr2R 22086628 65 - 62725911 --------GGGAUUAAAUUGACUCUAGUAGGGAGUCCUU---UUAUACG-AGCGACUCCCUAACGGAGUCAGAUUGA --------....((((.((((((((..((((((((((((---......)-)).)))))))))..)))))))).)))) ( -30.40, z-score = -5.61, R) >apiMel3.Group15 2995813 76 + 7856270 -CUUAACUGGGGUCAAAUUGACUCUAGUAGGGAGUCCCUGCAUUCAAUAUGGCGACUUCCUAAUAGAGUCAGGCUGA -...........(((..(((((((((.(((((((((((............)).))))))))).)))))))))..))) ( -30.60, z-score = -3.92, R) >triCas2.chrUn_44 245636 74 - 525920 UCUUAAUCGGGGUCAAAUUGACUCUAGUAGGGAGUCCGU---UACUUUAUAGCGACUCCCUAACGGAGUCAGGUUGA ............((((.((((((((..(((((((((.((---((.....)))))))))))))..)))))))).)))) ( -32.40, z-score = -4.72, R) >consensus UCCUGGUGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUU___UUA_AUG_AGCGACUCCCUAACGGAGUCAGAUUGA ............((((.((((((((..(((((((((((............)).)))))))))..)))))))).)))) (-28.73 = -28.69 + -0.03)

| Location | 121,092 – 121,163 |
|---|---|
| Length | 71 |
| Sequences | 15 |
| Columns | 77 |
| Reading direction | reverse |
| Mean pairwise identity | 85.56 |
| Shannon entropy | 0.33302 |
| G+C content | 0.46315 |
| Mean single sequence MFE | -27.87 |
| Consensus MFE | -25.61 |
| Energy contribution | -25.52 |
| Covariance contribution | -0.09 |
| Combinations/Pair | 1.14 |
| Mean z-score | -4.19 |
| Structure conservation index | 0.92 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.70 |
| SVM RNA-class probability | 0.999191 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 121092 71 - 27905053 UCAAUCUGACUCCGUUAGGGAGUCGCU-CAU--UA---AAGGACUCCCUACUAGAGUCAAUUUGAGCUCCACCAGGA ((((..((((((...(((((((((.((-...--..---.)))))))))))...))))))..))))..(((....))) ( -29.40, z-score = -4.53, R) >droGri2.scaffold_15074 5274722 72 + 7742996 UCAAUCUGACUCCGUUAGGGAGUCGCU-CGU-UAA---AAGGACUCCCUACUAGAGUCAAUUUGAACUGCAGCAGGA ((((..((((((...(((((((((.((-...-...---.)))))))))))...))))))..)))).(((...))).. ( -28.00, z-score = -3.79, R) >droVir3.scaffold_12822 1418816 71 - 4096053 UCAAUCUGACUCCGUUAGGGAGUCGCU-CG--UGA---AAGGACUCCCUACUAGAGUCAAUUUGAACUGCGGCAGGA ((((..((((((...(((((((((.((-..--...---.)))))))))))...))))))..)))).(((...))).. ( -28.50, z-score = -3.21, R) >droMoj3.scaffold_6540 34020290 72 - 34148556 UCAAUCUGACUCCGUUAGGGAGUCGUU-AAA-AAA---AAGGACUCCCUACUAGAGUCAAUUUGAACUGCAUGAGGA ((((..((((((...(((((((((.((-...-...---)).)))))))))...))))))..))))............ ( -28.80, z-score = -5.02, R) >droWil1.scaffold_181089 2543965 72 + 12369635 UCAAUCUGACUCCGUUAGGGAGUCGCU-UAU-AUA---AAGGACUCCCUACUAGAGUCAAUUUGAACUGCCGCAGGA ((((..((((((...(((((((((.((-(..-...---))))))))))))...))))))..)))).(((...))).. ( -29.50, z-score = -4.79, R) >dp4.chr2 5983258 72 + 30794189 UCAAUCUGACUCCGUUAGGGAGUCGCU-CAA-UAA---AAGGACUCCCUACUAGAGUCAAUUUGAACUGCUGCAGGA ((((..((((((...(((((((((.((-...-...---.)))))))))))...))))))..)))).(((...))).. ( -28.00, z-score = -4.18, R) >droPer1.super_6 6029783 72 + 6141320 UCAAUCUGACUCCGUUAGGGAGUCGCU-CAA-UAA---AAGGACUCCCUACUAGAGUCAAUUUGAACUGCUGCAGGA ((((..((((((...(((((((((.((-...-...---.)))))))))))...))))))..)))).(((...))).. ( -28.00, z-score = -4.18, R) >droSec1.super_6 240435 71 - 4358794 UCAAUCUGACUCCGUUAGGGAGUCGCU-CAU--UA---AAGGACUCCCUACUAGAGUCAAUUUGAGCUUCACCAGGA ((((..((((((...(((((((((.((-...--..---.)))))))))))...))))))..))))............ ( -28.20, z-score = -4.21, R) >droSim1.chr3R 194563 71 - 27517382 UCAAUCUGACUCCGUUAGGGAGUCGCU-CAU--UA---AAGGACUCCCUACUAGAGUCAAUUUGAGCUUCACCAGGA ((((..((((((...(((((((((.((-...--..---.)))))))))))...))))))..))))............ ( -28.20, z-score = -4.21, R) >droYak2.chr3R 481105 71 - 28832112 UCAAUCUGACUCCGUUAGGGAGUCGUU-CAU--UA---AAGGACUCCCUACUAGAGUCAAUUUGAGCUUCACCAGGA ((((..((((((...(((((((((.((-...--..---)).)))))))))...))))))..))))............ ( -28.30, z-score = -4.33, R) >droEre2.scaffold_4770 471047 71 - 17746568 UCAAUCUGACUCCGUUAGGGAGUCGCU-UAU--GG---AAGGACUCCCUACUAGAGUCAAUUUGAGCUUCACCAGGA ((((..((((((...(((((((((.((-(..--..---))))))))))))...))))))..))))............ ( -29.30, z-score = -3.73, R) >droAna3.scaffold_13340 11127735 72 - 23697760 UCAAUCUGACUCCGUUAGGGAGUCUCU-CGU-UAA---AAGGACUCCCUACUAGAGUCAAUUUGAACUGCGCCAGGA ((((..((((((...(((((((((.((-...-...---.)))))))))))...))))))..)))).(((...))).. ( -28.00, z-score = -3.83, R) >anoGam1.chr2R 22086628 65 + 62725911 UCAAUCUGACUCCGUUAGGGAGUCGCU-CGUAUAA---AAGGACUCCCUACUAGAGUCAAUUUAAUCCC-------- ......((((((...(((((((((.((-.......---.)))))))))))...))))))..........-------- ( -24.20, z-score = -4.91, R) >apiMel3.Group15 2995813 76 - 7856270 UCAGCCUGACUCUAUUAGGAAGUCGCCAUAUUGAAUGCAGGGACUCCCUACUAGAGUCAAUUUGACCCCAGUUAAG- ((((..((((((((.((((.((((.((............)))))).)))).))))))))..))))...........- ( -25.60, z-score = -3.84, R) >triCas2.chrUn_44 245636 74 + 525920 UCAACCUGACUCCGUUAGGGAGUCGCUAUAAAGUA---ACGGACUCCCUACUAGAGUCAAUUUGACCCCGAUUAAGA ((((..((((((...((((((((((((....))).---...)))))))))...))))))..))))............ ( -26.10, z-score = -4.14, R) >consensus UCAAUCUGACUCCGUUAGGGAGUCGCU_CAU_UAA___AAGGACUCCCUACUAGAGUCAAUUUGAACUGCACCAGGA ((((..((((((...(((((((((.((............)))))))))))...))))))..))))............ (-25.61 = -25.52 + -0.09)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:49:17 2011