| Sequence ID | dm3.chr3RHet |
|---|---|
| Location | 373,384 – 373,498 |
| Length | 114 |
| Max. P | 0.998001 |

| Location | 373,384 – 373,498 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.59 |
| Shannon entropy | 0.45768 |
| G+C content | 0.58466 |
| Mean single sequence MFE | -43.87 |
| Consensus MFE | -26.48 |
| Energy contribution | -26.65 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.37 |
| Mean z-score | -2.92 |
| Structure conservation index | 0.60 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.23 |
| SVM RNA-class probability | 0.998001 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3RHet 373384 114 + 2517507 CUAUAGUCACCGAUCGUGGCCACUACCUGGUCCUGAUCCACACU-CUGUACCACACCACAGCUACAGAGGCGACAACGAGGUGACUG--UGUUGUGGGCAGCAACUCGCCGCAACAC--- ....((((((((((((.(((((.....))))).)))))....((-(((((.(........).)))))))..........)))))))(--((((((((.(........))))))))))--- ( -44.30, z-score = -2.90, R) >droAna3.scaffold_12911 3477394 110 - 5364042 -------CACUGAUCGUGGCCACCACCUGGUCUUGAUCUGCUCUGCAAUGCCACACGA---CUGUAGAGGCACCUGCCGAGUGACCGGCCGUUGUGGGCAGCCACUUGCCACAACGGUAC -------....(((((.(((((.....))))).))))).((((.(((.((((.(((..---..)).).))))..))).)))).....((((((((((.(((....))))))))))))).. ( -43.60, z-score = -1.74, R) >droEre2.scaffold_4845 20484262 98 - 22589142 CUAUAGUCACCGAUCGUGGCCACCACCUGGUCCCGAUCCGCACU-C----------------UACAGAGGCGACAUCGAGGUGACUA--UGUUGUGGGCAGCAACGUGCCGCAACAC--- ..((((((((((((((.(((((.....))))).)))))(((.((-(----------------....)))))).......))))))))--)(((((((.((......)))))))))..--- ( -45.20, z-score = -4.40, R) >droYak2.chrU 25442332 114 - 28119190 CUACAGUCACCUAUCGGGGCCACCACCUUGACCCGAUCCGCACU-CAGUACCACACCAGAGCCACGGAUACGACAACAAGGUGGCUG--UGUUGUGGGCAGAAACGUAUCUUAACAC--- .(((((((((((.((((((......))))))..(((((((..((-(............)))...))))).))......)))))))))--))..(((...(((......)))...)))--- ( -33.60, z-score = -0.77, R) >droSec1.super_94 7367 114 - 93855 CCAUAGUCGCCGAUCGGGGCCACCACCUGGUCCUGAUCUGCACU-CUGUACCACACCACAGCUACAGAGACGUCAACGAAGCGACUG--UGUUGUGGGCAGCAACUCGCCGCAACAC--- ....((((((.(((((((((((.....)))))))))))....((-(((((.(........).)))))))...........))))))(--((((((((.(........))))))))))--- ( -48.30, z-score = -3.93, R) >droSim1.chrU 10210250 114 - 15797150 CUAUAGUCACCGAUCGUGGCCACCACCUGGUCCAGAUCCGCACU-CUGUACCACACCACAGCUACAGAGGCGACAACGAGGUGACUG--UGUUGUGGGCAGCAACUCGCCGCAACAC--- ....(((((((((((.((((((.....))).)))))))(((.((-(((((.(........).)))))))))).......)))))))(--((((((((.(........))))))))))--- ( -48.20, z-score = -3.80, R) >consensus CUAUAGUCACCGAUCGUGGCCACCACCUGGUCCUGAUCCGCACU_CUGUACCACACCACAGCUACAGAGGCGACAACGAGGUGACUG__UGUUGUGGGCAGCAACUCGCCGCAACAC___ ...(((((((((((((.(((((.....))))).)))))..............................(....).....))))))))..((((((((...........)))))))).... (-26.48 = -26.65 + 0.17)

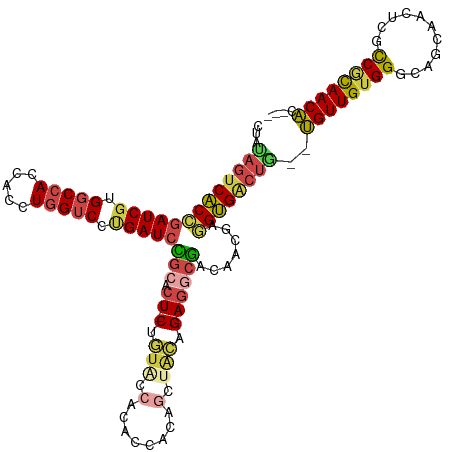
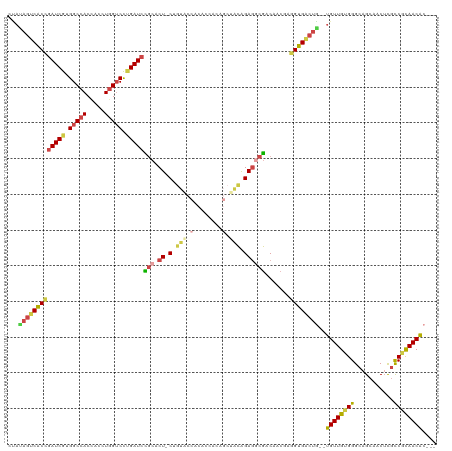
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:47:38 2011