
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 24,028,695 – 24,028,807 |
| Length | 112 |
| Max. P | 0.899914 |

| Location | 24,028,695 – 24,028,807 |
|---|---|
| Length | 112 |
| Sequences | 14 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.44 |
| Shannon entropy | 0.41251 |
| G+C content | 0.47483 |
| Mean single sequence MFE | -33.39 |
| Consensus MFE | -19.40 |
| Energy contribution | -19.53 |
| Covariance contribution | 0.13 |
| Combinations/Pair | 1.52 |
| Mean z-score | -1.96 |
| Structure conservation index | 0.58 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.15 |
| SVM RNA-class probability | 0.899914 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 24028695 112 - 24543557 --------GCAGCAUCUACGUUUGCUGGAGACUCAUCGUUCGGAUCGGCCAGCAUUGAUAUUACAGAUAUCAAGAUAGUCUCCACUGUAUGCACGGGUAACCAGCGCUCCGAUGCUUUUU --------..((((((...((.(((((((((((.((((((.((.....))))).((((((((...)))))))))))))))))))..))).))..((....))........)))))).... ( -34.90, z-score = -2.07, R) >apiMel3.GroupUn 347714064 112 - 399230636 --------GCAGCAUCAACAUUUGCACCACUUUCAUCGUUUGGCUCUGCUAACAUACUUACCACACUUAACAAUAUUUUUUCUACACUUUGAACUGGACUCCAUCUUUCUGCACUACUUU --------((((((........))).(((........(((.(((...))))))..........................(((........))).)))............)))........ ( -10.40, z-score = -0.81, R) >anoGam1.chr2R 44530203 112 + 62725911 --------GCCGCGUCCACGUUCGCCGGCGACUCGUCGUUCGGGUCGGCCAGCAUCGAAAUGACCGAUAUUAGAAUCGUCUCGACCGUGUGCACCGGAAGCCAGCGCUCCGAUGCCUUCU --------((((.(((.((((((((.(((((((((.....)))))).))).))((((.......))))....))).)))...))))).))(((.((((.((....)))))).)))..... ( -38.00, z-score = 0.35, R) >droGri2.scaffold_15116 1446200 112 + 1808639 --------GCAGCAUCAACAUUUGCUGGGGACUCAUCGUUGGGAUCCGCUAACAUUGAUAUCACGGAUAUCAAAAUAGUUUCCACUGUGUGCACCGGGAGCCAACGCUCUGAUGCUUUUU --------..(((((((.......(((((((((((....)))).)))((.....((((((((...)))))))).(((((....)))))..)).))))((((....))))))))))).... ( -35.70, z-score = -2.43, R) >droMoj3.scaffold_6540 4286880 112 - 34148556 --------GCAGCAUCAACGUUUGCUGGGGACUCGUCAUUGGGAUCCGCUAACAUUGAUAUCACGGAUAUCAAAAUAGUUUCCACUGUGUGUACCGGCAACCAACGCUCUGAUGCUUUUU --------..(((((((.((((((((((..((.((.((.(((((...((((...((((((((...))))))))..))))))))).)))).)).))))))...))))...))))))).... ( -36.90, z-score = -3.23, R) >droVir3.scaffold_12958 2012078 112 - 3547706 --------GCAGCAUCAACAUUUGCUGGGGAUUCGUCGUUGGGAUCCGCUAACAUUGAUAUCACGGAUAUCAAAAUAGUUUCCACUGUGUGCACCGGCAACCAACGCUCUGAUGCUUUUU --------..(((((((....(((((((((((((.......))))))((.....((((((((...)))))))).(((((....)))))..)).))))))).........))))))).... ( -33.72, z-score = -2.09, R) >droWil1.scaffold_181130 2843699 112 + 16660200 --------GCAGCAUCCACAUUUGCUGGAGACUCAUCGUUGGGAUCAGCUAGCAUUGAUAUCACAGAUAUCAAAAUUGUUUCCACUGUGUGUACAGGCAGCCAGCGCUCGGAUGCUUUCU --------..(((((((.....((((((((((.....(((((......))))).((((((((...))))))))....)))))))((((....)))))))((....))..))))))).... ( -36.20, z-score = -1.87, R) >droPer1.super_6 5670116 112 + 6141320 --------GCGGCAUCUACAUUUGCUGGAGACUCAUCGUUGGGAUCAGCCAACAUUGAUAUCACUGAUAUCAAGAUAGUUUCCACUGUAUGCACGGGUAACCAGCGCUCAGAUGCUUUUU --------..(((((((.....(((((((((((.((((((((......))))).((((((((...)))))))))))))))))))..))).((.(((....)).).))..))))))).... ( -38.20, z-score = -3.55, R) >dp4.chr2 5628146 112 + 30794189 --------GCGGCAUCUACAUUUGCUGGAGACUCGUCGUUGGGAUCAGCCAACAUUGAUAUCACUGAUAUCAAGAUAGUUUCCACUGUAUGCACGGGUAACCAGCGCUCAGAUGCUUUUU --------..(((((((.....((((((..((((((((((((......))))).((((((((...)))))))).(((((....)))))..).))))))..))))))...))))))).... ( -39.80, z-score = -3.78, R) >droEre2.scaffold_4784 23478588 112 - 25762168 --------GCAGCAUCAACGUUUGCUGGAGACUCAUCGUUCGGAUCGGCCAGCAUUGAUAUUACAGAUAUCAAGAUUGUCUCUACUGUGUGCACGGGUAACCAGCGCUCCGAUGCUUUUU --------..((((((.........(((((((..((((((.((.....))))).((((((((...))))))))))).)))))))....((((..((....)).))))...)))))).... ( -32.40, z-score = -0.91, R) >droYak2.chrU 23670302 112 - 28119190 --------GCAGCAUCAACGUUUGCUGGAGACUCAUCGUUUGGAUCGGCCAGCAUUGAUAUUACAGAUAUCAAGAUGGUCUCCACUGUAUGCACGGGUAACCAGCGCUCCGAUGCUUUUU --------..((((((...(..((((((..((((..(((.((((..(((((...((((((((...))))))))..)))))))))....)))...))))..))))))..).)))))).... ( -37.40, z-score = -2.50, R) >droSec1.super_64 132505 112 - 179039 --------GCAGCAUCUACGUUUGCUGGAGACUCAUCGUUCGGAUCGGCCAGCAUUGAUAUUACAGAUAUCAAGAUAGUCUCCACUGUAUGCACGGGUAACCACCGCUCCGAUGCUUUUU --------..((((((...((.(((((((((((.((((((.((.....))))).((((((((...)))))))))))))))))))..))).)).(((.......)))....)))))).... ( -35.90, z-score = -2.75, R) >droSim1.chr3h_random 1126106 112 - 1452968 --------GCAGCAUCUACGUUUGCUGGAGACUCAUCGUUCGGAUCGGCCAGCAUUGAUAUUACAGAUAUCAAGAUAGUCUCCACUGUAUGCACGGGUAACCACCGCUCCGAUGCUUUUU --------..((((((...((.(((((((((((.((((((.((.....))))).((((((((...)))))))))))))))))))..))).)).(((.......)))....)))))).... ( -35.90, z-score = -2.75, R) >triCas2.ChLG6 4577598 120 + 13544221 CUCUUACCGCAGCAUCGACAUUCGCAGGUGAUUCAUCGUUUGGGUCGGCUAACAUUGAAAUAACGGAUAUUAAAAUAGUCUCUACAGUAUGAACUGGCAGCCACCUUUCUGACGCUUUUU ...(((((...((..........)).))))).....((((.((((.((((..............(((.((((...)))).))).((((....))))..))))))))....))))...... ( -22.00, z-score = 1.00, R) >consensus ________GCAGCAUCAACAUUUGCUGGAGACUCAUCGUUGGGAUCGGCCAACAUUGAUAUCACAGAUAUCAAGAUAGUCUCCACUGUAUGCACGGGUAACCAGCGCUCCGAUGCUUUUU ..........((((((..........((((((.....(((.((.....))))).((((((((...))))))))....)))))).((((....))))..............)))))).... (-19.40 = -19.53 + 0.13)

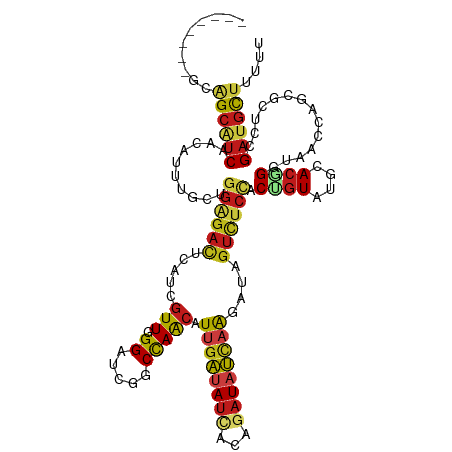
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:46:47 2011