
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 22,213,219 – 22,213,309 |
| Length | 90 |
| Max. P | 0.562479 |

| Location | 22,213,219 – 22,213,309 |
|---|---|
| Length | 90 |
| Sequences | 11 |
| Columns | 90 |
| Reading direction | forward |
| Mean pairwise identity | 86.61 |
| Shannon entropy | 0.26907 |
| G+C content | 0.61116 |
| Mean single sequence MFE | -36.71 |
| Consensus MFE | -24.52 |
| Energy contribution | -24.77 |
| Covariance contribution | 0.26 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.67 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.562479 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
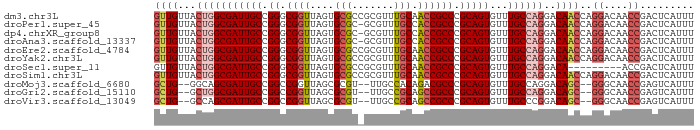
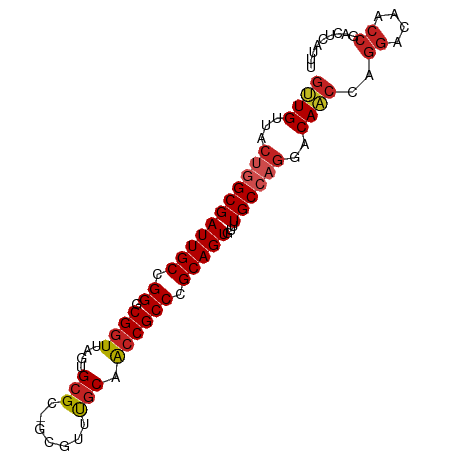
>dm3.chr3L 22213219 90 + 24543557 GUUGUUACUGGCGAUUGCCGGGCGGUUAGUGCGCCGCGUUUGCAACCGCCCGCAGUGUUUGCCAGGACAACCAGGACAACCGACUCAUUU ((((((.(((((((((((.((((((((.(((((...)))..)))))))))))))))...))))))))))))..((....))......... ( -37.10, z-score = -2.16, R) >droPer1.super_45 432528 89 - 618639 GUUGUUACUGGCGAUUGCCGGGCGGUUAGUGCGC-GCGUUUGCCACCGCCCGCAGUGUUUGCCAGGACAACCAGGACAACCGACUCAUUU ((((((.(((((((((((.(((((((..(.(((.-.....))))))))))))))))...))))))))))))..((....))......... ( -35.80, z-score = -1.79, R) >dp4.chrXR_group8 6296518 89 + 9212921 GUUGUUACUGGCGAUUGCCGGGCGGUUAGUGCGC-GCGUUUGCCACCGCCCGCAGUGUUUGCCAGGACAACCAGGACAACCGACUCAUUU ((((((.(((((((((((.(((((((..(.(((.-.....))))))))))))))))...))))))))))))..((....))......... ( -35.80, z-score = -1.79, R) >droAna3.scaffold_13337 21373314 89 + 23293914 GUUGUUACUGGCGAUUGCCGGGCGGUUAGUGCGC-GCGUUUGCCACCGCCCGCAGUGUUUGCCAGGACAACCAGGACAACCGACUCAUUU ((((((.(((((((((((.(((((((..(.(((.-.....))))))))))))))))...))))))))))))..((....))......... ( -35.80, z-score = -1.79, R) >droEre2.scaffold_4784 21884392 90 + 25762168 GUUGUUACUGGCGAUUGCCGGGCGGUUAGUGCGCCGCGUUUGCAACCGCCCGCAGUGUUUGCCAGGACAACCAGGACAACCGACUCAUUU ((((((.(((((((((((.((((((((.(((((...)))..)))))))))))))))...))))))))))))..((....))......... ( -37.10, z-score = -2.16, R) >droYak2.chr3L 22600009 90 + 24197627 GUUGUUACUGGCGAUUGCCGGGCGGUUAGUGCGCCGCGUUUGCAACCGCCCGCAGUGUUUGCCAGGACAACCAGGACAACCGACUCAUUU ((((((.(((((((((((.((((((((.(((((...)))..)))))))))))))))...))))))))))))..((....))......... ( -37.10, z-score = -2.16, R) >droSec1.super_11 2143785 81 + 2888827 GUUGUUACUGGCGAUUGCCGGGCGGUUAGUGCGCCGCGUUUGCAACCGCCCGCAGUGUUUGCCAGGACA---------ACCGACUCAUUU ((((((.(((((((((((.((((((((.(((((...)))..)))))))))))))))...))))))))))---------)).......... ( -36.40, z-score = -2.87, R) >droSim1.chr3L 21564214 90 + 22553184 GUUGUUACUGGCGAUUGCCGGGCGGUUAGUGCGCCGCGUUUGCAACCGCCCGCAGUGUUUGCCAGGACAACCAGGACAACCGACUCAUUU ((((((.(((((((((((.((((((((.(((((...)))..)))))))))))))))...))))))))))))..((....))......... ( -37.10, z-score = -2.16, R) >droMoj3.scaffold_6680 19558061 84 + 24764193 GCUG--GGCAGCGAUUGCCGGCCGGUUAGCGCGU--UUGCCACAGACGCCCGCAGUGUUUGCCAGGACAGC--GGGCAACCGAGUCAUUU ....--(((((...)))))((((((((.((((((--(((...)))))))((((..(((((....)))))))--))))))))).))).... ( -34.10, z-score = -0.76, R) >droGri2.scaffold_15110 6621368 84 - 24565398 GCUG--GCUGGCGAUUGCCGGCCGGUUAGCGCGU--UUGCCGCAGCCGCCCGCAGUGUUUGCCAGGACAGC--GGGCAACCGAGUCAUUU .(((--((..((.(((((.(((.((((.(((.(.--...))))))))))).)))))))..)))))(((..(--((....))).))).... ( -41.20, z-score = -1.77, R) >droVir3.scaffold_13049 14778765 84 + 25233164 GCUG--GCCAGCGAUUGCCGGCCGGUUAGCGCGU--UUGCCGCAGCCGCCCGCAGUGUUUGCCCGGACAGC--GGGCAACCGAGUCAUUU .(.(--((.(((.(((((.(((.((((.(((.(.--...))))))))))).)))))))).))).)(((..(--((....))).))).... ( -36.30, z-score = -0.56, R) >consensus GUUGUUACUGGCGAUUGCCGGGCGGUUAGUGCGC_GCGUUUGCAACCGCCCGCAGUGUUUGCCAGGACAACCAGGACAACCGACUCAUUU ((((...(((((((((((.((.((((....(((.......))).)))))).)))))...))))))..))))..((....))......... (-24.52 = -24.77 + 0.26)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:44:08 2011