
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 21,978,816 – 21,978,935 |
| Length | 119 |
| Max. P | 0.750426 |

| Location | 21,978,816 – 21,978,935 |
|---|---|
| Length | 119 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.22 |
| Shannon entropy | 0.26199 |
| G+C content | 0.59129 |
| Mean single sequence MFE | -41.63 |
| Consensus MFE | -31.07 |
| Energy contribution | -30.00 |
| Covariance contribution | -1.07 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.75 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.750426 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 21978816 119 + 24543557 AUCAUGUUC-GAGACGUUCAACUCCCCGGCCAUGUACGUGGCCAUCCAGGCCGUGCUCUCCCUGUACGCCUCCGGCCGUACCACCGGUAUCGUCCUGGACUCCGGUGACGGUGUCUCCCA .........-(((((............((((((....))))))....((((.((((.......))))))))...(((((..((((((..((......))..))))))))))))))))... ( -45.80, z-score = -2.19, R) >droSim1.chr3L 21318659 119 + 22553184 AUCAUGUUC-GAGACCUUCAACUCCCCGGCCAUGUACGUGGCCAUCCAGGCCGUGCUCUCCCUGUACGCCUCCGGCCGUACCACCGGUAUCGUCCUGGACUCCGGUGACGGUGUCUCCCA .........-(((((............((((((....))))))....((((.((((.......))))))))...(((((..((((((..((......))..))))))))))))))))... ( -44.90, z-score = -2.34, R) >droSec1.super_11 1904483 119 + 2888827 AUCAUGUUC-GAGACCUUCAACUCCCCGGCCAUGUACGUGGCCAUCCAGGCCGUGCUCUCCCUGUACGCCUCCGGCCGUACCACCGGUAUCGUCCUGGACUCCGGGGACGGUGUCUCCCA .........-(((((((.....(((((((((((....)))))).((((((.((((((......(((((((...)).)))))....)))).)).))))))....))))).)).)))))... ( -47.80, z-score = -2.24, R) >droYak2.chr3L_random 4416676 119 + 4797643 AUCAUGUUC-GAGACCUUUAACUCGCCUGCCAUGUACGUGGCCAUCCAAGCCGUGCUCUCCCUGUACGCCUCCGGCCGUACUACCGGUAUCGUCCUGGACUCCGGUGACGGUGUCUCCCA .........-(((((.............(((((....))))).......(((((.........(((((((...)).)))))((((((..((......))..))))))))))))))))... ( -36.70, z-score = -1.20, R) >droEre2.scaffold_4784 21653905 119 + 25762168 AUCAUGUUC-GAGACCUUCAACUCCCCGGCCAUGUACGUGGCCAUCCAGGCCGUGCUCUCCCUGUACGCCUCCGGCCGUACUACUGGCAUCGUCCUGGACUCUGGUGACGGUGUCUCCCA .........-(((((............((((((....)))))).((((((.((((((......(((((((...)).)))))....)))).)).))))))..(((....))).)))))... ( -44.40, z-score = -2.39, R) >droAna3.scaffold_13337 21113500 119 + 23293914 AUCAUGUUC-GAGACCUUCAACUCGCCUGCCAUGUACGUCGCCAUCCAGGCCGUGCUCUCCCUGUACGCCUCCGGCCGUACCACCGGCAUCGUGCUCGACUCCGGCGACGGCGUCUCCCA .........-(((((((.....(((((((((..(((((..(((....((((.((((.......))))))))..))))))))....))))(((....)))....))))).)).)))))... ( -39.30, z-score = -1.47, R) >dp4.chrXR_group8 6052108 119 + 9212921 AUUAUGUUU-GAGACAUUCAACUCCCCAGCCAUGUACGUCGCCAUCCAGGCGGUGCUCUCCCUGUACGCCUCCGGCCGUACCACCGGUAUCGUUUUGGAUUCCGGCGAUGGUGUCUCGCA .........-(((((((.........((....))..(((((((((((((((((((((......(((((((...)).)))))....))))))).)))))))...))))))))))))))... ( -41.30, z-score = -2.07, R) >droPer1.super_45 186606 120 - 618639 AUUAUGUUUUGAGACAUUCAACUCUCCAGCCAUGUACGUCGCCAUCCAGGCAGUGCUCUCCCUGUACGCCUCCGGCCGUACCACCGGUAUCGUUUUGGAUUCCGGUGAUGGUGUCUCGCA ..........(((((.............((((((((((..(((....((((.((((.......))))))))..))))))))((((((.(((......))).))))))))))))))))... ( -41.71, z-score = -2.99, R) >droWil1.scaffold_180949 4541257 119 - 6375548 AUUAUGUUU-GAGACCUUCAACUCACCGGCCAUGUAUGUGGCCAUUCAGGCUGUAUUGUCCCUGUAUGCCUCCGGUCGUACCACUGGCAUUGUGUUGGACUCUGGUGAUGGUGUUUCUCA .........-(((((..(((..(((((((((((....))))))..............((((....(((((...((.....))...)))))......))))...)))))))).)))))... ( -36.90, z-score = -0.88, R) >droMoj3.scaffold_6680 15303910 119 + 24764193 AUCAUGUUC-GAGACCUUCAACUCCCCAGCCAUGUACGUGGCCAUCCAGGCGGUGCUCUCGCUCUAUGCCUCCGGCCGUACCACUGGCAUUGUUCUCGAUUCCGGCGACGGUGUUUCCCA .......((-((((....(((....((((....((((..((((....(((((..((....))....)))))..))))))))..))))..))).))))))..(((....)))......... ( -33.90, z-score = 0.04, R) >droGri2.scaffold_15110 1816084 119 - 24565398 AUCAUGUUC-GAGACCUUCAACUCGCCAGCCAUGUACGUGGCCAUUCAGGCAGUUCUCUCCCUGUACGCCUCCGGUCGUACCACGGGCAUUGUCCUGGAUUCCGGUGACGGUGUCUCGCA ........(-(((((((.....(((((.(((((....))))).(((((((((((.(((.....(((((((...)).)))))...))).)))).)))))))...))))).)).)))))).. ( -41.50, z-score = -1.53, R) >droVir3.scaffold_13049 20949206 119 + 25233164 AUCAUGUUC-GAGACCUUCAACUCACCAGCCAUGUAUGUGGCCAUUCAGGCGGUUCUCUCGCUAUACGCCUCCGGUCGUACCACGGGUAUUGUCCUGGACUCCGGUGAUGGUGUCUCGCA ........(-(((((..(((..(((((.(((((....)))))..((((((((((...((((...((((((...)).))))...)))).)))).))))))....)))))))).)))))).. ( -41.30, z-score = -1.52, R) >anoGam1.chrU 31374882 119 + 59568033 AUCAUGUUU-GAGACCUUCAACUCGCCGGCCAUGUACGUCGCCAUCCAGGCCGUGCUGUCCCUGUACGCUUCCGGUCGUACCACCGGUAUUGUGCUGGACUCCGGCGAUGGUGUCUCCCA .........-(((((..(((..(((((((....(((((..(((.....)))))))).((((..(((((...(((((......)))))...))))).)))).)))))))))).)))))... ( -48.60, z-score = -3.35, R) >apiMel3.Group5 610801 119 - 13386189 AUCAUGUUC-GAAACCUUCAACAGUCCGGCCAUGUACGUGGCCAUCCAGGCCGUGCUGUCCCUGUACGCUUCAGGUCGUACCACCGGCAUCGUCCUGGACUCCGGGGACGGCGUCUCCCA ....((((.-((.....))))))....((((((....)))))).....((..((((((((((((...(.((((((.((..((...))...)).)))))))..))))))))))).)..)). ( -43.50, z-score = -1.16, R) >triCas2.ChLG3 1568130 119 + 32080666 AUCAUGUUU-GAGACCUUCAACACCCCCGCCAUGUAUGUCGCCAUCCAGGCCGUGCUCUCCUUGUACGCCUCCGGUCGUACCACCGGUAUUGUCUUGGACUCUGGCGAUGGUGUCACCCA ....((..(-((.(((....(((.........)))..((((((((((((((.((((.......))))))).(((((......)))))........))))...)))))))))).)))..)) ( -36.80, z-score = -1.32, R) >consensus AUCAUGUUC_GAGACCUUCAACUCCCCGGCCAUGUACGUGGCCAUCCAGGCCGUGCUCUCCCUGUACGCCUCCGGCCGUACCACCGGUAUCGUCCUGGACUCCGGUGACGGUGUCUCCCA ..........(((((.............(((..(((((..(((....((((.((((.......))))))))..))))))))....)))((((((((((...)))).)))))))))))... (-31.07 = -30.00 + -1.07)

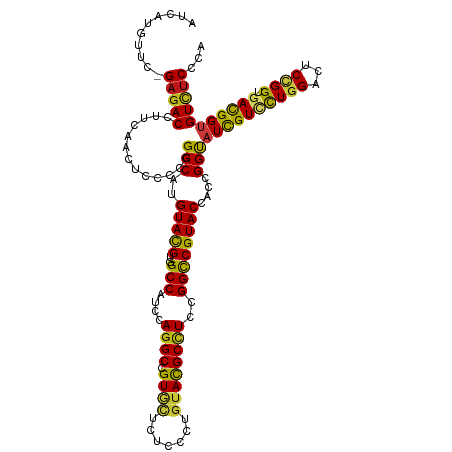
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:43:24 2011