
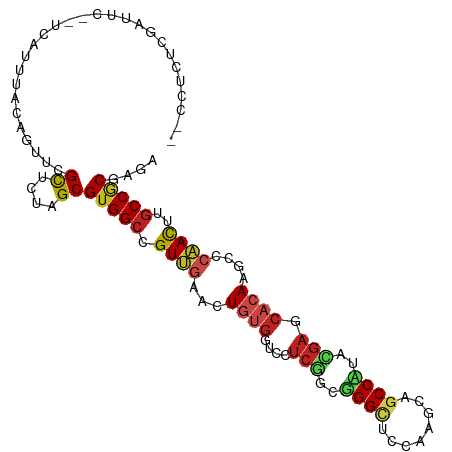
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 21,818,328 – 21,818,431 |
| Length | 103 |
| Max. P | 0.917719 |

| Location | 21,818,328 – 21,818,431 |
|---|---|
| Length | 103 |
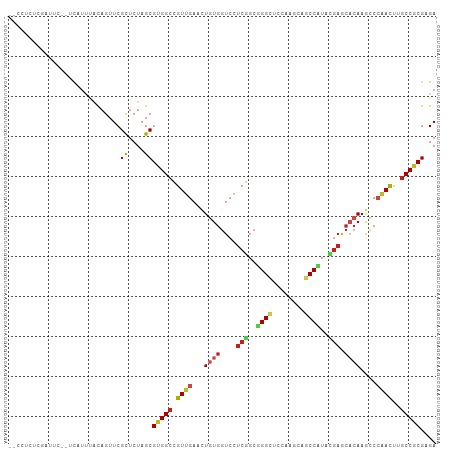
| Sequences | 12 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 73.84 |
| Shannon entropy | 0.56153 |
| G+C content | 0.57545 |
| Mean single sequence MFE | -34.23 |
| Consensus MFE | -20.58 |
| Energy contribution | -20.49 |
| Covariance contribution | -0.09 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.19 |
| Structure conservation index | 0.60 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.26 |
| SVM RNA-class probability | 0.917719 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
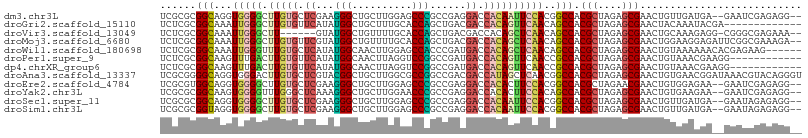
>dm3.chr3L 21818328 103 + 24543557 --CCUCUCGAUUC--UCAUCAACAGUUCGCUCUAGCGUGGCCGUGGAAUUGUGGUCCUCGGCGGGCUCCAAGCAGCCCUUCGAGCACAAGCCCCACCUGCCGCGCGA --...........--...................(((((((.((((..(((((...(((((.(((((......))))).))))))))))...))))..))))))).. ( -40.00, z-score = -1.94, R) >droGri2.scaffold_15110 1602410 94 - 24565398 -------------UCGUAUUUGUAGUUCGCUCUAGCGUGGCUGUUGAACUGUGGUCGUCAGCUGGUGCAAAGCAGCCAUAUGAACACAAGCCCAAUUUGCCGCGAGA -------------............(((((....))(((((.((((..(((((.((((..((((.(....).))))...)))).))).))..))))..)))))))). ( -26.70, z-score = 0.30, R) >droVir3.scaffold_13049 12851571 98 - 25233164 --UUUCUCGCCCG-CCUCUUUGCAGUUCGCUCUAGCGUGGCUGUUGAGCUGUGGUCGUCAGCUGGUGCAAAACAGCCAUAC------AAGCCCAAUUUGCCGCGAGA --..(((((((((-(..(((.(((((((((....))).)))))).)))..))))..((..((((........))))...))------..............)))))) ( -32.40, z-score = -0.63, R) >droMoj3.scaffold_6680 15089544 105 + 24764193 --UCUUUCGCCGAAUCUCCUUCCAGUUCGCUCUAGCGUGGCUGUUGAGCUGUGGUCGUCAGCUGGUGCAAAACAGCCAUACGAACACAAGCCCAAUUUGCCGCGAGA --............((((..........((....))(((((.((((.((((((.((((..((((........))))...)))).))).))).))))..))))))))) ( -34.80, z-score = -1.53, R) >droWil1.scaffold_180698 4938791 101 - 11422946 ------CUUCUCGUGUUUUUUACAGUUCGCUCUAGCGUGGCUGUUGAGCUGUGGUCAUCGGGUGGCUCCAAGUUGCCAUAUGAGCACAAACCCAAUUUGCCGCGAGA ------..((((((((((...(((((((((....))).)))))).))))((((.((((..((..((.....))..))..)))).)))).............)))))) ( -36.80, z-score = -2.49, R) >droPer1.super_9 36520 95 - 3637205 ------------CCUUCGUUUACAGUUCGCUCUAGCGUGGCGGUUGAACUGUGGUCAUCGGCCGGACCUAAGUUGCCAUAUGAACACAAGUCAAACUUGCCGCGAGA ------------.............(((((....))((((((.((((..((((.((((.(((..(((....))))))..)))).))))..))))...))))))))). ( -27.60, z-score = -0.32, R) >dp4.chrXR_group6 8024002 95 - 13314419 ------------CCUUCGUUUACAGUUCGCUCUAGCGUGGCGGUUGAACUGUGGUCAUCGGCCGGACCUAAGUUGCCAUAUGAACACAAGUCAAACUUGCCGCGAGA ------------.............(((((....))((((((.((((..((((.((((.(((..(((....))))))..)))).))))..))))...))))))))). ( -27.60, z-score = -0.32, R) >droAna3.scaffold_13337 20922349 107 + 23293914 ACCCUGUACGUUUAUCCGUUCACAGUUCGCUCUAGCGUGGCCGUUGAGCUAUGGUCGUCGGCCGGCGCCAAGCAGCCGUACGAGCACAAGUCCCACCUGCCCCGCGA ...(((((((......)))..)))).........(((.(((.((.(..((.((.((((((((..((.....)).)))).)))).))..))..).))..))).))).. ( -33.20, z-score = 0.13, R) >droEre2.scaffold_4784 21490004 103 + 25762168 --CCUCUCGAUUC--UUCUCCACAGUUCGUUCUAGCGUGGCCGUGGAAGUGUGGUCCUCGGCGGGCUCCAAGCAGCCCUUCGAGCACAAGCCCCACCUGCCACGCGA --...........--...................(((((((.((((...((((...(((((.(((((......))))).)))))))))....))))..))))))).. ( -39.10, z-score = -1.89, R) >droYak2.chr3L 22074724 103 + 24197627 --CCUCUCGAUUC--UUCUUCACAGUUCGCUCUAGCGUGGCUGUGGAAGUGUGGUCCUCGGCGGGUUCCAAGCAGCCCUUUGAGCCCAAACCCCACUUGCCGCGCGA --...........--...................(((((((.((((..((.(((..(((((.(((((......))))).))))).))).)).))))..))))))).. ( -36.10, z-score = -1.20, R) >droSec1.super_11 1739209 103 + 2888827 --CCUCUCUAUUC--UCAUCAACAGUUCGCUCUAGCGUGGCCGUGGAAUUGUGGUCCUCGGCGGGCUCCAAGCAGCCCUUCGAGCACAAGCCCCACCUGCCGCGCGA --...........--...................(((((((.((((..(((((...(((((.(((((......))))).))))))))))...))))..))))))).. ( -40.00, z-score = -2.47, R) >droSim1.chr3L 21162505 103 + 22553184 --CCUCUCUAUUC--UCAUCAACAGUUCGCUCUAGCGUGGCCGUGGAAUUGUGGUCCUCGGCGGGCUCCAAGCAGCCCUUCGAGCACAAGCCCCACCUACCGCGCGA --...........--...................((((((..((((..(((((...(((((.(((((......))))).))))))))))...))))...)))))).. ( -36.50, z-score = -1.95, R) >consensus __CCUCUCGAUUC__UCAUUUACAGUUCGCUCUAGCGUGGCCGUUGAACUGUGGUCCUCGGCGGGCUCCAAGCAGCCAUACGAGCACAAGCCCAACUUGCCGCGAGA ............................((....))(((((.((((...((((....(((..((((........))))..))).))))....))))..))))).... (-20.58 = -20.49 + -0.09)

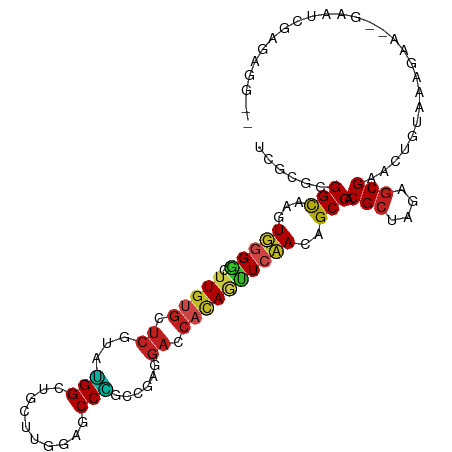
| Location | 21,818,328 – 21,818,431 |
|---|---|
| Length | 103 |
| Sequences | 12 |
| Columns | 107 |
| Reading direction | reverse |
| Mean pairwise identity | 73.84 |
| Shannon entropy | 0.56153 |
| G+C content | 0.57545 |
| Mean single sequence MFE | -35.81 |
| Consensus MFE | -17.23 |
| Energy contribution | -16.70 |
| Covariance contribution | -0.53 |
| Combinations/Pair | 1.43 |
| Mean z-score | -1.10 |
| Structure conservation index | 0.48 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.548402 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 21818328 103 - 24543557 UCGCGCGGCAGGUGGGGCUUGUGCUCGAAGGGCUGCUUGGAGCCCGCCGAGGACCACAAUUCCACGGCCACGCUAGAGCGAACUGUUGAUGA--GAAUCGAGAGG-- ..(((.(((..((((((.(((((((((..(((((......)))))..))))...))))))))))).))).))).........((.(((((..--..))))).)).-- ( -44.80, z-score = -1.88, R) >droGri2.scaffold_15110 1602410 94 + 24565398 UCUCGCGGCAAAUUGGGCUUGUGUUCAUAUGGCUGCUUUGCACCAGCUGACGACCACAGUUCAACAGCCACGCUAGAGCGAACUACAAAUACGA------------- ..(((.(((...(((((((.(((.((...((((((........))))))..)).))))))))))..))).(((....)))...........)))------------- ( -24.90, z-score = -0.29, R) >droVir3.scaffold_13049 12851571 98 + 25233164 UCUCGCGGCAAAUUGGGCUU------GUAUGGCUGUUUUGCACCAGCUGACGACCACAGCUCAACAGCCACGCUAGAGCGAACUGCAAAGAGG-CGGGCGAGAAA-- (((((((((...(((((((.------...(((.((((..((....)).)))).))).)))))))..))).(((....)))..((((......)-)))))))))..-- ( -37.50, z-score = -1.96, R) >droMoj3.scaffold_6680 15089544 105 - 24764193 UCUCGCGGCAAAUUGGGCUUGUGUUCGUAUGGCUGUUUUGCACCAGCUGACGACCACAGCUCAACAGCCACGCUAGAGCGAACUGGAAGGAGAUUCGGCGAAAGA-- ..(((((((...(((((((.(((.((((.((((((........)))))))))).))))))))))..))).(((....))).....(((.....))).))))....-- ( -37.90, z-score = -1.54, R) >droWil1.scaffold_180698 4938791 101 + 11422946 UCUCGCGGCAAAUUGGGUUUGUGCUCAUAUGGCAACUUGGAGCCACCCGAUGACCACAGCUCAACAGCCACGCUAGAGCGAACUGUAAAAAACACGAGAAG------ (((((.(((...(((((((.(((.((((.((((........))))....)))).))))))))))..))).(((....)))..............)))))..------ ( -33.00, z-score = -2.72, R) >droPer1.super_9 36520 95 + 3637205 UCUCGCGGCAAGUUUGACUUGUGUUCAUAUGGCAACUUAGGUCCGGCCGAUGACCACAGUUCAACCGCCACGCUAGAGCGAACUGUAAACGAAGG------------ ..(((.(((..(((.((((.(((.((((.((((.((....))...)))))))).))))))).))).))).(((....))).........)))...------------ ( -27.20, z-score = -0.63, R) >dp4.chrXR_group6 8024002 95 + 13314419 UCUCGCGGCAAGUUUGACUUGUGUUCAUAUGGCAACUUAGGUCCGGCCGAUGACCACAGUUCAACCGCCACGCUAGAGCGAACUGUAAACGAAGG------------ ..(((.(((..(((.((((.(((.((((.((((.((....))...)))))))).))))))).))).))).(((....))).........)))...------------ ( -27.20, z-score = -0.63, R) >droAna3.scaffold_13337 20922349 107 - 23293914 UCGCGGGGCAGGUGGGACUUGUGCUCGUACGGCUGCUUGGCGCCGGCCGACGACCAUAGCUCAACGGCCACGCUAGAGCGAACUGUGAACGGAUAAACGUACAGGGU (((((((((...((((.((.(((.((((.(((((((.....).)))))))))).)))))))))...))).(((....)))..))))))(((......)))....... ( -41.40, z-score = -0.51, R) >droEre2.scaffold_4784 21490004 103 - 25762168 UCGCGUGGCAGGUGGGGCUUGUGCUCGAAGGGCUGCUUGGAGCCCGCCGAGGACCACACUUCCACGGCCACGCUAGAACGAACUGUGGAGAA--GAAUCGAGAGG-- ..(((((((..((((((..((((((((..(((((......)))))..))))...)))).)))))).))))))).........((.(.((...--...)).).)).-- ( -43.60, z-score = -1.65, R) >droYak2.chr3L 22074724 103 - 24197627 UCGCGCGGCAAGUGGGGUUUGGGCUCAAAGGGCUGCUUGGAACCCGCCGAGGACCACACUUCCACAGCCACGCUAGAGCGAACUGUGAAGAA--GAAUCGAGAGG-- ((((..(((..(((((((.((((((.....)))..(((((......)))))..))).)).))))).))).(((....)))....))))....--...........-- ( -32.30, z-score = 0.65, R) >droSec1.super_11 1739209 103 - 2888827 UCGCGCGGCAGGUGGGGCUUGUGCUCGAAGGGCUGCUUGGAGCCCGCCGAGGACCACAAUUCCACGGCCACGCUAGAGCGAACUGUUGAUGA--GAAUAGAGAGG-- ..(((.(((..((((((.(((((((((..(((((......)))))..))))...))))))))))).))).))).........(((((.....--.))))).....-- ( -40.80, z-score = -1.12, R) >droSim1.chr3L 21162505 103 - 22553184 UCGCGCGGUAGGUGGGGCUUGUGCUCGAAGGGCUGCUUGGAGCCCGCCGAGGACCACAAUUCCACGGCCACGCUAGAGCGAACUGUUGAUGA--GAAUAGAGAGG-- ..(((.(((..((((((.(((((((((..(((((......)))))..))))...))))))))))).))).))).........(((((.....--.))))).....-- ( -39.10, z-score = -0.89, R) >consensus UCGCGCGGCAAGUGGGGCUUGUGCUCGUAUGGCUGCUUGGAGCCCGCCGAGGACCACAGUUCAACAGCCACGCUAGAGCGAACUGUAAAGAA__GAAUCGAGAGG__ ......(((...(((((.(((((.((...(((..........)))......)).))))))))))..))).(((....)))........................... (-17.23 = -16.70 + -0.53)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:43:03 2011