
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 21,789,437 – 21,789,554 |
| Length | 117 |
| Max. P | 0.500000 |

| Location | 21,789,437 – 21,789,554 |
|---|---|
| Length | 117 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.06 |
| Shannon entropy | 0.18766 |
| G+C content | 0.38717 |
| Mean single sequence MFE | -26.29 |
| Consensus MFE | -22.73 |
| Energy contribution | -22.30 |
| Covariance contribution | -0.43 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.25 |
| Structure conservation index | 0.86 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
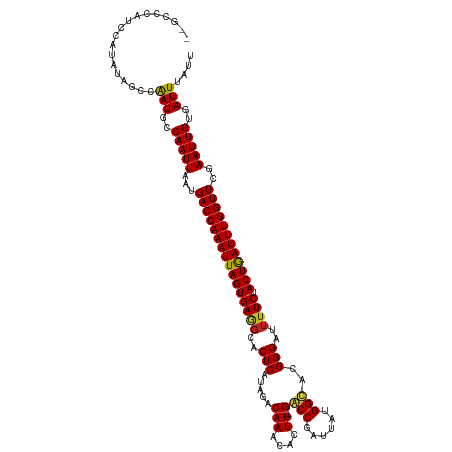
>dm3.chr3L 21789437 117 + 24543557 --GCCCAUCCACACAGCCAAUGCCAAUUAAUGACCAAAUUAGUGAGGCACUCAUAGACAAACAGUU-GGCCUAUUAUGGCACGGGAUUUUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU --................(((..(((((...((((((((((((((((..(((...(((.....)))-.(((......)))..)))..)))).))))))))))))..)))))..))).... ( -27.20, z-score = -0.76, R) >droSim1.chr3L 21132650 117 + 22553184 --GCCCAUCCAUACAGCCAAUGCCAAUUAAUGACCAAAUUAGUGAGGCACUCAUAGACAAACACUU-GGCCUAUUAUGGCACGGGAUUUUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU --................(((..(((((...((((((((((((((((..(((.....(((....))-)(((......)))..)))..)))).))))))))))))..)))))..))).... ( -27.00, z-score = -1.03, R) >droSec1.super_11 1706985 117 + 2888827 --GCCCAUCCAUACAGCCAAUGCCAAUUAAUGACCAAAUUAGUGAGGCACUCAUAGACAAACACUU-GGCCUAUUAUGGCACGGGAUUUUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU --................(((..(((((...((((((((((((((((..(((.....(((....))-)(((......)))..)))..)))).))))))))))))..)))))..))).... ( -27.00, z-score = -1.03, R) >droYak2.chr3L 5484848 117 - 24197627 --GCCCAUCCAUACAGCCAAUGCCAAUUAAUGACCAAAUUAGUGAGGCACUCAUAGACAAACACUU-GGCCUAUUAUGGCACGGGAUUUUCUACUAAUUUGGUUCGAAUUGUGAUUUAUU --................(((..(((((...((((((((((((((((..(((.....(((....))-)(((......)))..)))..)))).))))))))))))..)))))..))).... ( -26.90, z-score = -1.29, R) >droEre2.scaffold_4784 21461460 117 + 25762168 --GCCCAUCCAUACAGCCAAUGCCAAUUAAUGACCAAAUUAGUGAAGCACUCAUAGACAAACACUU-GGCCUAUUAUGGCACGGGAUUUUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU --................(((..(((((...((((((((((((((((..(((.....(((....))-)(((......)))..)))..)))).))))))))))))..)))))..))).... ( -27.40, z-score = -1.62, R) >droAna3.scaffold_13337 4101405 117 + 23293914 --GUCCAUCCAUAUAGCCGAUGCCAAUUAAUGACCAAAUUAGUGAGGCACUCAUAGACAAACACUU-GACCGAUUAUGGCUCGGGAUUUUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU --...((((.........)))).(((((...(((((((((((((((...)))...........(((-((((......)).))))).......))))))))))))..)))))......... ( -24.20, z-score = -0.28, R) >dp4.chrXR_group6 8897605 117 - 13314419 --GCCCAUCCAUAUAGCCGAUGCCAAUUAAUGACCAAAUUAGUGAAGCACUCAUAGACAAACACUU-GACCGAUUAUGGUACGGGAUUUUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU --...((((.........)))).(((((...((((((((((((((((..(((.....(((....))-)(((......)))..)))..)))).))))))))))))..)))))......... ( -24.60, z-score = -1.17, R) >droPer1.super_9 925898 117 - 3637205 --GCCCAUCCAUAUAGCCGAUGCCAAUUAAUGACCAAAUUAGUGAAGCACUCAUAGACAAACACUU-GACCGAUUAUGGUACGGGAUUUUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU --...((((.........)))).(((((...((((((((((((((((..(((.....(((....))-)(((......)))..)))..)))).))))))))))))..)))))......... ( -24.60, z-score = -1.17, R) >droWil1.scaffold_180698 5238610 111 + 11422946 ---------CAUAUAGGUAAUGCCAAUUAAUGACCAAAUUAGUGAAGCACUCAUAGACAAACACUUUGACCGCAUAUGGUACGGGUUUAUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU ---------......(((((..((((((...((((((((((((((.....)))((((.((((.((...((((....))))..)))))).)))))))))))))))..))))).)..))))) ( -25.50, z-score = -1.58, R) >droMoj3.scaffold_6680 18751753 118 + 24764193 ACAUACUCACAUAUAAGUAAUGCCAAUUAAUGACCAAAUUAGUGAGCCACUCAUGGACAA-CACUU-GGCCGCUUAUGGCACGGGAUUUUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU ...............(((((..((((((...(((((((((((((.((((....))).)..-..(((-(((((....)))).))))......)))))))))))))..))))).)..))))) ( -28.70, z-score = -2.00, R) >droGri2.scaffold_15110 14974391 116 - 24565398 --GCACCUACAUAUAAGUAAUGCCAAUUAAUGACCAAAUUAGUGAGGCACUCAUGGACAA-CACUU-GACCGCUUAUGGUACGGGAUUUUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU --(((..(((......))).)))(((((...((((((((((((((((......((.....-))(((-(((((....)))).))))..)))).))))))))))))..)))))......... ( -26.60, z-score = -1.50, R) >droVir3.scaffold_13049 24129017 113 + 25233164 -----CAUACAUAUAAGUAAUGCCAAUUAAUGACCAAAUUAGUGAGGCACUCAUGGACAA-CACUU-GACCGCUUAUGGCACGGGAUUUUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU -----..........(((((..((((((...((((((((((((((((..(((..((.((.-....)-).))((.....))..)))..)))).))))))))))))..))))).)..))))) ( -25.80, z-score = -1.56, R) >consensus __GCCCAUCCAUAUAGCCAAUGCCAAUUAAUGACCAAAUUAGUGAGGCACUCAUAGACAAACACUU_GACCGAUUAUGGCACGGGAUUUUCUACUGAUUUGGUUCGAAUUGUGAUUUAUU ..................(((..(((((...((((((((((((((((..(((...(.....)......(((......)))..)))..)))).))))))))))))..)))))..))).... (-22.73 = -22.30 + -0.43)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:43:00 2011