
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 20,628,048 – 20,628,138 |
| Length | 90 |
| Max. P | 0.824274 |

| Location | 20,628,048 – 20,628,138 |
|---|---|
| Length | 90 |
| Sequences | 10 |
| Columns | 97 |
| Reading direction | forward |
| Mean pairwise identity | 82.30 |
| Shannon entropy | 0.35936 |
| G+C content | 0.40020 |
| Mean single sequence MFE | -25.00 |
| Consensus MFE | -13.10 |
| Energy contribution | -13.80 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.52 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.81 |
| SVM RNA-class probability | 0.824274 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
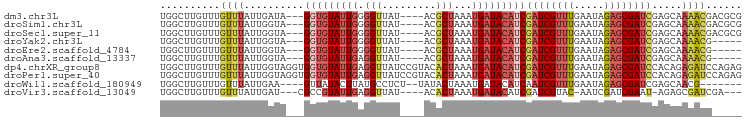
>dm3.chr3L 20628048 90 + 24543557 UGGCUUGUUUGUUUAUUGAUA---GGUGUAUUGGGGUUAU----ACGCUAAAUGAUACAUCGAUCGUUUGAAUAGAGCGAUCGAGCAAAACGACGCG ..((((((((((((.......---(((((((((.(((...----..)))...)))))))))((((((((.....)))))))))))).)))))).)). ( -28.30, z-score = -3.14, R) >droSim1.chr3L 19980548 90 + 22553184 UGGCUUGUUUGUUUAUUGGUA---GGUGUAUUGGGGUUAU----ACGCUAAAUGAUACAUCGAUCGUUUGAAUAGAGCGAUCGAGCAAAACGACGCG ..((((((((((((.......---(((((((((.(((...----..)))...)))))))))((((((((.....)))))))))))).)))))).)). ( -28.30, z-score = -2.99, R) >droSec1.super_11 517412 90 + 2888827 UGGCUUGUUUGUUUAUUGGUA---GGUGUAUUGGGGUUAU----ACGCUAAAUGAUACAUCGAUCGUUUGAAUAGAGCGAUCGAGCAAAACGACGCG ..((((((((((((.......---(((((((((.(((...----..)))...)))))))))((((((((.....)))))))))))).)))))).)). ( -28.30, z-score = -2.99, R) >droYak2.chr3L 4275050 85 - 24197627 UGGCUUGUUUGUUUAUUGGUA---GGUGUAUUGGGGUUAU----ACGCUAAAUGAUACAUCGAUCGUUUGAAUAGAGCGAUCGAGCAAAACG----- .(..((((((...........---(((((((((.(((...----..)))...)))))))))((((((((.....))))))))))))))..).----- ( -24.60, z-score = -2.80, R) >droEre2.scaffold_4784 20321824 85 + 25762168 UGGCUUGUUUGUUUAUUGGUA---GGUGUAUUGGGGUUAU----ACGCUAAAUGAUACAUCGAUCGUUUGAAUAGAGCGAUCGAGCAAAACG----- .(..((((((...........---(((((((((.(((...----..)))...)))))))))((((((((.....))))))))))))))..).----- ( -24.60, z-score = -2.80, R) >droAna3.scaffold_13337 10979625 85 + 23293914 UGGCUUGUUUGUUUAUUGGUA---GGUGUAUUGAGGUUAU----ACGCUAAAUGAUACAUCGAUCGUUUGAAUAGAGCGAUCGAGCAAAACG----- .(..((((.((((((((....---(((((((.......))----))))).))))).)))((((((((((.....))))))))))))))..).----- ( -23.70, z-score = -2.42, R) >dp4.chrXR_group8 8050750 97 + 9212921 UGGCUUGUUUGUUUAUUGGUAGGUGGUGUAUUGAGGUUAUCCGUACACUAAAUGAUACAUCGAUCGUUUGAAUAGAGCGAUCCACAGAGAUCCAGAG (((..(.(((((......(((..((((((((.((.....)).)))))))).....)))...((((((((.....)))))))).))))).).)))... ( -25.90, z-score = -1.51, R) >droPer1.super_40 243852 97 + 810552 UGGCUUGUUUGUUUAUUGGUAGGUGGUGUAUUGAGGUUAUCCGUACACUAAAUGAUACAUCGAUCGUUUGAAUAGAGCGAUCCACAGAGAUCCAGAG (((..(.(((((......(((..((((((((.((.....)).)))))))).....)))...((((((((.....)))))))).))))).).)))... ( -25.90, z-score = -1.51, R) >droWil1.scaffold_180949 2312907 84 + 6375548 UGGCUUGUUUGUUUAUUGAA----GUUAUACUUAUGCCUCU--UAUACUAAAUGAUACAUCAAUCGUUUGAAUAGAGCGAUCGAGCAACG------- ..(((((.((((((....((----(.....)))........--.....((((((((......))))))))....)))))).)))))....------- ( -16.40, z-score = -1.02, R) >droVir3.scaffold_13049 6517933 85 - 25233164 UGGCUUGUUUGUUUAUUGAU---CGCCGUAUUGAGGUUAU----ACACUAAAUGAUACAUCGAUCGUAC-AAUCGAUCGAAU-AGAGCGAUCGA--- ...............(((((---(((((((((.((.....----...))....))))).(((((((...-...)))))))..-.).))))))))--- ( -24.00, z-score = -1.75, R) >consensus UGGCUUGUUUGUUUAUUGGUA___GGUGUAUUGAGGUUAU____ACGCUAAAUGAUACAUCGAUCGUUUGAAUAGAGCGAUCGAGCAAAACG_____ ..........((((..........(((((((((.(((.........)))...)))))))))((((((((.....)))))))).....))))...... (-13.10 = -13.80 + 0.70)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:40:25 2011