| Sequence ID | dm3.chr3L |
|---|---|
| Location | 19,558,233 – 19,558,321 |
| Length | 88 |
| Max. P | 0.998537 |

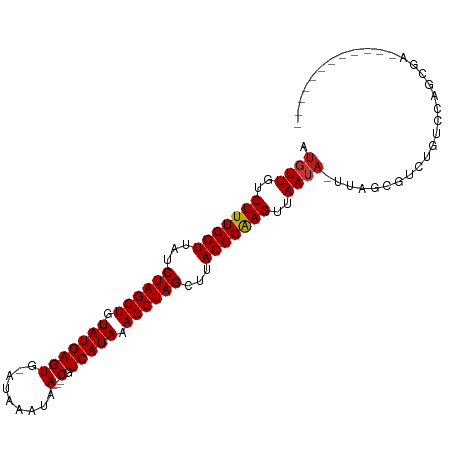
| Location | 19,558,233 – 19,558,321 |
|---|---|
| Length | 88 |
| Sequences | 15 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 86.26 |
| Shannon entropy | 0.29235 |
| G+C content | 0.36524 |
| Mean single sequence MFE | -26.51 |
| Consensus MFE | -20.11 |
| Energy contribution | -20.20 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.05 |
| Mean z-score | -3.59 |
| Structure conservation index | 0.76 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.39 |
| SVM RNA-class probability | 0.998537 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 19558233 88 + 24543557 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUAAAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGCGUCUGCCCAGCGA------------ ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-)..((.........))..------------ ( -26.70, z-score = -3.35, R) >droSim1.chr3L 18890429 88 + 22553184 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUAAAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGCGUCUGACCAGCGA------------ ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-)..((.........))..------------ ( -26.70, z-score = -3.33, R) >droSec1.super_29 261386 88 + 700605 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUAAAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGCGUCUGACCAGCGA------------ ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-)..((.........))..------------ ( -26.70, z-score = -3.33, R) >droYak2.chr3L 3208136 88 - 24197627 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUAAAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGCGUCUGACCAGCGA------------ ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-)..((.........))..------------ ( -26.70, z-score = -3.33, R) >droEre2.scaffold_4784 19355679 88 + 25762168 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUAAAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGCGUCUGACCAGCGA------------ ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-)..((.........))..------------ ( -26.70, z-score = -3.33, R) >droAna3.scaffold_13337 5961401 88 - 23293914 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUAAAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGCGUCUGUCCAGCGA------------ ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-)..((.........))..------------ ( -26.70, z-score = -3.51, R) >dp4.chrXR_group8 7634829 88 + 9212921 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUAAAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGCGUCUGUCCAGCGA------------ ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-)..((.........))..------------ ( -26.70, z-score = -3.51, R) >droPer1.super_48 412969 88 + 581423 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUAAAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGCGUCUGUCCAGCGA------------ ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-)..((.........))..------------ ( -26.70, z-score = -3.51, R) >droWil1.scaffold_180727 842235 100 + 2741493 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUACAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGCGUCUGUUCAUCAGUCAUGUGAACGA ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-).......((((((.((....)))))))). ( -31.00, z-score = -3.42, R) >droVir3.scaffold_13049 13137025 88 + 25233164 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUAAAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGUGUCUGCUCAACGU------------ ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-).................------------ ( -25.60, z-score = -3.60, R) >droMoj3.scaffold_6680 10537605 88 - 24764193 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUAAAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGUGUCUGCUCAGCGU------------ ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-).................------------ ( -25.60, z-score = -3.09, R) >droGri2.scaffold_15110 23882751 88 + 24565398 AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG-AUAAAUAA---CGUCAUAAAGCUAGCUUACCGAAGUUAAUA-UUAGUGUCUCUUCAACGU------------ ((((((.(((((((...((((((.(((((((.-.......)---).))))).))))))...))))))).)))))-).................------------ ( -25.60, z-score = -4.13, R) >anoGam1.chr2L 34274958 90 - 48795086 AUGUUCUCUUUGGUUAUCUAGCUGUAUGAGUGUAUUUUAAA---CGUCAUAAAGCUAGCAUACCGAAGUUAAUAAUUGACUGAUACUCAUCGA------------ .((((..(((((((...((((((.(((((.(((.......)---))))))).))))))...)))))))..)))).((((.((.....))))))------------ ( -25.10, z-score = -3.51, R) >apiMel3.Group1 7627111 79 - 25854376 ACAUUUUCUUUGGUUAUCUAGCUGUAUGAGUA-UUAUUCGA---CAUCAUAAAGCUAGGUUACCGGAGUUAAGC-UCC-UCGCCA-------------------- ...........(((.((((((((.(((((((.-.......)---).))))).)))))))).)))((((.....)-)))-......-------------------- ( -25.90, z-score = -4.43, R) >triCas2.chrUn_11 115138 89 + 1365317 AUAUUUUCUUUGGUUAUCUAGCUGUAUGAGUA-UUGUUUAAUAACAUCAUAAAGCUAGGUUACCGGAGUUAAAA-CCAGUUUCCAUACAUC-------------- ...((..(((((((.((((((((.(((((.((-((....))))...))))).)))))))).)))))))..))..-................-------------- ( -25.30, z-score = -4.53, R) >consensus AUGUUGUCUUUGGUUAUCUAGCUGUAUGAGUG_AUAAAUAA___CGUCAUAAAGCUAGCUUACCGAAGUUAAUA_UUAGCGUCUGUCCAGCGA____________ .((((..(((((((...((((((.(((((.................))))).))))))...)))))))..))))............................... (-20.11 = -20.20 + 0.08)

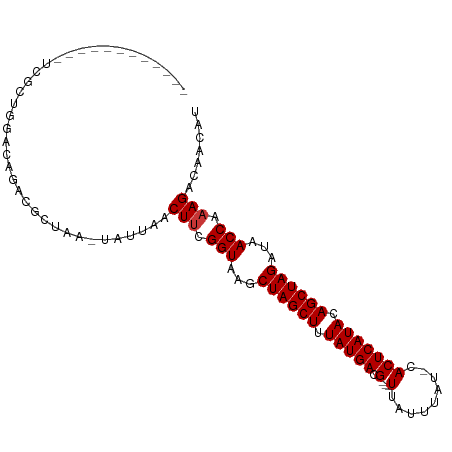
| Location | 19,558,233 – 19,558,321 |
|---|---|
| Length | 88 |
| Sequences | 15 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 86.26 |
| Shannon entropy | 0.29235 |
| G+C content | 0.36524 |
| Mean single sequence MFE | -20.00 |
| Consensus MFE | -16.34 |
| Energy contribution | -16.48 |
| Covariance contribution | 0.13 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.89 |
| Structure conservation index | 0.82 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.83 |
| SVM RNA-class probability | 0.995658 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 19558233 88 - 24543557 ------------UCGCUGGGCAGACGCUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUUUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ------------..(.(((((....)))..-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).))).)).).. ( -20.80, z-score = -2.78, R) >droSim1.chr3L 18890429 88 - 22553184 ------------UCGCUGGUCAGACGCUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUUUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ------------..((.(......)))...-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).)))....... ( -19.10, z-score = -2.36, R) >droSec1.super_29 261386 88 - 700605 ------------UCGCUGGUCAGACGCUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUUUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ------------..((.(......)))...-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).)))....... ( -19.10, z-score = -2.36, R) >droYak2.chr3L 3208136 88 + 24197627 ------------UCGCUGGUCAGACGCUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUUUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ------------..((.(......)))...-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).)))....... ( -19.10, z-score = -2.36, R) >droEre2.scaffold_4784 19355679 88 - 25762168 ------------UCGCUGGUCAGACGCUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUUUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ------------..((.(......)))...-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).)))....... ( -19.10, z-score = -2.36, R) >droAna3.scaffold_13337 5961401 88 + 23293914 ------------UCGCUGGACAGACGCUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUUUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ------------..((.(......)))...-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).)))....... ( -19.10, z-score = -2.69, R) >dp4.chrXR_group8 7634829 88 - 9212921 ------------UCGCUGGACAGACGCUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUUUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ------------..((.(......)))...-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).)))....... ( -19.10, z-score = -2.69, R) >droPer1.super_48 412969 88 - 581423 ------------UCGCUGGACAGACGCUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUUUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ------------..((.(......)))...-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).)))....... ( -19.10, z-score = -2.69, R) >droWil1.scaffold_180727 842235 100 - 2741493 UCGUUCACAUGACUGAUGAACAGACGCUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUGUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ..(((((((....)).))))).........-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).)))....... ( -23.40, z-score = -2.81, R) >droVir3.scaffold_13049 13137025 88 - 25233164 ------------ACGUUGAGCAGACACUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUUUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ------------..((((((......))..-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).))).)))).. ( -21.20, z-score = -4.08, R) >droMoj3.scaffold_6680 10537605 88 + 24764193 ------------ACGCUGAGCAGACACUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUUUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ------------..................-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).)))....... ( -18.60, z-score = -2.96, R) >droGri2.scaffold_15110 23882751 88 - 24565398 ------------ACGUUGAAGAGACACUAA-UAUUAACUUCGGUAAGCUAGCUUUAUGACG---UUAUUUAU-CACUCAUACAGCUAGAUAACCAAAGACAACAU ------------..((((.((.....))..-......(((.(((...((((((.(((((.(---(.......-.))))))).))))))...))).))).)))).. ( -20.90, z-score = -4.13, R) >anoGam1.chr2L 34274958 90 + 48795086 ------------UCGAUGAGUAUCAGUCAAUUAUUAACUUCGGUAUGCUAGCUUUAUGACG---UUUAAAAUACACUCAUACAGCUAGAUAACCAAAGAGAACAU ------------..(((........))).........(((.(((...((((((.(((((.(---(.........))))))).))))))...))).)))....... ( -18.80, z-score = -2.37, R) >apiMel3.Group1 7627111 79 + 25854376 --------------------UGGCGA-GGA-GCUUAACUCCGGUAACCUAGCUUUAUGAUG---UCGAAUAA-UACUCAUACAGCUAGAUAACCAAAGAAAAUGU --------------------......-(((-(.....))))(((...((((((.(((((.(---(.......-.))))))).))))))...)))........... ( -21.10, z-score = -3.08, R) >triCas2.chrUn_11 115138 89 - 1365317 --------------GAUGUAUGGAAACUGG-UUUUAACUCCGGUAACCUAGCUUUAUGAUGUUAUUAAACAA-UACUCAUACAGCUAGAUAACCAAAGAAAAUAU --------------......(((..(((((-........)))))...((((((.(((((((((....)))).-...))))).))))))....))).......... ( -21.50, z-score = -3.63, R) >consensus ____________UCGCUGGACAGACGCUAA_UAUUAACUUCGGUAAGCUAGCUUUAUGACG___UUAUUUAU_CACUCAUACAGCUAGAUAACCAAAGACAACAU .....................................(((.(((...((((((.(((((.................))))).))))))...))).)))....... (-16.34 = -16.48 + 0.13)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:38:04 2011