| Sequence ID | dm3.chr3L |
|---|---|
| Location | 19,455,932 – 19,456,072 |
| Length | 140 |
| Max. P | 0.962252 |

| Location | 19,455,932 – 19,456,072 |
|---|---|
| Length | 140 |
| Sequences | 11 |
| Columns | 159 |
| Reading direction | reverse |
| Mean pairwise identity | 73.57 |
| Shannon entropy | 0.55370 |
| G+C content | 0.45007 |
| Mean single sequence MFE | -39.87 |
| Consensus MFE | -23.64 |
| Energy contribution | -24.68 |
| Covariance contribution | 1.04 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.59 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.70 |
| SVM RNA-class probability | 0.962252 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 19455932 140 - 24543557 --------CCAUGUCGACGUUGGUU--GAUUUUCAACAGCAGCGCAAACGCAGCAAAGUUUAUCCUCAAUUAAACUUUUUGUUGUUGCGUAAAAUUGCGCUUGAAGUUCG-CCCCGCCAUCUGUCUCUU-UGCCU-CUGAGCUUUGUACGCGC------ --------....((((((....)))--)))........((((((((((((((((((((((((........)))))).....)))))))))....))))))).((((((((-....((............-.))..-.))))))))....))..------ ( -36.52, z-score = -0.99, R) >droSim1.chr3L 18786983 140 - 22553184 --------CCAUGUCGACGUUGGUU--GAUUUUCAACAGCAGCGCAAACGCAGCAAAGUUUAUCCUCAAUUAAACUUUUUGUUGUUGCGUAAAAUUGCGCUUGAAGUUCG-CCCCGCCAUCUGUCUCUU-UGCCU-CUGAGCUUUGUGCGUGU------ --------.(((((.((((.(((((--((.(((((.....((((((((((((((((((((((........)))))).....)))))))))....)))))))))))).)))-....))))..))))....-.((..-....)).....))))).------ ( -38.20, z-score = -1.46, R) >droSec1.super_29 157278 148 - 700605 --------UCCUGGCGACGUUGGUU--GAUUUUCAACAGCCACGCAAACGCAGCAAAGUUUAUCCUCCAUUUAACUUUUUGUUGUUGCGUGAAAUUGCGCUUGAAGUUCGCCCCCGCCAUCUGUCUCUU-UGCCUCCUCACCUUUGGGUGUGUGCGUGU --------...(((((.....(((.--((.((((((..(((((((((..((((.((((((............))))))))))..))))))).....))..)))))).)))))..)))))..........-.(((....((((....)))).).)).... ( -37.30, z-score = -0.34, R) >droYak2.chr3L 3112137 140 + 24197627 --------CCAUGUCGACGUUGGUU--GAUUUUCAACAGCAGCGCAAACGCAGCAAAGUUUAUCCUCAAUUAAACUUUUUGUUGUUGCGUAAAAUUGCGCUUGAAGUUCG-CCCCGCCAUCUGUCUCUU-UGCCU-CUGCGCUUUGUGCGCGU------ --------.......((((.(((((--((.(((((.....((((((((((((((((((((((........)))))).....)))))))))....)))))))))))).)))-....))))..))))....-.....-..((((.....))))..------ ( -41.80, z-score = -2.16, R) >droEre2.scaffold_4784 19263151 140 - 25762168 --------CCAUGUCGACGUUGGUU--GAUUUUCAACAGCAGCGCAAACGCAGCAAAGUUUAUCCUCAAUUAAACUUUUUGUUGUUGCGUAAAAUUGCGCUUGAAGUUCG-CCCCGCCAUCUGUCUCUU-UGCCU-UUGCGCUUUGUGUGCGU------ --------.......((((.(((((--((.(((((.....((((((((((((((((((((((........)))))).....)))))))))....)))))))))))).)))-....))))..))))....-.....-..((((.....))))..------ ( -39.90, z-score = -1.80, R) >droAna3.scaffold_13337 5269495 139 + 23293914 --------CCAUGUCGACGUUGGUU--GAUUUUCAACAGCAGCGCAAACGCAGCAAAGUUUAUCCUCAAUUAAACUUUUUGUUGUUGCGUAAAAUUGCGCUUGAAGUUGG-CCUCAGUCUUUGUGUGAG-UGAAUACGUGGUUGUGCGAAU-------- --------((((((((((((((.((--(.....)))))))((((((((((((((((((((((........)))))).....)))))))))....)))))))....)))))-).(((.((.......)).-)))....))))..........-------- ( -42.90, z-score = -1.94, R) >dp4.chrXR_group8 7539561 146 - 9212921 --------CCAUGUCGACGUUGGUU--GAUUUUCAACAGCAGCGCAAACGCAGCAAAGUUUAUCCUCAAUUAAACUUUUUGUUGUUGCGGAAAAUUGCGCUUGAAGUUGGGUCCA--CAUCCGUCUAUG-UGUGCGUAUGCUCACACACAUGUGCUAGU --------.(((((.((((...((.--(((..(((((..((((((((.((((((((((((((........)))))).....)))))))).....)))))).))..)))))))).)--)...))))...(-((((.(....).))))))))))....... ( -46.50, z-score = -2.06, R) >droPer1.super_48 317562 146 - 581423 --------CCAUGUCGACGUUGGUU--GAUUUUCAACAGCAGCGCAAACGCAGCAAAGUUUAUCCUCAAUUAAACUUUUUGUUGUUGCGGAAAAUUGCGCUUGAAGUUGGGUCCA--CAUCCGUCUAUG-UGUGCGUAUGCUCACACACAUGUGCUAGU --------.(((((.((((...((.--(((..(((((..((((((((.((((((((((((((........)))))).....)))))))).....)))))).))..)))))))).)--)...))))...(-((((.(....).))))))))))....... ( -46.50, z-score = -2.06, R) >droWil1.scaffold_180727 750728 137 - 2741493 CCAUUCCGGCGUAGUUUUGUGGGGUAGAAUUUUCAACAGCAGCGCAAACGCAGCAAAGUUUAUCCUCAAUUAAACUUUUUGUUGUUGCGUAAAAUUGCGCAAAAUGUGUUUUUUGGGGGAGUGAGCAUCAUGUGUGU---------------------- .((((((.((((.(((...((..(......)..))..))).))))..(((((((((((((((........)))))).....))))))))).(((..((((.....))))..)))...)))))).((((...))))..---------------------- ( -37.60, z-score = -0.89, R) >droVir3.scaffold_13049 13036716 119 - 25233164 ---------------------AGUU--GAUUUUCAACGGCAGCGCAAACGCAGCAAAGUUUAUCCUCAAUUAAACUUUUUGUUGUUGCGUAAAAUUGCGCCAGAGUUGGGGUUC---GUUCAGUUUGUC-UGUGCCCGGGUGUGCA------------- ---------------------.(((--(.....)))).((((((((((((((((((((((((........)))))).....)))))))))....)))))).....((.((((..---...(((.....)-)).)))).))..))).------------- ( -37.10, z-score = -1.60, R) >droGri2.scaffold_14375 79 123 + 21476 --------CACUGUCGACGUUGGCU--GAUUUUGAACAGCAGCGCAAACGCAGAAAAGUUUAUCCUCAAUUAAACUUUUUGUAGUUGCGUAAAAUUGCGCCGGAGUUCAG-UU----UGUCCGUUUGAG-UGUAUGUGU-------------------- --------((((...((((..(((.--....((((((..(.(((((((((((((((((((((........)))))))))).....)))))....)))))).)..))))))-..----.)))))))..))-)).......-------------------- ( -34.20, z-score = -1.22, R) >consensus ________CCAUGUCGACGUUGGUU__GAUUUUCAACAGCAGCGCAAACGCAGCAAAGUUUAUCCUCAAUUAAACUUUUUGUUGUUGCGUAAAAUUGCGCUUGAAGUUCG_CCCCGCCAUCUGUCUCUU_UGCCU_CUGGGCUUUGUGCGUGU______ ...............((((((((.........)))))....(((((((((((((((((((((........))))))).....))))))))....))))))......................))).................................. (-23.64 = -24.68 + 1.04)


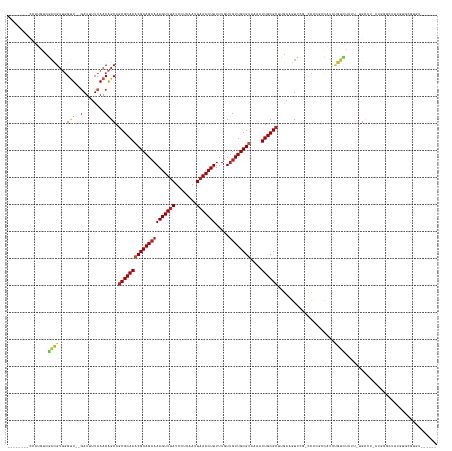
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:37:50 2011