
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 5,580,588 – 5,580,721 |
| Length | 133 |
| Max. P | 0.999928 |

| Location | 5,580,588 – 5,580,691 |
|---|---|
| Length | 103 |
| Sequences | 13 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 67.39 |
| Shannon entropy | 0.70189 |
| G+C content | 0.59902 |
| Mean single sequence MFE | -43.44 |
| Consensus MFE | -16.08 |
| Energy contribution | -16.55 |
| Covariance contribution | 0.47 |
| Combinations/Pair | 1.50 |
| Mean z-score | -2.43 |
| Structure conservation index | 0.37 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.74 |
| SVM RNA-class probability | 0.994882 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
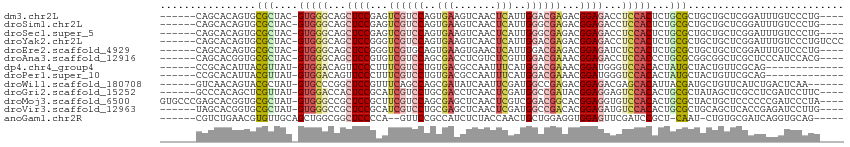
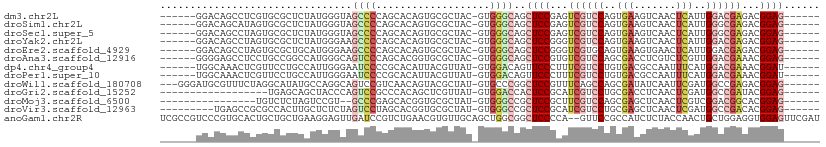
>dm3.chr2L 5580588 103 + 23011544 ----CAGGGACAAAUCCGAGCAGCAGCGCAGAGUGGAGGUCUCCGUCUCGUCCAAUGAGUUGACUUCACUGGACGACUCGGAGCUGCCCAC-GUAGCGCACUGUGCUG------ ----((((((....)))..((((..((((...((((.((.(((((..(((((((.((((.....)))).)))))))..)))))))..))))-...)))).)))).)))------ ( -46.60, z-score = -2.89, R) >droSim1.chr2L 5374998 103 + 22036055 ----CAGGGACAAAUCCGAGCAGCAGCGCAGAGUGGAGGUCUCCGUCUCGCCCAAUGAGUUGACUUCACUGGACGACUCGGAGCUGCCCAC-GUAGCGCACUGUGCUG------ ----((((((....)))..((((..((((...((((.((.(((((..(((.(((.((((.....)))).))).)))..)))))))..))))-...)))).)))).)))------ ( -41.60, z-score = -1.44, R) >droSec1.super_5 3658430 103 + 5866729 ----CAGGGACAAAUCCGAGCAGCAGCGCAGAGUGGAGGUCUCCGUCUCGCCCAAUGAGUUGACUUCACUGGACGACUCGGAGCUGCCCAC-GUAGCGCACUGUGCUG------ ----((((((....)))..((((..((((...((((.((.(((((..(((.(((.((((.....)))).))).)))..)))))))..))))-...)))).)))).)))------ ( -41.60, z-score = -1.44, R) >droYak2.chr2L 8710037 107 - 22324452 GGGACAGGGACAAAUCCGAGCAGCAGCGCAGAGUGGAGGUCUCCGUCUCGUCCAAUGAGUUGACUUCACUGGACGACCCGGAGCUGCCCAC-GUAGCGCACUGUGCUG------ ((.(((((((....)))........((((...((((.((.(((((..(((((((.((((.....)))).)))))))..)))))))..))))-...)))).)))).)).------ ( -49.40, z-score = -2.95, R) >droEre2.scaffold_4929 5666817 103 + 26641161 ----CAGGGACAAAUCCGAGCAGCAGCGCAGAGUGGAGAUCUCCGUCUCGUCCAAUGAGUUCACUUCACUGCACGACCCGGAGCUGCCCAC-GUAGCGCACUGUGCUG------ ----((((((....)))..((((..((((...((((.(..(((((..((((.((.((((.....)))).)).))))..)))))...)))))-...)))).)))).)))------ ( -38.00, z-score = -1.37, R) >droAna3.scaffold_12916 11028313 103 + 16180835 ----CGUGGAUGGGAGCGAGCCGCCGCGCAGGGUGGAGGUCUCCGUUUCGUCCAACGAGACGAGGUCGCUGGACGACACGGAGCUGCCCAC-GUAGCGCACCGUGCUG------ ----...(.(((((.(((...))))((((.(.((((.((.((((((.(((((((.(((.......))).))))))).))))))))..))))-.).)))).)))).)..------ ( -49.30, z-score = -1.47, R) >dp4.chr4_group4 3330620 94 - 6586962 -------------CUGCGAACAGUAGCAUAGUGUGGACCCAUCCGUUUCGUCCAUGAAAUUGGCGUCACAGGACGAAAGGGAACUGUCCAC-AUAACGUAAUGUGCGG------ -------------(((((....((.((...((((((((...(((.((((((((.(((........)))..)))))))).)))...))))))-))...)).)).)))))------ ( -35.80, z-score = -3.41, R) >droPer1.super_10 1155104 94 - 3432795 -------------CUGCGAACAGUAGCAUAGUGUGGACCCAUCCGUUUCGUCCAUGAAAUUGGCGUCACAGGACGAAAGGGAACUGUCCAC-AUAACGUAAUGUGCGG------ -------------(((((....((.((...((((((((...(((.((((((((.(((........)))..)))))))).)))...))))))-))...)).)).)))))------ ( -35.80, z-score = -3.41, R) >droWil1.scaffold_180708 8714433 101 + 12563649 ------UUGAGUCAGAUGAACAGCAUCGUAAUGUGCUCGUCUCCGUCUCGGCCAUCGAAUUGAUAUCGCUGGCUGAAACGGAGCCGGGCAC-AUAGCGUACUGUUGAC------ ------....((((.....((((...(((.(((((((((.((((((.(((((((.(((.......))).))))))).)))))).)))))))-)).)))..))))))))------ ( -49.90, z-score = -6.05, R) >droGri2.scaffold_15252 3073793 103 + 17193109 ----GAAGGAUCGAGGCGAGCUAUAGCGCAGUGUGGACUCCUCCGUAUCGGCCAUCGAGUUGAGGUCGCAGGACGAUGCGGAGUGGUCCAC-AUAACGAGCUGUGGGC------ ----................(((((((...((((((((..((((((((((.((..(((.......)))..)).))))))))))..))))))-)).....)))))))..------ ( -46.70, z-score = -3.37, R) >droMoj3.scaffold_6500 13911852 109 + 32352404 ----UAGGGAUCGGGGGGAGCAGUAGCGCAGUGUGGACACCUCCGUGCCGUCCGACGAGUUGAGCUCGCUGGACGAAGCGGAGCGGCCCAC-AUAGCGCACCGUGCUCGGGCAC ----.............((((((..((((.((((((.(..((((((..((((((.((((.....)))).))))))..))))))..).))))-)).))))..).)))))...... ( -54.70, z-score = -2.65, R) >droVir3.scaffold_12963 3526117 103 + 20206255 ----CAAGGAUCUCGGUGAGCUGCAGCGCAGUGUGGACAUCUCCGUGUCGGCCAUCGAGUUGAGCUCGCAGGACGAUGCGGAGCGGCCCAC-AUAGCGCACCGUGCUA------ ----...((....(((((.((....))((.((((((.(..((((((((((.((..((((.....))))..)).))))))))))..).))))-)).)).)))))..)).------ ( -46.50, z-score = -1.69, R) >anoGam1.chr2R 42439965 99 - 62725911 -----CUGCACCUGAUCGCACAG-AUUG-AGCGGAUCGAACUCCACCUCCAGCAGUUGGUAGAGAUGGCGGAAC--UGGGGAGCCGCCAGCUGCAACACGUUCAGACG------ -----.(((........)))..(-.(((-((((((......)))..........((((.(((...((((((..(--....)..)))))).)))))))..)))))).).------ ( -28.80, z-score = 0.59, R) >consensus ____CAGGGACAAAUGCGAGCAGCAGCGCAGUGUGGAGGUCUCCGUCUCGUCCAAUGAGUUGACUUCACUGGACGACACGGAGCUGCCCAC_GUAGCGCACUGUGCUG______ .........................((((...((((....((((...(((.((..(((.......)))..)).)))...))))....))))....))))............... (-16.08 = -16.55 + 0.47)


| Location | 5,580,588 – 5,580,691 |
|---|---|
| Length | 103 |
| Sequences | 13 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 67.39 |
| Shannon entropy | 0.70189 |
| G+C content | 0.59902 |
| Mean single sequence MFE | -45.37 |
| Consensus MFE | -20.74 |
| Energy contribution | -21.05 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.52 |
| Mean z-score | -3.52 |
| Structure conservation index | 0.46 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 4.95 |
| SVM RNA-class probability | 0.999928 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 5580588 103 - 23011544 ------CAGCACAGUGCGCUAC-GUGGGCAGCUCCGAGUCGUCCAGUGAAGUCAACUCAUUGGACGAGACGGAGACCUCCACUCUGCGCUGCUGCUCGGAUUUGUCCCUG---- ------.(((((((((((....-(((((...(((((..(((((((((((.......)))))))))))..)))))...)))))..))))))).)))).(((....)))...---- ( -49.10, z-score = -3.85, R) >droSim1.chr2L 5374998 103 - 22036055 ------CAGCACAGUGCGCUAC-GUGGGCAGCUCCGAGUCGUCCAGUGAAGUCAACUCAUUGGGCGAGACGGAGACCUCCACUCUGCGCUGCUGCUCGGAUUUGUCCCUG---- ------.(((((((((((....-(((((...(((((..(((((((((((.......)))))))))))..)))))...)))))..))))))).)))).(((....)))...---- ( -48.50, z-score = -3.24, R) >droSec1.super_5 3658430 103 - 5866729 ------CAGCACAGUGCGCUAC-GUGGGCAGCUCCGAGUCGUCCAGUGAAGUCAACUCAUUGGGCGAGACGGAGACCUCCACUCUGCGCUGCUGCUCGGAUUUGUCCCUG---- ------.(((((((((((....-(((((...(((((..(((((((((((.......)))))))))))..)))))...)))))..))))))).)))).(((....)))...---- ( -48.50, z-score = -3.24, R) >droYak2.chr2L 8710037 107 + 22324452 ------CAGCACAGUGCGCUAC-GUGGGCAGCUCCGGGUCGUCCAGUGAAGUCAACUCAUUGGACGAGACGGAGACCUCCACUCUGCGCUGCUGCUCGGAUUUGUCCCUGUCCC ------.(((((((((((....-(((((...(((((..(((((((((((.......)))))))))))..)))))...)))))..))))))).)))).((((........)))). ( -52.70, z-score = -4.01, R) >droEre2.scaffold_4929 5666817 103 - 26641161 ------CAGCACAGUGCGCUAC-GUGGGCAGCUCCGGGUCGUGCAGUGAAGUGAACUCAUUGGACGAGACGGAGAUCUCCACUCUGCGCUGCUGCUCGGAUUUGUCCCUG---- ------.(((((((((((....-(((((...(((((..((((.((((((.(....))))))).))))..)))))...)))))..))))))).)))).(((....)))...---- ( -47.90, z-score = -2.95, R) >droAna3.scaffold_12916 11028313 103 - 16180835 ------CAGCACGGUGCGCUAC-GUGGGCAGCUCCGUGUCGUCCAGCGACCUCGUCUCGUUGGACGAAACGGAGACCUCCACCCUGCGCGGCGGCUCGCUCCCAUCCACG---- ------.(((.((.(((((...-(((((...((((((.(((((((((((.......))))))))))).))))))...)))))...))))).)))))..............---- ( -52.80, z-score = -3.93, R) >dp4.chr4_group4 3330620 94 + 6586962 ------CCGCACAUUACGUUAU-GUGGACAGUUCCCUUUCGUCCUGUGACGCCAAUUUCAUGGACGAAACGGAUGGGUCCACACUAUGCUACUGUUCGCAG------------- ------..(((((.(((((..(-((((((...(((.((((((((.((((........)))))))))))).)))...)))))))..))).)).)))..))..------------- ( -36.10, z-score = -4.18, R) >droPer1.super_10 1155104 94 + 3432795 ------CCGCACAUUACGUUAU-GUGGACAGUUCCCUUUCGUCCUGUGACGCCAAUUUCAUGGACGAAACGGAUGGGUCCACACUAUGCUACUGUUCGCAG------------- ------..(((((.(((((..(-((((((...(((.((((((((.((((........)))))))))))).)))...)))))))..))).)).)))..))..------------- ( -36.10, z-score = -4.18, R) >droWil1.scaffold_180708 8714433 101 - 12563649 ------GUCAACAGUACGCUAU-GUGCCCGGCUCCGUUUCAGCCAGCGAUAUCAAUUCGAUGGCCGAGACGGAGACGAGCACAUUACGAUGCUGUUCAUCUGACUCAA------ ------((((((((((((..((-((((.((.(((((((((.((((.(((.......))).)))).))))))))).)).))))))..)).)))))).....))))....------ ( -49.20, z-score = -6.88, R) >droGri2.scaffold_15252 3073793 103 - 17193109 ------GCCCACAGCUCGUUAU-GUGGACCACUCCGCAUCGUCCUGCGACCUCAACUCGAUGGCCGAUACGGAGGAGUCCACACUGCGCUAUAGCUCGCCUCGAUCCUUC---- ------((....(((((((..(-((((((..(((((.((((.((..(((.......)))..)).)))).)))))..)))))))..)))....)))).))...........---- ( -37.50, z-score = -3.06, R) >droMoj3.scaffold_6500 13911852 109 - 32352404 GUGCCCGAGCACGGUGCGCUAU-GUGGGCCGCUCCGCUUCGUCCAGCGAGCUCAACUCGUCGGACGGCACGGAGGUGUCCACACUGCGCUACUGCUCCCCCCGAUCCCUA---- ......(((((.(..((((..(-(((((((.(((((..((((((.(((((.....))))).))))))..)))))).)))))))..))))..)))))).............---- ( -54.10, z-score = -3.76, R) >droVir3.scaffold_12963 3526117 103 - 20206255 ------UAGCACGGUGCGCUAU-GUGGGCCGCUCCGCAUCGUCCUGCGAGCUCAACUCGAUGGCCGACACGGAGAUGUCCACACUGCGCUGCAGCUCACCGAGAUCCUUG---- ------.....(((((.((..(-((((((..(((((..(((.((..((((.....))))..)).)))..)))))..)))))))..))((....)).))))).........---- ( -43.00, z-score = -1.98, R) >anoGam1.chr2R 42439965 99 + 62725911 ------CGUCUGAACGUGUUGCAGCUGGCGGCUCCCCA--GUUCCGCCAUCUCUACCAACUGCUGGAGGUGGAGUUCGAUCCGCU-CAAU-CUGUGCGAUCAGGUGCAG----- ------(..((((.(((((((.(((((((((..(....--)..)))))).(((((((..(....)..)))))))........)))-))))-....))).))))..)...----- ( -34.30, z-score = -0.56, R) >consensus ______CAGCACAGUGCGCUAC_GUGGGCAGCUCCGCGUCGUCCAGUGAAGUCAACUCAUUGGACGAGACGGAGACCUCCACACUGCGCUGCUGCUCGCAUUUGUCCCUG____ ................(((....(((((...((((...((((((..(((.......)))..))))))...))))...)))))...))).......................... (-20.74 = -21.05 + 0.31)

| Location | 5,580,624 – 5,580,721 |
|---|---|
| Length | 97 |
| Sequences | 13 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 62.55 |
| Shannon entropy | 0.80766 |
| G+C content | 0.60746 |
| Mean single sequence MFE | -37.68 |
| Consensus MFE | -10.07 |
| Energy contribution | -10.32 |
| Covariance contribution | 0.26 |
| Combinations/Pair | 1.53 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.27 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.60 |
| SVM RNA-class probability | 0.757977 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
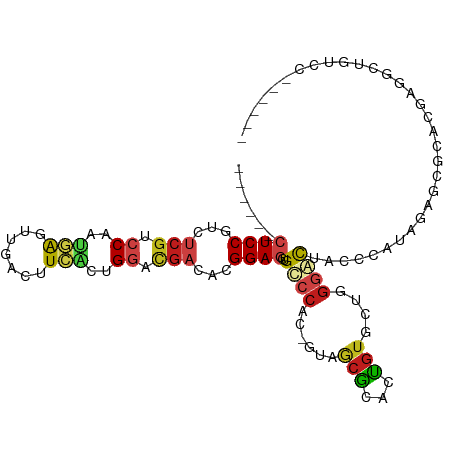
>dm3.chr2L 5580624 97 + 23011544 ------CUCCGUCUCGUCCAAUGAGUUGACUUCACUGGACGACUCGGAGCUGCCCAC-GUAGCGCACUGUGCUGGGGCUACCCAUAGAGCGCACGAGGCUGUCC------ ------(((((..(((((((.((((.....)))).)))))))..)))))..(((..(-((.((((.(((...((((....))))))).))))))).))).....------ ( -44.60, z-score = -3.26, R) >droSim1.chr2L 5375034 97 + 22036055 ------CUCCGUCUCGCCCAAUGAGUUGACUUCACUGGACGACUCGGAGCUGCCCAC-GUAGCGCACUGUGCUGGGGCUACCCAUAGAGCGCACUAUGCUGUCC------ ------.((((..(((.(((.((((.....)))).))).)))..))))(((((....-)))))(((..((((((((....))))......))))..))).....------ ( -36.20, z-score = -1.65, R) >droSec1.super_5 3658466 97 + 5866729 ------CUCCGUCUCGCCCAAUGAGUUGACUUCACUGGACGACUCGGAGCUGCCCAC-GUAGCGCACUGUGCUGGGGCUACCCAUAGAGCGCACUAGGCUGUCC------ ------(((((..(((.(((.((((.....)))).))).)))..)))))..(((...-((.((((.(((...((((....))))))).))))))..))).....------ ( -37.00, z-score = -1.29, R) >droYak2.chr2L 8710077 97 - 22324452 ------CUCCGUCUCGUCCAAUGAGUUGACUUCACUGGACGACCCGGAGCUGCCCAC-GUAGCGCACUGUGCUGGGGCUUCCCAUAGAGCGCACUAGGCUGUCC------ ------(((((..(((((((.((((.....)))).)))))))..)))))..(((...-((.((((.(((...((((....))))))).))))))..))).....------ ( -41.00, z-score = -2.50, R) >droEre2.scaffold_4929 5666853 97 + 26641161 ------CUCCGUCUCGUCCAAUGAGUUCACUUCACUGCACGACCCGGAGCUGCCCAC-GUAGCGCACUGUGCUGGGGCUUCCCAUGCAGCGCACUAGGCUGUCC------ ------(((((..((((.((.((((.....)))).)).))))..)))))..(((...-.(((.((.(((((.((((....))))))))).)).)))))).....------ ( -35.80, z-score = -1.29, R) >droAna3.scaffold_12916 11028349 97 + 16180835 ------CUCCGUUUCGUCCAACGAGACGAGGUCGCUGGACGACACGGAGCUGCCCAC-GUAGCGCACCGUGCUGGGACUGCCCAUGGCCGGCAGGAGGCUCCCC------ ------((((((.(((((((.(((.......))).))))))).))))))..((((.(-.((((((...)))))).).(((((.......)))))).))).....------ ( -43.40, z-score = -1.23, R) >dp4.chr4_group4 3330647 97 - 6586962 ------AUCCGUUUCGUCCAUGAAAUUGGCGUCACAGGACGAAAGGGAACUGUCCAC-AUAACGUAAUGUGCGGGGAUUCCCAAUGGCAGGAACGAGUUUGCCA------ ------.(((.((((((((.(((........)))..)))))))).)))......(((-((......))))).(((....)))..(((((((......)))))))------ ( -32.30, z-score = -1.44, R) >droPer1.super_10 1155131 97 - 3432795 ------AUCCGUUUCGUCCAUGAAAUUGGCGUCACAGGACGAAAGGGAACUGUCCAC-AUAACGUAAUGUGCGGGGAUUCCCAAUGGCAGGAACGAGUUUGCCA------ ------.(((.((((((((.(((........)))..)))))))).)))......(((-((......))))).(((....)))..(((((((......)))))))------ ( -32.30, z-score = -1.44, R) >droWil1.scaffold_180708 8714467 100 + 12563649 ------CUCCGUCUCGGCCAUCGAAUUGAUAUCGCUGGCUGAAACGGAGCCGGGCAC-AUAGCGUACUGUUGACGGACUGCCUGGCAUAUGCCUAGAAACGCAUCCC--- ------.(((((.(((((((.(((.......))).))))))).)))))((((((((.-.....((.(((....)))))))))))))..((((........))))...--- ( -40.10, z-score = -3.28, R) >droGri2.scaffold_15252 3073829 85 + 17193109 ------CUCCGUAUCGGCCAUCGAGUUGAGGUCGCAGGACGAUGCGGAGUGGUCCAC-AUAACGAGCUGUGGGCGGACUGGGUAGCUGCUCA------------------ ------((((((((((.((..(((.......)))..)).))))))))))..((((((-(........))))))).....((((....)))).------------------ ( -33.20, z-score = -0.87, R) >droMoj3.scaffold_6500 13911888 85 + 32352404 ------CUCCGUGCCGUCCGACGAGUUGAGCUCGCUGGACGAAGCGGAGCGGCCCAC-AUAGCGCACCGUGCUCGGGC--ACGGACUAGAGACA---------------- ------.(((((((((((((.((((.....)))).)))))((.((((.(((......-....))).))))..)).)))--))))).........---------------- ( -40.80, z-score = -2.53, R) >droVir3.scaffold_12963 3526153 94 + 20206255 ------CUCCGUGUCGGCCAUCGAGUUGAGCUCGCAGGACGAUGCGGAGCGGCCCAC-AUAGCGCACCGUGCUAGGACUAGAGAGCAAGUGGCGCGGCUCA--------- ------...(((((((.((..((((.....))))..)).)))))))((((.((((((-.((((((...)))))).(.((....)))..)))).)).)))).--------- ( -39.80, z-score = -0.86, R) >anoGam1.chr2R 42439992 108 - 62725911 AUCGAACUCCACCUCCAGCAGUUGGUAGAGAUGGCGGAAC--UGGGGAGCCGCCAGCUGCAACACGUUCAGACGGAUCAACUCCUUCAGCAGCAGUGCACGGGACGGCGA ......(((((..(((.((.(((......))).)))))..--))))).((((((.(((((.....((....))(((.....)))....))))).(....).)).)))).. ( -33.40, z-score = 0.82, R) >consensus ______CUCCGUCUCGUCCAAUGAGUUGACUUCACUGGACGACACGGAGCUGCCCAC_GUAGCGCACUGUGCUGGGACUACCCAUAGAGCGCACGAGGCUGUCC______ ......((((...(((.((..(((.......)))..)).)))...))))..((((......(((...)))....))))................................ (-10.07 = -10.32 + 0.26)

| Location | 5,580,624 – 5,580,721 |
|---|---|
| Length | 97 |
| Sequences | 13 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 62.55 |
| Shannon entropy | 0.80766 |
| G+C content | 0.60746 |
| Mean single sequence MFE | -40.12 |
| Consensus MFE | -13.58 |
| Energy contribution | -14.03 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.58 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.34 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.86 |
| SVM RNA-class probability | 0.995903 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
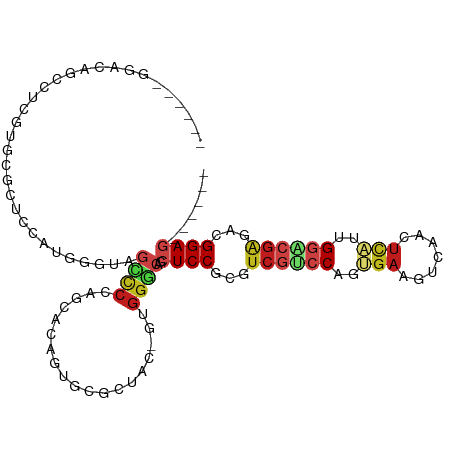
>dm3.chr2L 5580624 97 - 23011544 ------GGACAGCCUCGUGCGCUCUAUGGGUAGCCCCAGCACAGUGCGCUAC-GUGGGCAGCUCCGAGUCGUCCAGUGAAGUCAACUCAUUGGACGAGACGGAG------ ------.....(((((((((((.((.((((....))))....)).)))).))-).))))..(((((..(((((((((((.......)))))))))))..)))))------ ( -48.30, z-score = -4.00, R) >droSim1.chr2L 5375034 97 - 22036055 ------GGACAGCAUAGUGCGCUCUAUGGGUAGCCCCAGCACAGUGCGCUAC-GUGGGCAGCUCCGAGUCGUCCAGUGAAGUCAACUCAUUGGGCGAGACGGAG------ ------.(...((.(((((((((...((((....))))....))))))))).-))...)..(((((..(((((((((((.......)))))))))))..)))))------ ( -44.70, z-score = -3.30, R) >droSec1.super_5 3658466 97 - 5866729 ------GGACAGCCUAGUGCGCUCUAUGGGUAGCCCCAGCACAGUGCGCUAC-GUGGGCAGCUCCGAGUCGUCCAGUGAAGUCAACUCAUUGGGCGAGACGGAG------ ------.....(((((((((((.((.((((....))))....)).)))).))-.)))))..(((((..(((((((((((.......)))))))))))..)))))------ ( -46.90, z-score = -3.51, R) >droYak2.chr2L 8710077 97 + 22324452 ------GGACAGCCUAGUGCGCUCUAUGGGAAGCCCCAGCACAGUGCGCUAC-GUGGGCAGCUCCGGGUCGUCCAGUGAAGUCAACUCAUUGGACGAGACGGAG------ ------.....(((((((((((.((.((((....))))....)).)))).))-.)))))..(((((..(((((((((((.......)))))))))))..)))))------ ( -50.00, z-score = -4.44, R) >droEre2.scaffold_4929 5666853 97 - 26641161 ------GGACAGCCUAGUGCGCUGCAUGGGAAGCCCCAGCACAGUGCGCUAC-GUGGGCAGCUCCGGGUCGUGCAGUGAAGUGAACUCAUUGGACGAGACGGAG------ ------.....(((((((((((((..((((....))))...)))))))))..-..))))..(((((..((((.((((((.(....))))))).))))..)))))------ ( -50.00, z-score = -4.12, R) >droAna3.scaffold_12916 11028349 97 - 16180835 ------GGGGAGCCUCCUGCCGGCCAUGGGCAGUCCCAGCACGGUGCGCUAC-GUGGGCAGCUCCGUGUCGUCCAGCGACCUCGUCUCGUUGGACGAAACGGAG------ ------((....))..(((((.....((((....)))).((((........)-))))))))((((((.(((((((((((.......))))))))))).))))))------ ( -47.60, z-score = -1.72, R) >dp4.chr4_group4 3330647 97 + 6586962 ------UGGCAAACUCGUUCCUGCCAUUGGGAAUCCCCGCACAUUACGUUAU-GUGGACAGUUCCCUUUCGUCCUGUGACGCCAAUUUCAUGGACGAAACGGAU------ ------(((((..........)))))..(((....))).(((((......))-)))......(((.((((((((.((((........)))))))))))).))).------ ( -31.10, z-score = -2.04, R) >droPer1.super_10 1155131 97 + 3432795 ------UGGCAAACUCGUUCCUGCCAUUGGGAAUCCCCGCACAUUACGUUAU-GUGGACAGUUCCCUUUCGUCCUGUGACGCCAAUUUCAUGGACGAAACGGAU------ ------(((((..........)))))..(((....))).(((((......))-)))......(((.((((((((.((((........)))))))))))).))).------ ( -31.10, z-score = -2.04, R) >droWil1.scaffold_180708 8714467 100 - 12563649 ---GGGAUGCGUUUCUAGGCAUAUGCCAGGCAGUCCGUCAACAGUACGCUAU-GUGCCCGGCUCCGUUUCAGCCAGCGAUAUCAAUUCGAUGGCCGAGACGGAG------ ---(((..((((..((.(((....))).(((.....)))...)).))))...-...)))..(((((((((.((((.(((.......))).)))).)))))))))------ ( -40.60, z-score = -2.96, R) >droGri2.scaffold_15252 3073829 85 - 17193109 ------------------UGAGCAGCUACCCAGUCCGCCCACAGCUCGUUAU-GUGGACCACUCCGCAUCGUCCUGCGACCUCAACUCGAUGGCCGAUACGGAG------ ------------------..............((((((..((.....))...-))))))..(((((.((((.((..(((.......)))..)).)))).)))))------ ( -24.00, z-score = -0.59, R) >droMoj3.scaffold_6500 13911888 85 - 32352404 ----------------UGUCUCUAGUCCGU--GCCCGAGCACGGUGCGCUAU-GUGGGCCGCUCCGCUUCGUCCAGCGAGCUCAACUCGUCGGACGGCACGGAG------ ----------------.........(((((--(((.((((..(((.(((...-))).)))))))......((((.(((((.....))))).)))))))))))).------ ( -41.00, z-score = -2.48, R) >droVir3.scaffold_12963 3526153 94 - 20206255 ---------UGAGCCGCGCCACUUGCUCUCUAGUCCUAGCACGGUGCGCUAU-GUGGGCCGCUCCGCAUCGUCCUGCGAGCUCAACUCGAUGGCCGACACGGAG------ ---------.....((((((...((((..........)))).))))))(..(-((.((((((((.(((......)))))))((.....)).)))).)))..)..------ ( -32.90, z-score = 0.39, R) >anoGam1.chr2R 42439992 108 + 62725911 UCGCCGUCCCGUGCACUGCUGCUGAAGGAGUUGAUCCGUCUGAACGUGUUGCAGCUGGCGGCUCCCCA--GUUCCGCCAUCUCUACCAACUGCUGGAGGUGGAGUUCGAU ..((((((...((((..((..(.((.(((.....))).)).)...))..))))...))))))......--......(((((((((.(....).)))))))))........ ( -33.30, z-score = 0.91, R) >consensus ______GGACAGCCUCGUGCGCUCCAUGGGUAGCCCCAGCACAGUGCGCUAC_GUGGGCAGCUCCGCGUCGUCCAGUGAAGUCAACUCAUUGGACGAGACGGAG______ ................................((((......((....)).....))))..((((...((((((..(((.......)))..))))))...))))...... (-13.58 = -14.03 + 0.45)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:19:25 2011