
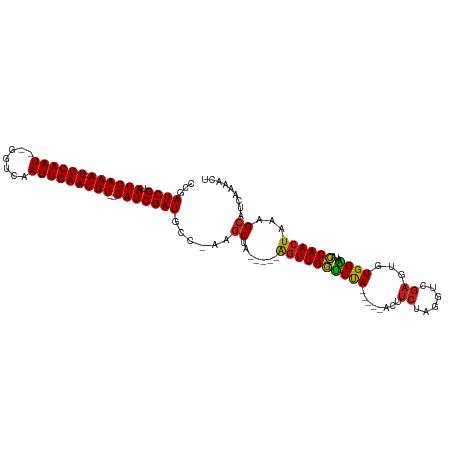
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 17,381,973 – 17,382,076 |
| Length | 103 |
| Max. P | 0.947914 |

| Location | 17,381,973 – 17,382,076 |
|---|---|
| Length | 103 |
| Sequences | 12 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 86.52 |
| Shannon entropy | 0.27693 |
| G+C content | 0.38492 |
| Mean single sequence MFE | -28.32 |
| Consensus MFE | -15.84 |
| Energy contribution | -15.12 |
| Covariance contribution | -0.73 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.93 |
| Structure conservation index | 0.56 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.53 |
| SVM RNA-class probability | 0.947914 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
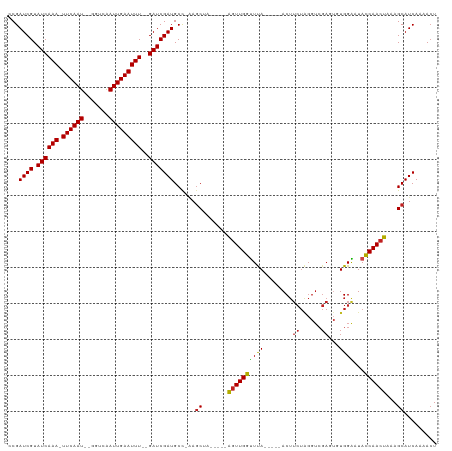
>dm3.chr3L 17381973 103 + 24543557 CCGAUCGUAUCAAA-UUCAAU--GGACGAUUGAAUUU--GAUCGAUGCC-AAGCUA-----AGUUGGUUUA-----ACUUCUAGGUCGAGUGUGGAAUACCAACUAAAGCAUCAAAACU ...((((.((((((-((((((--.....)))))))))--)))))))...-..(((.-----(((((((...-----..(((((.(.....).))))).)))))))..)))......... ( -29.30, z-score = -3.20, R) >droEre2.scaffold_4784 9295006 103 - 25762168 CCGAUCGUAUCAAA-UUCAAU--GAACGAUUGAAUUU--GAUCGAUGCC-AAGCUA-----AGUUGGUUUA-----ACUUCUAGGUCGAGUGUGGAAUACCAACUAAAGCAUCAAAACU ...((((.((((((-((((((--.....)))))))))--)))))))...-..(((.-----(((((((...-----..(((((.(.....).))))).)))))))..)))......... ( -29.30, z-score = -3.60, R) >droYak2.chr3L 7511024 103 - 24197627 CCGAUCGUAUCAAA-UUCAAU--GGACGAUUGAAUUU--GAUCGAUGCC-AAGCUA-----AGUUGGUUUA-----ACUUCUAGGUCGAGUGUGGAAUACCAACUAAAGCAUCAAAACU ...((((.((((((-((((((--.....)))))))))--)))))))...-..(((.-----(((((((...-----..(((((.(.....).))))).)))))))..)))......... ( -29.30, z-score = -3.20, R) >droSec1.super_0 9433717 103 + 21120651 CCGAUCGUAUCAAA-UUCAAU--GGACGAUUGAAUUU--GAUCGAUGCC-AAGCUA-----AGUUGGUUUA-----ACUUCUAGGUCGAGUGUGGAAUACCAACUAAAGCAUCAAAACU ...((((.((((((-((((((--.....)))))))))--)))))))...-..(((.-----(((((((...-----..(((((.(.....).))))).)))))))..)))......... ( -29.30, z-score = -3.20, R) >droSim1.chr3L 16722974 103 + 22553184 CCGAUCGUAUCAAA-UUCAAU--GGACGAUUGAAUUU--GAUCGAUGCC-AAGCUA-----AGUUGGUUUA-----ACUUCUAGGUCGAGUGUGGAAUACCAACUAAAGCAUCAAAACU ...((((.((((((-((((((--.....)))))))))--)))))))...-..(((.-----(((((((...-----..(((((.(.....).))))).)))))))..)))......... ( -29.30, z-score = -3.20, R) >dp4.chrXR_group6 4056356 104 - 13314419 CCGAUCGUAUCAAAAUUCAAU--GGUCAAUUGAAUUU--GAUCGAUGCC-CAGCUA-----AGUUGGUUUA-----ACUUCUAGGUCGAGUGUGGAAUACCAACUAAAGCAUCAAAACU ...((((.((((((.((((((--.....)))))))))--)))))))...-..(((.-----(((((((...-----..(((((.(.....).))))).)))))))..)))......... ( -26.20, z-score = -2.41, R) >droPer1.super_35 464841 104 + 973955 CCGAUCGUAUCAAAAUUCAAU--GGUCAAUUGAAUUU--GAUCGAUGCC-CAGCUA-----AGUUGGUUUA-----ACUUCUAGGUCGAGUGUGGAAUACCAACUAAAGCAUCAAAACU ...((((.((((((.((((((--.....)))))))))--)))))))...-..(((.-----(((((((...-----..(((((.(.....).))))).)))))))..)))......... ( -26.20, z-score = -2.41, R) >droWil1.scaffold_180698 1627558 116 - 11422946 CCGAUCGUAUCAAAAUUCAAUG-GUCAAAUUGAAUUU--GAUCGAUGCCCAAACUUUUGUUGGUUGGUUUAUUUAAACUUCUAGGUCGAGUGUGGGAUACCAACUAAAGCAUCAAAACU ..(((((......((((((((.-.....)))))))))--))))(((((.(((....)))(((((((((..((((..((((.......))))...))))))))))))).)))))...... ( -27.90, z-score = -1.78, R) >droAna3.scaffold_13337 8334763 105 + 23293914 CCGAUCGUAUCAAA-UUCAAUUCGGUCGAUUGAAUUU--GAUCGAUGCC-AAGCUA-----AGUUGGUUUA-----ACUUCUAGGUCGAGUGUGGAAUACCAACUAAAGCAUCAAAACU ...((((.((((((-(((((((.....))))))))))--)))))))...-..(((.-----(((((((...-----..(((((.(.....).))))).)))))))..)))......... ( -30.00, z-score = -3.27, R) >droVir3.scaffold_13049 19328219 109 - 25233164 ACGAUCGUAUCAAAAUUCAAU--GGUCAAUUGAAUUUUUGAUCGAUGCC-AAGCAA--AAAAGUUGCUUCA-----ACUUCUGGGUCGACACUGGGCUAGCAACUAAAGCAUCAAAACU ..(((((....((((((((((--.....)))))))))))))))(((((.-((((((--.....))))))..-----....((((.(((....))).))))........)))))...... ( -27.60, z-score = -2.69, R) >droMoj3.scaffold_6680 20526360 109 - 24764193 UCGAUCGUAUCAAAAUUCAAU--GGUCAAUUGAAUUUUGGAUCGAUGCU-GAGCAA--AAAAAUUGCCUCA-----ACUUCUGGGUCGACUCUGGGCUAGCAACUAAAGCAUCAAAACU ..((((....(((((((((((--.....)))))))))))))))((((((-((((((--.....))).))).-----....((((.(((....))).)))).......))))))...... ( -30.40, z-score = -3.45, R) >droGri2.scaffold_15110 13183850 96 + 24565398 -CGAUCGUAUCAAAAUUCAAU--GGUCAAUUGAAUUUUUGAUCGAUGCC-AAGCAG--AAAAGUUGCCUCA-----AC------------ACUGGGCUAGCAACUAAAGCAUCAAAAGU -.(((((....((((((((((--.....)))))))))))))))((((((-.....)--...((((((((((-----..------------..))))...))))))...)))))...... ( -25.00, z-score = -2.73, R) >consensus CCGAUCGUAUCAAA_UUCAAU__GGUCAAUUGAAUUU__GAUCGAUGCC_AAGCUA_____AGUUGGUUUA_____ACUUCUAGGUCGAGUGUGGAAUACCAACUAAAGCAUCAAAACU ..((((.....(((.((((((.......)))))))))..))))(((((.............((((((((((.....(((....)))......))))...))))))...)))))...... (-15.84 = -15.12 + -0.73)

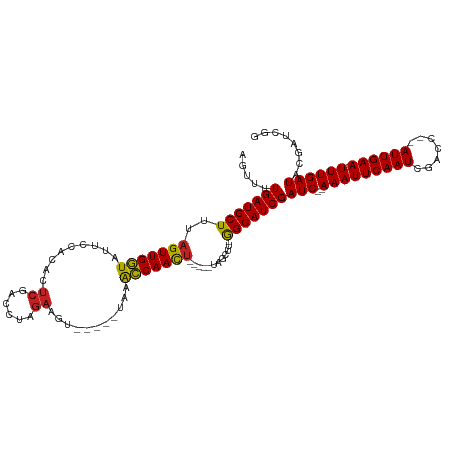
| Location | 17,381,973 – 17,382,076 |
|---|---|
| Length | 103 |
| Sequences | 12 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 86.52 |
| Shannon entropy | 0.27693 |
| G+C content | 0.38492 |
| Mean single sequence MFE | -25.94 |
| Consensus MFE | -18.13 |
| Energy contribution | -17.59 |
| Covariance contribution | -0.54 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.31 |
| Structure conservation index | 0.70 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.34 |
| SVM RNA-class probability | 0.928673 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 17381973 103 - 24543557 AGUUUUGAUGCUUUAGUUGGUAUUCCACACUCGACCUAGAAGU-----UAAACCAACU-----UAGCUU-GGCAUCGAUC--AAAUUCAAUCGUCC--AUUGAA-UUUGAUACGAUCGG .....(((((((..(((((((.......((((......).)))-----...)))))))-----......-)))))))(((--(((((((((.....--))))))-))))))........ ( -26.30, z-score = -2.68, R) >droEre2.scaffold_4784 9295006 103 + 25762168 AGUUUUGAUGCUUUAGUUGGUAUUCCACACUCGACCUAGAAGU-----UAAACCAACU-----UAGCUU-GGCAUCGAUC--AAAUUCAAUCGUUC--AUUGAA-UUUGAUACGAUCGG .....(((((((..(((((((.......((((......).)))-----...)))))))-----......-)))))))(((--(((((((((.....--))))))-))))))........ ( -26.30, z-score = -2.72, R) >droYak2.chr3L 7511024 103 + 24197627 AGUUUUGAUGCUUUAGUUGGUAUUCCACACUCGACCUAGAAGU-----UAAACCAACU-----UAGCUU-GGCAUCGAUC--AAAUUCAAUCGUCC--AUUGAA-UUUGAUACGAUCGG .....(((((((..(((((((.......((((......).)))-----...)))))))-----......-)))))))(((--(((((((((.....--))))))-))))))........ ( -26.30, z-score = -2.68, R) >droSec1.super_0 9433717 103 - 21120651 AGUUUUGAUGCUUUAGUUGGUAUUCCACACUCGACCUAGAAGU-----UAAACCAACU-----UAGCUU-GGCAUCGAUC--AAAUUCAAUCGUCC--AUUGAA-UUUGAUACGAUCGG .....(((((((..(((((((.......((((......).)))-----...)))))))-----......-)))))))(((--(((((((((.....--))))))-))))))........ ( -26.30, z-score = -2.68, R) >droSim1.chr3L 16722974 103 - 22553184 AGUUUUGAUGCUUUAGUUGGUAUUCCACACUCGACCUAGAAGU-----UAAACCAACU-----UAGCUU-GGCAUCGAUC--AAAUUCAAUCGUCC--AUUGAA-UUUGAUACGAUCGG .....(((((((..(((((((.......((((......).)))-----...)))))))-----......-)))))))(((--(((((((((.....--))))))-))))))........ ( -26.30, z-score = -2.68, R) >dp4.chrXR_group6 4056356 104 + 13314419 AGUUUUGAUGCUUUAGUUGGUAUUCCACACUCGACCUAGAAGU-----UAAACCAACU-----UAGCUG-GGCAUCGAUC--AAAUUCAAUUGACC--AUUGAAUUUUGAUACGAUCGG ......(((((((.(((((((.......((((......).)))-----...)))))))-----.....)-))))))((((--(((((((((.....--)))))))))......)))).. ( -23.70, z-score = -1.59, R) >droPer1.super_35 464841 104 - 973955 AGUUUUGAUGCUUUAGUUGGUAUUCCACACUCGACCUAGAAGU-----UAAACCAACU-----UAGCUG-GGCAUCGAUC--AAAUUCAAUUGACC--AUUGAAUUUUGAUACGAUCGG ......(((((((.(((((((.......((((......).)))-----...)))))))-----.....)-))))))((((--(((((((((.....--)))))))))......)))).. ( -23.70, z-score = -1.59, R) >droWil1.scaffold_180698 1627558 116 + 11422946 AGUUUUGAUGCUUUAGUUGGUAUCCCACACUCGACCUAGAAGUUUAAAUAAACCAACCAACAAAAGUUUGGGCAUCGAUC--AAAUUCAAUUUGAC-CAUUGAAUUUUGAUACGAUCGG .((((....(((((.((((((.........((......)).((((....))))..)))))).)))))..))))(((((((--(((((((((.....-.)))))).)))))).))))... ( -25.00, z-score = -1.59, R) >droAna3.scaffold_13337 8334763 105 - 23293914 AGUUUUGAUGCUUUAGUUGGUAUUCCACACUCGACCUAGAAGU-----UAAACCAACU-----UAGCUU-GGCAUCGAUC--AAAUUCAAUCGACCGAAUUGAA-UUUGAUACGAUCGG .....(((((((..(((((((.......((((......).)))-----...)))))))-----......-)))))))(((--((((((((((....).))))))-))))))........ ( -26.80, z-score = -2.59, R) >droVir3.scaffold_13049 19328219 109 + 25233164 AGUUUUGAUGCUUUAGUUGCUAGCCCAGUGUCGACCCAGAAGU-----UGAAGCAACUUUU--UUGCUU-GGCAUCGAUCAAAAAUUCAAUUGACC--AUUGAAUUUUGAUACGAUCGU ......((((((..(((((((.........(((((......))-----))))))))))...--......-))))))((((.((((((((((.....--)))))))))).....)))).. ( -27.10, z-score = -1.94, R) >droMoj3.scaffold_6680 20526360 109 + 24764193 AGUUUUGAUGCUUUAGUUGCUAGCCCAGAGUCGACCCAGAAGU-----UGAGGCAAUUUUU--UUGCUC-AGCAUCGAUCCAAAAUUCAAUUGACC--AUUGAAUUUUGAUACGAUCGA ......((((((..(((.....((((((..((......))..)-----)).))).......--..))).-))))))(((((((((((((((.....--)))))))))))....)))).. ( -28.74, z-score = -2.67, R) >droGri2.scaffold_15110 13183850 96 - 24565398 ACUUUUGAUGCUUUAGUUGCUAGCCCAGU------------GU-----UGAGGCAACUUUU--CUGCUU-GGCAUCGAUCAAAAAUUCAAUUGACC--AUUGAAUUUUGAUACGAUCG- .........((.......))..(((.(((------------(.-----.(((....)))..--).))).-)))(((((((((((.((((((.....--))))))))))))).))))..- ( -24.70, z-score = -2.31, R) >consensus AGUUUUGAUGCUUUAGUUGGUAUUCCACACUCGACCUAGAAGU_____UAAACCAACU_____UAGCUU_GGCAUCGAUC__AAAUUCAAUCGACC__AUUGAA_UUUGAUACGAUCGG ......((((((..(((((((.........((......))...........)))))))............))))))((((..(((((((((.......)))))).))).....)))).. (-18.13 = -17.59 + -0.54)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:33:34 2011