| Sequence ID | dm3.chr3L |
|---|---|
| Location | 17,308,080 – 17,308,218 |
| Length | 138 |
| Max. P | 0.999324 |

| Location | 17,308,080 – 17,308,218 |
|---|---|
| Length | 138 |
| Sequences | 13 |
| Columns | 159 |
| Reading direction | forward |
| Mean pairwise identity | 81.10 |
| Shannon entropy | 0.39410 |
| G+C content | 0.37310 |
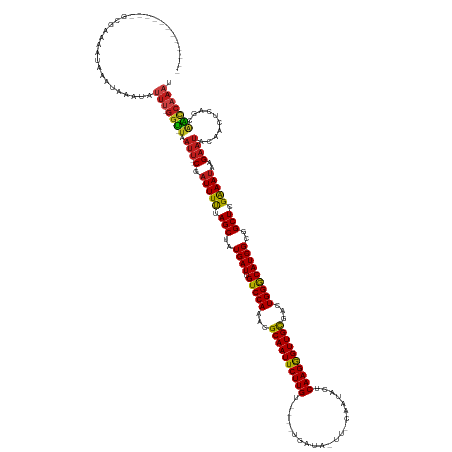
| Mean single sequence MFE | -39.32 |
| Consensus MFE | -25.64 |
| Energy contribution | -25.15 |
| Covariance contribution | -0.49 |
| Combinations/Pair | 1.20 |
| Mean z-score | -3.65 |
| Structure conservation index | 0.65 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.79 |
| SVM RNA-class probability | 0.999324 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 17308080 138 + 24543557 ----------GAGCGAAAAUAAAUAAAUAUUU-GGU-UAAUU--CGAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAUA-UU-CAAUAUUCAAGGGUUGCGACUGGGCAUC-GCGGCUCGAAAUAAGAAUACAACUCGGCAACCAAAU ----------..................((((-(((-(.(((--(.(((((.(((..((((.(((((..((((((((((((----((...-..-)))....)))))))))))..))))))))-)..))).)))))..)))).(.....)..)))))))) ( -39.30, z-score = -3.74, R) >droEre2.scaffold_4784 9220727 138 - 25762168 ----------AAGCGAAAAUAAAUAAAUAUUU-GGU-UAAUU--CGAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAUA-UU-CAAUAUUCAAGGGUUGCGACUGGGCAUC-GCGGCUCGAAAUAAGAAUACAACUCGGCAACCAAAU ----------..................((((-(((-(.(((--(.(((((.(((..((((.(((((..((((((((((((----((...-..-)))....)))))))))))..))))))))-)..))).)))))..)))).(.....)..)))))))) ( -39.30, z-score = -3.91, R) >droYak2.chr3L 7434916 138 - 24197627 ----------AAGCGAAAAUAAAUAAAUAUUU-GGU-UAAUU--CGAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAUA-UU-CAAUAUUCAAGGGUUGCGACUGGGCAUC-GCGGCUCGAAAUAAGAAUACAACUCGGCAACCAAAU ----------..................((((-(((-(.(((--(.(((((.(((..((((.(((((..((((((((((((----((...-..-)))....)))))))))))..))))))))-)..))).)))))..)))).(.....)..)))))))) ( -39.30, z-score = -3.91, R) >droSec1.super_0 9359453 138 + 21120651 ----------GAGCGAAAAUAAAUAAAUAUUU-GGU-UAAUU--CAAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAUA-UU-CAAUAUUCAAGGGUUGCGACUGGGCAUC-GCGGCUCGAAAUAAGAAUACAACUCGGCAACCAAAU ----------..................((((-(((-(....--..(((((.(((..((((.(((((..((((((((((((----((...-..-)))....)))))))))))..))))))))-)..))).)))))..((.......))...)))))))) ( -38.60, z-score = -3.90, R) >droSim1.chr3L 16646156 138 + 22553184 ----------GAGCGAAAAUAAAUAAAUAUUU-GGU-UAAUU--CGAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAUA-UU-CAAUAUUCAAGGGUUGCGACUGGGCAUC-GCGGCUCGAAAUAAGAAUACAACUCGGCAACCAAAU ----------..................((((-(((-(.(((--(.(((((.(((..((((.(((((..((((((((((((----((...-..-)))....)))))))))))..))))))))-)..))).)))))..)))).(.....)..)))))))) ( -39.30, z-score = -3.74, R) >droAna3.scaffold_13337 8265324 137 + 23293914 ------------ACCUGAAAAACAAAAUAUUUUGGU-UAAUU--CAAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAAU-UC-AAAUACUCAAGGGUUGCGACUGGGCAUC-GCGGCUCGAAAUACGAAUACAACGAAGAGAACCAUU ------------....................((((-(..((--(.(((((.(((..((((.(((((..((((((((((((----((...-.)-)).....)))))))))))..))))))))-)..))).)))))..(....)...)))...))))).. ( -37.80, z-score = -3.84, R) >dp4.chrXR_group6 3976172 148 - 13314419 AUACGAGUACUCGUAUCAGUAUCAAAAUAUUU-GGU-UAAUU--CGAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAUA-UC-CAAUACUCAAGGGUUGUGACUGGACAUC-GCGGCUCGAAAUAAGAAUACAACCCAGCAACCAAAA ((((((....)))))).............(((-(((-(.(((--(.(((((.(((..((((.(((((..(((((((((((.----..((.-..-..))...)))))))))))..))))))))-)..))).)))))..)))).(......).))))))). ( -41.40, z-score = -3.88, R) >droPer1.super_35 387434 148 + 973955 AUACGAGUACUCGUAUCAGUAUCAAAAUAUUU-GGU-UAAUU--CGAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAUA-UC-CAAUACUCAAGGGUUGUGACUGGACAUC-GCGGCUCGAAAUAAGAAUACAACCCAUCAACCAAAA ((((((....)))))).............(((-(((-(.(((--(.(((((.(((..((((.(((((..(((((((((((.----..((.-..-..))...)))))))))))..))))))))-)..))).)))))..))))..........))))))). ( -41.00, z-score = -3.96, R) >droWil1.scaffold_180955 2086465 134 + 2875958 -------------GAUAGAAAACAAAAUAUUU-GGU-UAAUU--CGAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAUU-UC-AAAUACUCAAGGGUUGCAUCUGGGCAUC-GCGGCUCGAAAUAAGAAUACA-CCCAAUAACCAAAG -------------................(((-(((-(((((--(.(((((.(((..((((.(((((...(((((((((((----((...-.)-)).....))))))))))...))))))))-)..))).)))))..))))...-.....)))))))). ( -37.10, z-score = -4.13, R) >droVir3.scaffold_13049 19241613 136 - 25233164 ----------GCGC-UAAGUGUAUAAAUAUUU-GGU-UAAUU--CGAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAUU-UC-AAAUACUCAAGGGUUGCGACUGGGCAUC-GCGGCUCGAAAUGAGAAUCCAACCCA-CAACCAAAC ----------..((-(((((((....))))))-)))-..(((--(.(((((.(((..((((.(((((..((((((((((((----((...-.)-)).....)))))))))))..))))))))-)..))).)))))..))))........-......... ( -40.20, z-score = -3.46, R) >droMoj3.scaffold_6680 20447164 136 - 24764193 ------------CUAAAUACAAACAUAUAUUU-GGU-UAAUU--CGAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAUU-UUUAAAUACUCAAGGGUUGCAUCUGGGCAUC-GCGGCUCGAAAUGAGAAUCCAACACA-AAGCCAAAA ------------.................(((-(((-(.(((--(.(((((.(((..((((.(((((...((((((((((.----..(((-....)))...))))))))))...))))))))-)..))).)))))..))))........-.))))))). ( -36.30, z-score = -3.60, R) >droGri2.scaffold_15110 13121809 133 + 24565398 ---------------GAACUUAACAAACAUUU-GGU-GAAUU--CGAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU----UGAUU-UC-AAAAACUCAAGGGUUGCAUCUGGGCAUC-GCGGCUCGAAAUGAGAAUACAACACAAAAAUCAAAA ---------------..............(((-(.(-(.(((--(.(((((.(((..((((.(((((...(((((((((((----((...-.)-)).....))))))))))...))))))))-)..))).)))))..)))).))...))))........ ( -35.80, z-score = -3.60, R) >anoGam1.chr2L 27524535 143 - 48795086 ----------------GAAGAAGAAGAAACCCAGGCAUCAUCAACAAUUUCUAGCUCUGAUUGUCCAAACGCAAUUCUUGUCCUGUGCUGGUUUAGCUACUCAAGAGUUGUGACUGGACAUCCGUGGCUCGGAAGCGGAUAUCAACGGGACAGUGCCAG ----------------.................(((((..((.....(((((((((.((..((((((..(((((((((((....((((((...)))).)).)))))))))))..))))))..)).)))).)))))((........)).))..))))).. ( -45.70, z-score = -1.83, R) >consensus ____________GCGAAAAUAAAUAAAUAUUU_GGU_UAAUU__CGAUUUUUAGCUAUGAUUGUCCAAACGCAAUUCUUGU____UGAUA_UU_CAAUACUCAAGGGUUGCGACUGGGCAUC_GCGGCUCGAAAUAAGAAUACAACUCAGCAACCAAAU ..............................................(((((.((((.....((((((...((((((((((.....................))))))))))...)))))).....)))).)))))........................ (-25.64 = -25.15 + -0.49)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:33:26 2011