
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 5,492,172 – 5,492,283 |
| Length | 111 |
| Max. P | 0.609709 |

| Location | 5,492,172 – 5,492,283 |
|---|---|
| Length | 111 |
| Sequences | 12 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 87.08 |
| Shannon entropy | 0.27569 |
| G+C content | 0.43156 |
| Mean single sequence MFE | -30.53 |
| Consensus MFE | -16.89 |
| Energy contribution | -16.70 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.55 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.609709 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 5492172 111 + 23011544 -GUCGACAUGUA----AUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCGUUAUGAUGGGGCAAUUAUAACUGUCAGAUUUGACAGCAUUACAGGCUCUC--- -...((((..((----((((.(.(((.....)))).))))))...)))).....(((((((...)))))))..(((((........(((((......)))))........))))).--- ( -31.89, z-score = -2.17, R) >droPer1.super_5 6399585 108 - 6813705 -CUCGACAUAUA----AUUGCGCGCUUUCCUAGCCGCAAUUAAAUUGUCAUAGACAUGACGCAGCAUUAUGAUGGGGCAAUUAUAACUGUCAGAUCUGACAGCAUUACGG-CUC----- -((((.((((((----((((((.(((.....)))))))))))....(((((....))))).......)))).))))((........((((((....)))))).......)-)..----- ( -31.26, z-score = -2.44, R) >dp4.chr4_group1 4803014 108 + 5278887 -CUCGACAUAUA----AUUGCGCGCUUUCCUAGCCGCAAUUAAAUUGUCAUAGACAUGACGCAGCAUUAUGAUGGGGCAAUUAUAACUGUCAGAUCUGACAGCAUUACGG-CUC----- -((((.((((((----((((((.(((.....)))))))))))....(((((....))))).......)))).))))((........((((((....)))))).......)-)..----- ( -31.26, z-score = -2.44, R) >droAna3.scaffold_12916 6523739 110 - 16180835 -GCCGACAUGUA----AUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCAUUAUGAUGGGGCAAUUAUAACUGUCAGAUCUGACAGCAUUACAG-CUCUC--- -(((((((..((----((((.(.(((.....)))).))))))...)))).....(((((.......)))))....)))........((((((....))))))........-.....--- ( -27.90, z-score = -1.12, R) >droEre2.scaffold_4929 5577206 111 + 26641161 -GUCGACAUGUA----AUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCGUUAUGAUGGGGCAAUUAUAACUGUCAGAUUUGACAGCAUUACAGGCUCUC--- -...((((..((----((((.(.(((.....)))).))))))...)))).....(((((((...)))))))..(((((........(((((......)))))........))))).--- ( -31.89, z-score = -2.17, R) >droYak2.chr2L 8620659 111 - 22324452 -CUCGACAUGUA----AUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCAUUAUGAUGGGGCAAUUAUAACUGUCAGAUUUGACAGCAUUACAGGCUCUC--- -...((((..((----((((.(.(((.....)))).))))))...)))).....(((((.......)))))..(((((........(((((......)))))........))))).--- ( -27.19, z-score = -1.14, R) >droSec1.super_5 3570719 111 + 5866729 -GUCGACAUGUA----AUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCGUUAUGAUGGGGCAAUUAUAGCUGUCAGAUUUGACAGCAUUACAGGCUCUC--- -...((((..((----((((.(.(((.....)))).))))))...)))).....(((((((...)))))))..(((((.......((((((......)))))).......))))).--- ( -35.44, z-score = -2.82, R) >droSim1.chr2L 5295032 111 + 22036055 -GUCGACAUGUA----AUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCGUUAUGAUGGGGCAAUUAUAACUGUCAGAUUUGACAGCAUUACAGGCUCUC--- -...((((..((----((((.(.(((.....)))).))))))...)))).....(((((((...)))))))..(((((........(((((......)))))........))))).--- ( -31.89, z-score = -2.17, R) >droWil1.scaffold_180708 663056 118 + 12563649 -CUCGACAUGUAUGUAAUUGCGCGAUUUUCUACCAACAAUUAAAUUGUCAUAGAGAUGACGAAGCAUUAUGACGGCGCAAUUAAAACUGUCAGAUUUGACAGCAUUACUACAUUGAAAC -.((((..((((..((((((((((((((.............)))))((((((...(((......))))))))).)))))))))...(((((......)))))......))))))))... ( -26.52, z-score = -1.97, R) >droVir3.scaffold_12963 451767 110 - 20206255 -ACCGACAUGUA----AUUGCGCGCUUUCCUAGCCACAAUUAAACUGUCAUAGACAUGACGCAGCAUUAUGAAGUCGCAAUUAUAGCUGUCACUUUUGACAGCACACAUGCCUCC---- -...((((..((----((((.(.(((.....)))).))))))...))))..((.((((..((.((........)).)).......(((((((....)))))))...)))).))..---- ( -30.30, z-score = -3.10, R) >droMoj3.scaffold_6500 23448010 110 + 32352404 -GUCGACAUGUA----AUUGCGCGCUUUCCUAGCCACAAUUAAACUGUCAUAGACAUGACGCAGCAUUAUGACGUCGCAAUUAUAGCUGUCACUUUUGACAGCAACUACACGCCC---- -(((((((..((----((((.(.(((.....)))).))))))...))))...)))..((((((......)).)))).........(((((((....)))))))............---- ( -31.00, z-score = -3.04, R) >droGri2.scaffold_15252 5973767 110 - 17193109 CUCUGACAUCUA----AUUGCGCGCUUUCCUAGCCACAAUUAAACUGUCAUAGACAUGACGCAGCAUUAUGACGUCGCAAUUAUAGCUGUCAUUUCUGACAGCCAACA-GCUCCU---- ((.(((((..((----((((.(.(((.....)))).))))))...))))).))....((((((......)).))))((.......(((((((....))))))).....-))....---- ( -29.80, z-score = -3.07, R) >consensus _GUCGACAUGUA____AUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCAUUAUGAUGGGGCAAUUAUAACUGUCAGAUUUGACAGCAUUACAGGCUCUC___ ..................((((((((.....)))............(((((....))))))).)))((((((........))))))((((((....))))))................. (-16.89 = -16.70 + -0.19)

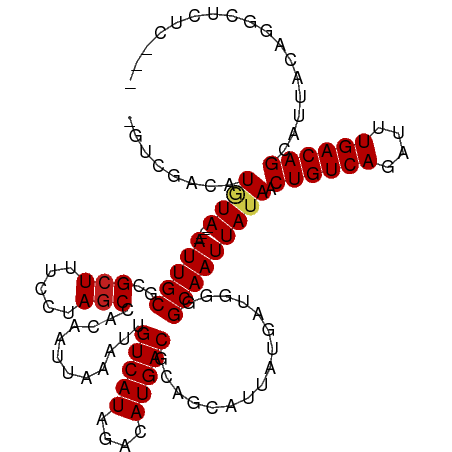

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:19:12 2011