| Sequence ID | dm3.chr2L |
|---|---|
| Location | 595,406 – 595,509 |
| Length | 103 |
| Max. P | 0.837760 |

| Location | 595,406 – 595,498 |
|---|---|
| Length | 92 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.35 |
| Shannon entropy | 0.32260 |
| G+C content | 0.39892 |
| Mean single sequence MFE | -25.94 |
| Consensus MFE | -19.06 |
| Energy contribution | -19.39 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.73 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.86 |
| SVM RNA-class probability | 0.837760 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
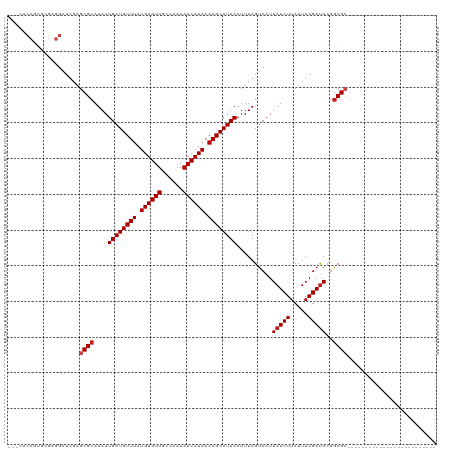
>dm3.chr2L 595406 92 - 23011544 ---CUCCGUGGGCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAGAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC------------------------- ---.((((....))))....((((....((((((((.((((((......)))))).))))))))..........(((((.....)))))..))))------------------------- ( -25.20, z-score = -1.95, R) >droSim1.chr2L 601853 92 - 22036055 ---CUCCGUGGGCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAGAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC------------------------- ---.((((....))))....((((....((((((((.((((((......)))))).))))))))..........(((((.....)))))..))))------------------------- ( -25.20, z-score = -1.95, R) >droSec1.super_14 578020 92 - 2068291 ---CUCCGUGGGCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAGAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC------------------------- ---.((((....))))....((((....((((((((.((((((......)))))).))))))))..........(((((.....)))))..))))------------------------- ( -25.20, z-score = -1.95, R) >droYak2.chr2L 584002 92 - 22324452 ---CUCCGUGGGCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAGAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC------------------------- ---.((((....))))....((((....((((((((.((((((......)))))).))))))))..........(((((.....)))))..))))------------------------- ( -25.20, z-score = -1.95, R) >droEre2.scaffold_4929 651134 92 - 26641161 ---CUCCGUGGGCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAGAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGACACGGCGC------------------------- ---.((((....))))....((((....((((((((.((((((......)))))).)))))))).....(((.(((......))))))...))))------------------------- ( -26.10, z-score = -2.18, R) >droAna3.scaffold_12916 1598084 91 - 16180835 ----CCCGUGGGCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGACGC------------------------- ----...(((.((.......)).)))..((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))..------------------------- ( -22.50, z-score = -1.68, R) >dp4.chr4_group3 10699567 120 + 11692001 UUCCGCCGCUCGAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUAUGGCGCUAUAUCAGAGGGAGAGCAGCAGAGC (((.((.((((((...))))))((....((((((((.((((((......)))))).))))))))............(((((((..((((((....))))))..))))))))).)).))). ( -35.50, z-score = -2.41, R) >droPer1.super_1 7812654 120 + 10282868 UUCCGCCGCUCGAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUAUGGCGCUAUAUCAGAGGGAGAGCAGCAGAGC (((.((.((((((...))))))((....((((((((.((((((......)))))).))))))))............(((((((..((((((....))))))..))))))))).)).))). ( -35.50, z-score = -2.41, R) >droWil1.scaffold_180772 6261938 90 + 8906247 -----GGCUACGAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACAGCGA------------------------- -----.(((.(((...))))))((....((((((((.((((((......)))))).))))))))..........(((((.....)))))..))..------------------------- ( -18.80, z-score = -0.96, R) >droVir3.scaffold_12963 19054141 100 - 20206255 ---GCCCGAGCGAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGCAGCGCAGC----------------- ---..((......)).....((((....((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))...))))...----------------- ( -26.40, z-score = -1.34, R) >droMoj3.scaffold_6500 8216182 100 + 32352404 ---GCCCGAGCGAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGCCGCGCAGC----------------- ---..((......)).....((((....((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))...))))...----------------- ( -26.50, z-score = -1.28, R) >droGri2.scaffold_15252 6953477 92 - 17193109 ---------CCGAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGGAGCAGA------------------- ---------.............((....((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))...))...------------------- ( -19.20, z-score = -0.69, R) >consensus ___CUCCGUGCGAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC_________________________ ....................((((....((((((((.((((((......)))))).))))))))..........(((((.....)))))..))))......................... (-19.06 = -19.39 + 0.33)


| Location | 595,406 – 595,509 |
|---|---|
| Length | 103 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 86.13 |
| Shannon entropy | 0.27779 |
| G+C content | 0.40930 |
| Mean single sequence MFE | -30.23 |
| Consensus MFE | -19.55 |
| Energy contribution | -20.03 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.65 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.60 |
| SVM RNA-class probability | 0.758953 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
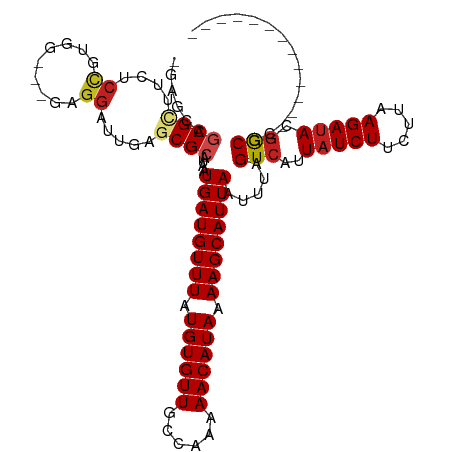
>dm3.chr2L 595406 103 - 23011544 -GAGCAGCGCUUCUCCGUGG---GCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAGAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC------------- -..((.((((((.((((...---.))))..))))))....((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))))------------- ( -32.60, z-score = -2.53, R) >droSim1.chr2L 601853 103 - 22036055 -GAGCAGCGCUUCUCCGUGG---GCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAGAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC------------- -..((.((((((.((((...---.))))..))))))....((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))))------------- ( -32.60, z-score = -2.53, R) >droSec1.super_14 578020 103 - 2068291 -GAGCAGCGCUUCUCCGUGG---GCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAGAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC------------- -..((.((((((.((((...---.))))..))))))....((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))))------------- ( -32.60, z-score = -2.53, R) >droYak2.chr2L 584002 104 - 22324452 GAAGCAGCGCUUCUCCGUGG---GCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAGAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC------------- ...((.((((((.((((...---.))))..))))))....((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))))------------- ( -32.60, z-score = -2.56, R) >droEre2.scaffold_4929 651134 104 - 26641161 GAAGCAGCGCUUCUCCGUGG---GCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAGAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGACACGGCGC------------- ...((.((((((.((((...---.))))..))))))....((((((((.((((((......)))))).)))))))).....(((.(((......))))))...))..------------- ( -32.20, z-score = -2.35, R) >droAna3.scaffold_12916 1598084 102 - 16180835 GAAG-AGCGCAUC-CCGUGG---GCGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGACGC------------- ....-.((((.((-(((...---.)))...))))))....((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))..------------- ( -28.00, z-score = -2.20, R) >dp4.chr4_group3 10699579 119 + 11692001 -GAGCUUCGGUUUUCCGCCGCUCGAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUAUGGCGCUAUAUCAGAGGGA -((((..(((....)))..))))...(((..(((((....((((((((.((((((......)))))).)))))))).......(((.((((.....))))))))))))..)))....... ( -35.30, z-score = -2.43, R) >droPer1.super_1 7812666 119 + 10282868 -GAGCUUCGGUUUUCCGCCGCUCGAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUAUGGCGCUAUAUCAGAGGGA -((((..(((....)))..))))...(((..(((((....((((((((.((((((......)))))).)))))))).......(((.((((.....))))))))))))..)))....... ( -35.30, z-score = -2.43, R) >droWil1.scaffold_180772 6261938 101 + 8906247 -AAAUAGCGUUCGGCU--AC---GAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACAGCGA------------- -.....((((((((((--..---..)).))))))))....((((((((.((((((......)))))).)))))))).....((...(((((.....)))))..))..------------- ( -25.10, z-score = -2.00, R) >droVir3.scaffold_12963 19054149 103 - 20206255 -GAGGAGCGUGCGCCCGAGC---GAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC------------- -.....((((((((.(((..---.....))).))))))..((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))))------------- ( -27.90, z-score = -1.39, R) >droMoj3.scaffold_6500 8216190 103 + 32352404 -GAGGAGCGUGCGCCCGAGC---GAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC------------- -.....((((((((.(((..---.....))).))))))..((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))))------------- ( -27.90, z-score = -1.39, R) >droGri2.scaffold_15252 6953483 97 - 17193109 -AAGAAGCGAGC------CC---GAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGG------------- -.........((------(.---...(((((((.......((((((((.((((((......)))))).)))))))).)))))))..(((((.....))))).)))..------------- ( -20.60, z-score = -0.73, R) >consensus _GAGCAGCGCUUCUCCGUGG___GAGGAUUGAGCGCAUAAUGAUGUUUAUGUGUUGCCAAAAACAUAAAAGCAUUAAUUUAGUCAUUAUCUUCUUAAGAUACGGCGC_____________ ......((((....((.........)).....))))....((((((((.((((((......)))))).)))))))).....(((..(((((.....))))).)))............... (-19.55 = -20.03 + 0.48)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:06:15 2011