| Sequence ID | dm3.chr3L |
|---|---|
| Location | 16,643,566 – 16,643,686 |
| Length | 120 |
| Max. P | 0.906098 |

| Location | 16,643,566 – 16,643,686 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.56 |
| Shannon entropy | 0.33958 |
| G+C content | 0.50446 |
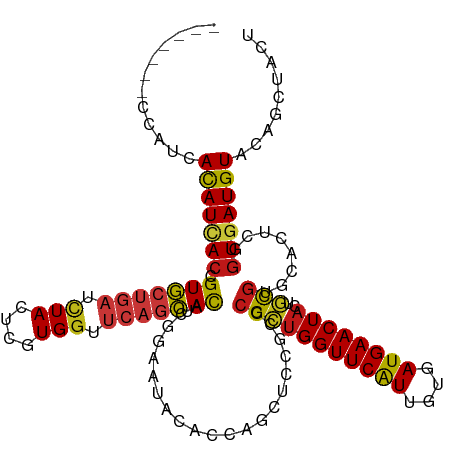
| Mean single sequence MFE | -38.20 |
| Consensus MFE | -24.18 |
| Energy contribution | -24.31 |
| Covariance contribution | 0.13 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.63 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.18 |
| SVM RNA-class probability | 0.906098 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
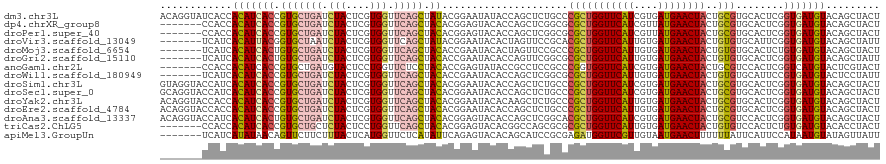
>dm3.chr3L 16643566 120 - 24543557 ACAGGUAUCACCACAUCACCGUGCUGAUCUACUCGUGGUUCAGCUAUACGGAAUAUACCAGCUCUGCCCGCUGGUUCAUCGUGAUGAACUACUGCGUGCACUCGGUGAUGUACAGCUACU ...((.....))(((((((((.(((((.(((....))).))))).....((......)).....(((.(((((((((((....))))))))..))).)))..)))))))))......... ( -40.70, z-score = -2.20, R) >dp4.chrXR_group8 8505377 113 - 9212921 -------CCACCACAUCACCGUGCUGAUCUACUCGUGGUUCAGCUACACGGAGUACACCAGCUCGGCGCGCUGGUUCAUCGUUAUGAACUACUGCGUGCACUCGGUGAUGUACAGCUACU -------.....(((((((((.((((...((((((((.........))).)))))...))))...((((((((((((((....))))))))..))))))...)))))))))......... ( -45.10, z-score = -3.42, R) >droPer1.super_40 713573 113 - 810552 -------CCACCACAUCACCGUGCUGAUCUACUCGUGGUUCAGCUACACGGAGUACACCAGCUCGGCGCGCUGGUUCAUCGUUAUGAACUACUGCGUGCACUCGGUGAUGUACAGCUACU -------.....(((((((((.((((...((((((((.........))).)))))...))))...((((((((((((((....))))))))..))))))...)))))))))......... ( -45.10, z-score = -3.42, R) >droVir3.scaffold_13049 3735946 113 - 25233164 -------UCAUCACAUUACGGUGCUAAUCUACUCGUGGUUCAGCUAUACGGAAUACACUAGUUCCGCACGCUGGUUCAUUGUGAUGAACUACUGUGUGCAUUCGGUGAUGUACAGCUAUU -------.(((((((((((((((......))).))))))..........(((((......)))))((((((((((((((....))))))))..)))))).....)))))).......... ( -34.30, z-score = -1.86, R) >droMoj3.scaffold_6654 2240725 113 + 2564135 -------UCAUCACAUCACUGUGCUGAUCUACUCGUGGUUCAGCUACACCGAAUACACUAGUUCCGCCCGCUGGUUCAUUGUGAUGAACUACUGUGUGCACUCUGUGAUGUACAGCUACU -------.....(((((((((((((((.(((....))).))))).)))..((((......)))).((.(((((((((((....))))))))..))).)).....)))))))......... ( -32.60, z-score = -1.42, R) >droGri2.scaffold_15110 13619415 113 + 24565398 -------UCAUCACAUCACUGUGCUGAUCUACUCGUGGUUCAGCUACACCGAAUACACCAGUUCGGCGCGCUGGUUCAUUGUGAUGAACUACUGUGUGCACUCGGUGAUGUACAGCUAUU -------.....(((((((((.(((((.(((....))).))))).....(((((......)))))((((((((((((((....))))))))..))))))...)))))))))......... ( -41.10, z-score = -2.87, R) >anoGam1.chr2L 33217784 113 - 48795086 -------CCACCACAUCACCGUGCUGAUGUACUCCUGGUUCUCCUACACCGAGUAUACCGCCUCCGCCCGGUGGUUCAUCGUGAUGAACUACUGCGUCCACUCGGUCAUGUACUCGUACU -------.((.(((......))).))..((((....(((........)))((((((((((.....((.(((((((((((....))))))))))).)).....))))...)))))))))). ( -35.30, z-score = -2.38, R) >droWil1.scaffold_180949 4918528 113 - 6375548 -------UCAUCACAUCACCGUGCUGAUCUACUCGUGGUUCAGCUACACCGAAUACACCAGCUCGGCGCGCUGGUUCAUUGUGAUGAACUACUGUGUGCAUUCCGUGAUGUACUCCUAUU -------.....(((((((((.((((...((.(((..((......))..))).))...)))).))((((((((((((((....))))))))..)))))).....)))))))......... ( -36.40, z-score = -2.48, R) >droSim1.chr3L 15974341 120 - 22553184 GUAGGUACCAUCACAUCACCGUGCUGAUCUACUCGUGGUUCAGCUACACGGAAUACACCAGCUCUGCCCGCUGGUUCAUCGUGAUGAACUACUGCGUGCACUCGGUGAUGUACAGCUACU ((((((((.(((((....(((((((((.(((....))).)))))...)))).............(((.(((((((((((....))))))))..))).)))....)))))))))..)))). ( -41.70, z-score = -2.07, R) >droSec1.super_0 8721426 120 - 21120651 GCAGGUACCAUCACAUCACCGUGCUGAUCUACUCGUGGUUCAGCUACACGGAAUACACCAGCUCUGCCCGCUGGUUCAUCGUGAUGAACUACUGCGUGCACUCGGUGAUGUACAGCUACU ((((....((((((....(((((((((.(((....))).)))))...)))).....((((((.......)))))).....)))))).....))))((((((.....).)))))....... ( -41.70, z-score = -2.01, R) >droYak2.chr3L 6770466 120 + 24197627 ACAGGUACCACCACAUCACCGUGCUGAUCUACUCGUGGUUCAGCUACACGGAAUACACAAGCUCUGCCCGCUGGUUCAUUGUGAUGAACUACUGCGUGCACUCGGUGAUGUACAGCUACU ...((.....))(((((((((((((((.(((....))).))))).)).................(((.(((((((((((....))))))))..))).)))...))))))))......... ( -39.60, z-score = -1.71, R) >droEre2.scaffold_4784 8577389 120 + 25762168 ACAGGUACCACCACAUCACCGUGCUGAUCUACUCGUGGUUCAGCUACACGGAAUACACCAGCUCUGCCCGCUGGUUCAUUGUGAUGAACUACUGCGUGCACUCGGUGAUGUACAGCUACU ...((.....))(((((((((((((((.(((....))).))))).))..((......)).....(((.(((((((((((....))))))))..))).)))...))))))))......... ( -40.20, z-score = -1.76, R) >droAna3.scaffold_13337 9342958 120 + 23293914 ACAGGUACCAUCACAUCACUGUGCUGAUCUACUCGUGGUUCAGCUACACGGAGUACACCAGCUCGGCACGCUGGUUCAUCGUGAUGAACUACUGCGUCCACUCGGUGAUGUACAGCUACU ...((((.(...(((((((((.((((...((((((((.........))).)))))...))))..((.((((((((((((....))))))))..))))))...)))))))))...).)))) ( -42.30, z-score = -1.92, R) >triCas2.ChLG5 1838125 113 - 18847211 -------CCACCACAUCACCGUGCUGCUCUACUCCUGGUUCAGCUACACGGAGUACACGGCCAGCGCGCGCUGGUUCAUUGUGAUGAACUACUGUGUCCACUCUGUGAUGUACACCUACU -------.....(((((((((((..(((..((.....))..)))..))))((((((((((.((((....))))((((((....))))))..))))))..)))).)))))))......... ( -39.60, z-score = -2.43, R) >apiMel3.GroupUn 96224275 113 - 399230636 -------UCAUCAUAUAACAGUUCUUCUUUACUCAUGGUUCUCAUAUUCAGAGUACACAGCAUCCGCGAGAUGGUUCGUUGUAAUGAACUUUUUUAUUCAUUCCAUAAUGUAUAGUUAUU -------.......((((((((((.....(((((.(((.........)))))))).(((((..(((.....)))...)))))...)))))....(((.((((....)))).)))))))). ( -17.30, z-score = -0.53, R) >consensus _______CCAUCACAUCACCGUGCUGAUCUACUCGUGGUUCAGCUACACGGAAUACACCAGCUCCGCCCGCUGGUUCAUUGUGAUGAACUACUGCGUGCACUCGGUGAUGUACAGCUACU ............(((((((.(((((((.(((....))).))))).)).....................(((((((((((....))))))))..)))........)))))))......... (-24.18 = -24.31 + 0.13)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:31:43 2011