
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 16,184,929 – 16,185,055 |
| Length | 126 |
| Max. P | 0.981128 |

| Location | 16,184,929 – 16,185,055 |
|---|---|
| Length | 126 |
| Sequences | 13 |
| Columns | 132 |
| Reading direction | forward |
| Mean pairwise identity | 81.55 |
| Shannon entropy | 0.42936 |
| G+C content | 0.39236 |
| Mean single sequence MFE | -30.45 |
| Consensus MFE | -21.53 |
| Energy contribution | -22.36 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.71 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.07 |
| SVM RNA-class probability | 0.981128 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
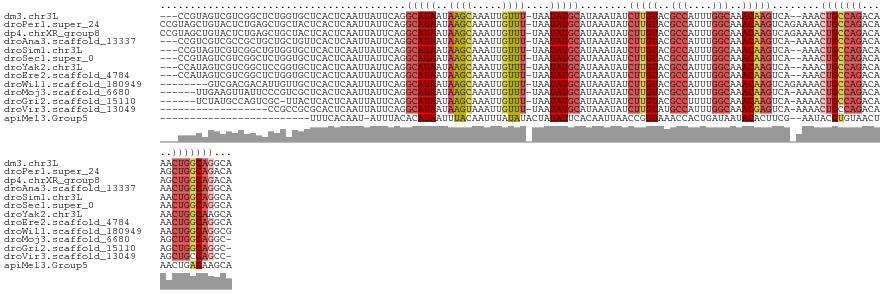
>dm3.chr3L 16184929 126 + 24543557 ---CCGUAGUCGUCGGCUCUGGUGCUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUACGCCAUUUGGCAAACAAGUCA--AAACUGCCAGACAAACUGGCAGGCA ---(((.(((.....))).)))(((.........((((...(((((..((((.....))))-...)))))...)))).(((((..(((....)))..)))))...--...(((((((.....)))))))))) ( -33.70, z-score = -2.22, R) >droPer1.super_24 368747 131 - 1556852 CCGUAGCUGUACUCUGAGCUGCUACUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUACGCCAUUUGGCAAACAAGUCAGAAAACUGCCAGACAAGCUGGCAGACA ..((((((........))))))..............(((..(((((..((((.....))))-...)))))........(((((..(((....)))..)))))...)))..(((((((.....)))))))... ( -36.60, z-score = -2.64, R) >dp4.chrXR_group8 1868436 131 + 9212921 CCGUAGCUGUACUCUGAGCUGCUACUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUACGCCAUUUGGCAAACAAGUCAGAAAACUGCCAGACAAGCUGGCAGACA ..((((((........))))))..............(((..(((((..((((.....))))-...)))))........(((((..(((....)))..)))))...)))..(((((((.....)))))))... ( -36.60, z-score = -2.64, R) >droAna3.scaffold_13337 12850706 127 - 23293914 ---CCGUCGUCGCCGCUGCUGCUGUUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUACGCCAUUUGGCAAACAAGUCA-AAAACUGCCAGACAAACUGGCAGGCA ---........(((((....))............((((...(((((..((((.....))))-...)))))...)))).(((((..(((....)))..)))))...-.....((((((.....))))))))). ( -31.00, z-score = -1.02, R) >droSim1.chr3L 15517191 126 + 22553184 ---CCGUAGUCGUCGGCUGUGGUGCUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUACGCCAUUUGGCAAACAAGUCA--AAACUGCCAGACAAACUGGCAGGCA ---(((((((.....)))))))(((.........((((...(((((..((((.....))))-...)))))...)))).(((((..(((....)))..)))))...--...(((((((.....)))))))))) ( -35.10, z-score = -2.20, R) >droSec1.super_0 8279526 126 + 21120651 ---CCGUAGUCGUCGGCUCUGGUGCUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUACGCCAUUUGGCAAACAAGUCA--AAACUGCCAGACAAACUGGCAGGCA ---(((.(((.....))).)))(((.........((((...(((((..((((.....))))-...)))))...)))).(((((..(((....)))..)))))...--...(((((((.....)))))))))) ( -33.70, z-score = -2.22, R) >droYak2.chr3L 2188707 126 - 24197627 ---CCAUAGUCGUCGGCUCCGGUGCUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUACGCCAUUUGGCAAACAAGUCA--AAACUGCCAGACAAACUGGCAAGCA ---((..(((.....)))..))((((........((((...(((((..((((.....))))-...)))))...)))).(((((..(((....)))..)))))...--....((((((.....)))))))))) ( -29.20, z-score = -1.46, R) >droEre2.scaffold_4784 18427082 126 + 25762168 ---CCAUAGUCGUCGGCUCUGGUGCUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUACGCCAUUUGGCAAACAAGUCA--AAACUGCCAGACAAACUGGCAGGCA ---(((.(((.....))).)))(((.........((((...(((((..((((.....))))-...)))))...)))).(((((..(((....)))..)))))...--...(((((((.....)))))))))) ( -34.40, z-score = -2.71, R) >droWil1.scaffold_180949 6360527 123 - 6375548 --------GUCGACGACAUUGUUGCUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUACGCCAUUUGGCAAACAAGUCAGAAAACUGCCAGACAAACUGGCAGGCG --------......(((.(((((.......(((.((((...(((((..((((.....))))-...)))))...))))..)))...(((....))).))))))))......(((((((.....)))))))... ( -31.50, z-score = -2.49, R) >droMoj3.scaffold_6680 24621418 123 - 24764193 ------UUGAAGUUAUUCCCGUCGCUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUACGCCAUUUGGCAAACAAGUCA-AAAACUGCCAGACAAGCUGGCAGGC- ------((((........................((((...(((((..((((.....))))-...)))))...)))).(((((..(((....)))..))))))))-)...(((((((.....)))))))..- ( -29.80, z-score = -2.15, R) >droGri2.scaffold_15110 2290334 122 + 24565398 ------UCUAUGCCAGUCGC-UUACUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUACGCCUUUUGGCAAACAAGUCA-AAAACUGCCAGACAAGCUGGCAGGC- ------....((((((..((-.(((.........((((...(((((..((((.....))))-...)))))...))))....))).))...)))))).........-....(((((((.....)))))))..- ( -30.42, z-score = -2.35, R) >droVir3.scaffold_13049 1153698 111 + 25233164 ------------------CCGCCGCGCACUCAAUUAUUCAGGCAUAAUAAGUAAAUUGUUU-UAAUAUGCAUAAAUAUCUUGUAUGCCAUUUGGCAAACGAGUCA-AAAACUGCCAGACAAGCUGCCAGCC- ------------------..((.(.(((((........((((((((.((((........))-)).)))))........(((((.((((....)))).)))))...-....))).......)).)))).)).- ( -24.06, z-score = -0.84, R) >apiMel3.Group5 2419291 104 - 13386189 -------------------------UUUCACAAU-AUUUACACAUAAUUUACAAUUUAUAUACUAUAUUCACAAUUAACCGGUAAACCACUGAUAAUACACUUCG--AAUACGUGUAACUAACUGACAAGCA -------------------------..(((...(-(.((((((((((........))))......(((((..........((....))...((.........)))--)))).)))))).))..)))...... ( -9.80, z-score = -0.99, R) >consensus ___CCGUAGUCGUCGGCUCUGGUGCUCACUCAAUUAUUCAGGCAUAAUAAGCAAAUUGUUU_UAAUAUGCAUAAAUAUCUUGUACGCCAUUUGGCAAACAAGUCA_AAAACUGCCAGACAAACUGGCAGGCA .........................................(((((..((((.....))))....)))))........(((((..(((....)))..)))))........(((((((.....)))))))... (-21.53 = -22.36 + 0.84)


| Location | 16,184,929 – 16,185,055 |
|---|---|
| Length | 126 |
| Sequences | 13 |
| Columns | 132 |
| Reading direction | reverse |
| Mean pairwise identity | 81.55 |
| Shannon entropy | 0.42936 |
| G+C content | 0.39236 |
| Mean single sequence MFE | -33.28 |
| Consensus MFE | -25.45 |
| Energy contribution | -25.84 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.76 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.73 |
| SVM RNA-class probability | 0.963808 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
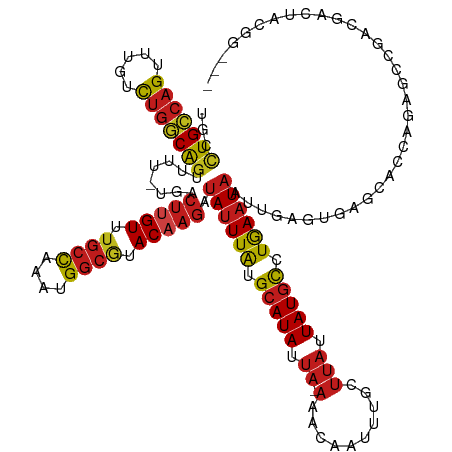
>dm3.chr3L 16184929 126 - 24543557 UGCCUGCCAGUUUGUCUGGCAGUUU--UGACUUGUUUGCCAAAUGGCGUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAGCACCAGAGCCGACGACUACGG--- ...(((((((.....)))))))(((--((.(((((.((((....)))).))))).((((((.(((((.((-(..........))).))))).)))))).............)))))(((.......)))--- ( -34.10, z-score = -1.95, R) >droPer1.super_24 368747 131 + 1556852 UGUCUGCCAGCUUGUCUGGCAGUUUUCUGACUUGUUUGCCAAAUGGCGUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAGUAGCAGCUCAGAGUACAGCUACGG ...(((((((.....)))))))....(((.(((((.((((....)))).))))).((((((.(((((.((-(..........))).))))).)))))).((((((........))))))....)))...... ( -37.00, z-score = -1.31, R) >dp4.chrXR_group8 1868436 131 - 9212921 UGUCUGCCAGCUUGUCUGGCAGUUUUCUGACUUGUUUGCCAAAUGGCGUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAGUAGCAGCUCAGAGUACAGCUACGG ...(((((((.....)))))))....(((.(((((.((((....)))).))))).((((((.(((((.((-(..........))).))))).)))))).((((((........))))))....)))...... ( -37.00, z-score = -1.31, R) >droAna3.scaffold_13337 12850706 127 + 23293914 UGCCUGCCAGUUUGUCUGGCAGUUUU-UGACUUGUUUGCCAAAUGGCGUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAACAGCAGCAGCGGCGACGACGG--- .(((((((((.....))))))(((.(-((.(((((.((((....)))).))))).((((((.(((((.((-(..........))).))))).))))))..........))).)))...)))........--- ( -37.10, z-score = -1.65, R) >droSim1.chr3L 15517191 126 - 22553184 UGCCUGCCAGUUUGUCUGGCAGUUU--UGACUUGUUUGCCAAAUGGCGUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAGCACCACAGCCGACGACUACGG--- ...(((((((.....)))))))...--...(((((.((((....)))).))))).((((((.(((((.((-(..........))).))))).)))))).....(((.....)))..(((.......)))--- ( -34.60, z-score = -2.09, R) >droSec1.super_0 8279526 126 - 21120651 UGCCUGCCAGUUUGUCUGGCAGUUU--UGACUUGUUUGCCAAAUGGCGUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAGCACCAGAGCCGACGACUACGG--- ...(((((((.....)))))))(((--((.(((((.((((....)))).))))).((((((.(((((.((-(..........))).))))).)))))).............)))))(((.......)))--- ( -34.10, z-score = -1.95, R) >droYak2.chr3L 2188707 126 + 24197627 UGCUUGCCAGUUUGUCUGGCAGUUU--UGACUUGUUUGCCAAAUGGCGUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAGCACCGGAGCCGACGACUAUGG--- ((((..((((((..((.((((....--...(((((.((((....)))).)))))........(((.(((.-....))).)))......)))).))..))))).)..)))).((....))..........--- ( -34.80, z-score = -1.94, R) >droEre2.scaffold_4784 18427082 126 - 25762168 UGCCUGCCAGUUUGUCUGGCAGUUU--UGACUUGUUUGCCAAAUGGCGUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAGCACCAGAGCCGACGACUAUGG--- ((((((((((.....)))))))...--...(((((.((((....)))).))))).((((((.(((((.((-(..........))).))))).)))))).........)))(((.((.......)).)))--- ( -34.40, z-score = -2.03, R) >droWil1.scaffold_180949 6360527 123 + 6375548 CGCCUGCCAGUUUGUCUGGCAGUUUUCUGACUUGUUUGCCAAAUGGCGUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAGCAACAAUGUCGUCGAC-------- ...(((((((.....))))))).....((((((((.((((....)))).))))((((((..(((......-.......(..((((((((......)))).))))..))))..))))))))))..-------- ( -33.41, z-score = -2.08, R) >droMoj3.scaffold_6680 24621418 123 + 24764193 -GCCUGCCAGCUUGUCUGGCAGUUUU-UGACUUGUUUGCCAAAUGGCGUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAGCGACGGGAAUAACUUCAA------ -..(((((((.....)))))))((((-((.(((((.((((....)))).))))).((((((.(((((.((-(..........))).))))).)))))).............)))))).........------ ( -32.30, z-score = -1.65, R) >droGri2.scaffold_15110 2290334 122 - 24565398 -GCCUGCCAGCUUGUCUGGCAGUUUU-UGACUUGUUUGCCAAAAGGCGUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAGUAA-GCGACUGGCAUAGA------ -...((((((((((.((.((......-...(((((.((((....)))).))))).((((((.(((((.((-(..........))).))))).)))))).....)).)))))-))...)))))....------ ( -35.00, z-score = -2.05, R) >droVir3.scaffold_13049 1153698 111 - 25233164 -GGCUGGCAGCUUGUCUGGCAGUUUU-UGACUCGUUUGCCAAAUGGCAUACAAGAUAUUUAUGCAUAUUA-AAACAAUUUACUUAUUAUGCCUGAAUAAUUGAGUGCGCGGCGG------------------ -.((((((.(((.....)))......-..((((((.((((....)))).))....((((((.(((((.((-(..........))).))))).))))))...)))))).))))..------------------ ( -31.30, z-score = -1.48, R) >apiMel3.Group5 2419291 104 + 13386189 UGCUUGUCAGUUAGUUACACGUAUU--CGAAGUGUAUUAUCAGUGGUUUACCGGUUAAUUGUGAAUAUAGUAUAUAAAUUGUAAAUUAUGUGUAAAU-AUUGUGAAA------------------------- .......((((...(((((((((..--(((.(((((((((..(..(((........)))..)....)))))))))...))).....)))))))))..-)))).....------------------------- ( -17.50, z-score = -0.09, R) >consensus UGCCUGCCAGUUUGUCUGGCAGUUUU_UGACUUGUUUGCCAAAUGGCGUACAAGAUAUUUAUGCAUAUUA_AAACAAUUUGCUUAUUAUGCCUGAAUAAUUGAGUGAGCACCAGAGCCGACGACUACGG___ ...(((((((.....)))))))........(((((.((((....)))).))))).((((((.(((((...................))))).)))))).................................. (-25.45 = -25.84 + 0.39)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:30:48 2011