
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 15,855,051 – 15,855,150 |
| Length | 99 |
| Max. P | 0.793960 |

| Location | 15,855,051 – 15,855,150 |
|---|---|
| Length | 99 |
| Sequences | 8 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 82.50 |
| Shannon entropy | 0.31979 |
| G+C content | 0.44680 |
| Mean single sequence MFE | -25.35 |
| Consensus MFE | -16.22 |
| Energy contribution | -16.12 |
| Covariance contribution | -0.09 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.64 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.793960 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 15855051 99 + 24543557 CUUUCUGUUCUAUUUGCCUAGUCCAUGCUGACCAUAUGGCUUAUGCAUAAAUCAUCGU------GUAUGAUGUGACCACCGCUCCACGUCAUCAUC--AUCAUGAUC------ ..............(((.(((.(((((.......))))).))).)))......(((((------(.((((((((((...........)))).))))--)))))))).------ ( -22.30, z-score = -1.43, R) >droAna3.scaffold_13337 11991102 107 - 23293914 CUUUCGUCCCUAUUUGCUUAGUCCAUGUAGCCCAUAUGGCUUAUGCAUAAAUCAUCAU------GUAUGAUGUGGCCACCGCUCCACGUCAUCAUCGUAUCAUCAUCAUCAUG ..............(((..((.((((((.....))))))))...)))......((.((------(.(((((((((........)))))))))))).))............... ( -22.40, z-score = -1.70, R) >droEre2.scaffold_4784 18108227 96 + 25762168 --UUCUGUUCUAUUUGACUAGUCCAUGCUGGCCAUAUGGCUUAUGCAUAAAUCAUCA-------GCGUGAUGUGACCACCGCUCCACGUCAUCAUC--AUCACGAUC------ --..(((.((.....)).)))...((((.((((....))))...)))).........-------.((((((((((..((........))..))).)--))))))...------ ( -22.50, z-score = -0.94, R) >droYak2.chr3L 18466775 97 + 24197627 --CUCUGUUCUAUUUGUCUAGUCCAUGCUGGCCAUAUGGCUUAUGCAUAAAUCAUCAU------GUAUGAUGUGACCACCGCUCCACGUCAUCAUC--AUCAUGAUC------ --....(..(((......)))..)((((.((((....))))...)))).....(((((------(.((((((((((...........)))).))))--)))))))).------ ( -26.30, z-score = -2.41, R) >droSec1.super_0 7962723 99 + 21120651 CUUUCUGUUCUAUUUGCCUAGUCCAUGCUGGCCAUAUGGCUUAUGCAUAAAUCAUCUU------GUAUGAUGUGACCACCGCUCCACGUCAUCAUC--AUCAUGAUC------ ......(..(((......)))..)((((.((((....))))...))))..(((((..(------(.((((((((..........))))))))...)--)..))))).------ ( -22.80, z-score = -1.59, R) >droSim1.chr3L 15200525 99 + 22553184 CUUUCUGUUCUAUUUGCCUAGUCCAUGCUGGCCAUAUGGCUUAUGCAUAAAUCAUCGU------GUAUGAUGUGACCACCGCUCCACGUCAUCAUC--AUCAUGAUC------ ......(..(((......)))..)((((.((((....))))...)))).....(((((------(.((((((((((...........)))).))))--)))))))).------ ( -25.90, z-score = -2.24, R) >droPer1.super_9 1913888 101 + 3637205 ----CACUCUGGUGUGCAUUUUCCAUGUGGGCCAUAUGGCUUAUGCAUAAAUCAUCGUGUACUCGUAUGAUGUGGCCAACUCUCCACGUCAUCGUC--AUCAUCAUC------ ----(((..((((((((((...(((((((...)))))))...))))))..))))..)))....((.(((((((((........)))))))))))..--.........------ ( -30.30, z-score = -2.62, R) >dp4.chrXR_group6 3618003 101 + 13314419 ----CACUCUGGUGUGCAUUUUCCAUGUGGGCCAUAUGGCUUAUGCAUAAAUCAUCGUGUUCUCGUAUGAUGUGGCCAACUCUCCACGUCAUCGUC--AUCAUCAUC------ ----(((..((((((((((...(((((((...)))))))...))))))..))))..)))....((.(((((((((........)))))))))))..--.........------ ( -30.30, z-score = -2.93, R) >consensus __UUCUGUUCUAUUUGCCUAGUCCAUGCUGGCCAUAUGGCUUAUGCAUAAAUCAUCGU______GUAUGAUGUGACCACCGCUCCACGUCAUCAUC__AUCAUGAUC______ ........................((((.((((....))))...))))................(.((((((((..........))))))))).................... (-16.22 = -16.12 + -0.09)

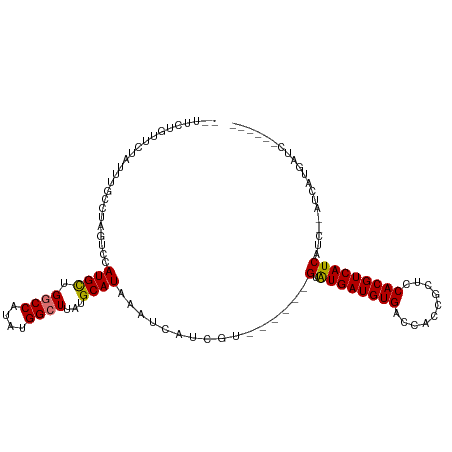
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:30:08 2011