
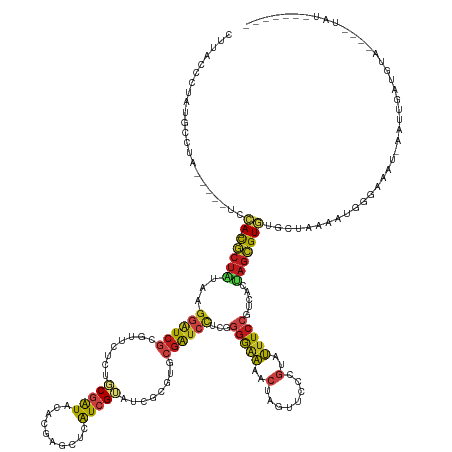
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 13,879,265 – 13,879,416 |
| Length | 151 |
| Max. P | 0.985509 |

| Location | 13,879,265 – 13,879,416 |
|---|---|
| Length | 151 |
| Sequences | 13 |
| Columns | 161 |
| Reading direction | forward |
| Mean pairwise identity | 70.79 |
| Shannon entropy | 0.65079 |
| G+C content | 0.46311 |
| Mean single sequence MFE | -41.57 |
| Consensus MFE | -14.03 |
| Energy contribution | -14.54 |
| Covariance contribution | 0.51 |
| Combinations/Pair | 1.58 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.34 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.86 |
| SVM RNA-class probability | 0.837349 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
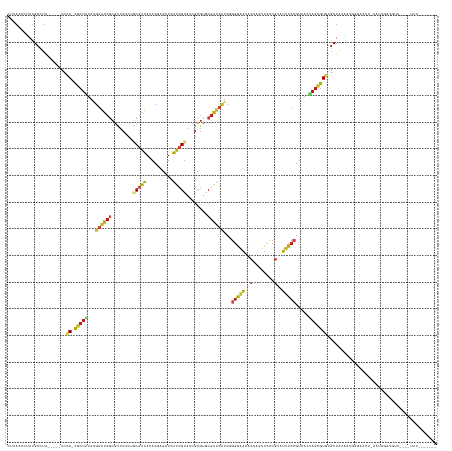
>dm3.chr3L 13879265 151 + 24543557 CUUACCCUAUGCCUA-----UCCA-CGCUAUAAGGAUCGCGUUCUCUGCGAUAGACGAGCUCAUCGUAUCGCGUGCGAUCCUCGGGGAAAACUAAUUCCCGUAUUUCCGUCACUAGCGUGUGCUAAGAUGGGAAAUUGAUUGAUGUA----UAUUAAGUUU ...........((((-----((((-(((((..(((((((((.....((((((..((((.....)))))))))))))))))))((((((.......))))))............)))))))......))))))...(((((.......----.))))).... ( -45.70, z-score = -1.96, R) >droSim1.chr3L 13234858 150 + 22553184 CUUACCCUAUGCCUA-----UCCA-CGCUAUAAGGAUCGCGUUCUCUGCGAUAGACGAGCUCAUCGUAUCGCGUGCGAUCCUCGGGGAAAACUAAUUCCCGUAUUUCCGUCACUAGCGUGUGCUAAGAUGAGAAAUUAAUUAAUGUA----UAUUAGUUU- ((((.(.((.((...-----..((-(((((..(((((((((.....((((((..((((.....)))))))))))))))))))((((((.......))))))............))))))).)))).).))))((((((((.......----.))))))))- ( -43.30, z-score = -2.31, R) >droSec1.super_0 6021718 150 + 21120651 CUUACCCUAUGCCUA-----UCCA-CGCUAUAAGGAUCGCGUUCUCUGCGAUAGACGAGCUCAUCGUAUCGCGUGCGAUCCUCGGGGAAAACUAAUUCCCGUAUUUCCGUCACUAGCGUGUGCUAAGAUGGGAAAUUAAUAGAUGUA----UAAUAGUUU- ..(((.((((.((((-----((((-(((((..(((((((((.....((((((..((((.....)))))))))))))))))))((((((.......))))))............)))))))......))))))......))))..)))----.........- ( -47.20, z-score = -2.61, R) >droYak2.chr3L 13963003 153 + 24197627 CUUACCCUAUGCCUA-----UCCA-CGCUAUAAGGAUCGCGUUCUCUGCGAUAUACGAGCUCAUCGUAUCGCGUGCGAUCCUCGGGGAAAACCAGUUCCCGUAUUUCCGUCACUAGCGUGUGCUGAAAUGGAUGAU-AAUUGAUAUAUACAUAUUAGUUU- ......((((..(..-----..((-(((((..(((((((((.....(((((..(((((.....)))))))))))))))))))((((((.......))))))............)))))))....)..)))).....-(((((((((....))))))))).- ( -45.40, z-score = -2.14, R) >droEre2.scaffold_4784 13869619 154 + 25762168 CUUACCCUAUGCCUA-----UCCA-CGCUAUAAGGAUCGCGUUCUCUGCGAUAGACGAGCUCAUCGUAUCGCGUGCGAUCCUCGGGGAAAACUAAUUCCCGUAUUUCCGUCACUAGCGUGUGCUGCAAUGGGAAAUUUAUUGAUGUAAAUAUAUUAGUUU- ....(((..(((...-----..((-(((((..(((((((((.....((((((..((((.....)))))))))))))))))))((((((.......))))))............)))))))....)))..)))......((((((((....))))))))..- ( -49.90, z-score = -3.36, R) >droAna3.scaffold_13337 11230642 144 - 23293914 CUUACCCUAUGCCUA-----UCCA-CGCUGUAAGGAUCGCGUUCCCGUCGAUACACCAACUCGUCGUAUCGCGUGCGAUCCUCGGGGAACACAAGUUCCCGGAUUUCCGUCACCAGCGUGUGCUGGAAACCGAAAA-GAUUUAUGGA---AUUU------- ......(((((...(-----((((-(((((...((((((((....((.((((((((......)).))))))))))))))))((.((((((....)))))).))..........)))))))..(.(....).)....-))).))))).---....------- ( -48.40, z-score = -2.65, R) >dp4.chrXR_group6 5699703 145 + 13314419 CUCACCCGAUGCCUG-----UCCA-CGCUAUAAGGAUCGCGUUCCCGGCGAUAGACGAGCUCAUCGUAUCGCGUGCGAUCCUCGGGGAACACAAGUUCUCGGAUCUCCGUCACUAGCGUGUGCUGGUAGGGGUAUAACAAUCAGAUAAAUG---------- ...((((..((((.(-----..((-(((((...((((((((....).(((((..((((.....)))))))))..)))))))((.((((((....)))))).))..........)))))))..).)))).))))..................---------- ( -54.70, z-score = -2.59, R) >droWil1.scaffold_180949 3471344 140 - 6375548 CUUACCCUAUGCCUA-----UCCA-CGCUAUAGGGAUCUCGUUCUCUUCGAUAAACCAACUCAUCGUAGCGUGUUCGAUCCUCGGGAAAAACGAGUUCACGAAUUUCCGUCACUAGCGUAUGCUGGCAAAUUAA------UUAUGGG---AUUUU------ ....(((..((((..-----...(-(((((..((((..((((.....((((...(((.((.....)).).))..))))..((((.......))))...))))..)))).....)))))).....))))......------....)))---.....------ ( -32.30, z-score = -0.52, R) >droGri2.scaffold_15110 9203137 147 - 24565398 CUUACCCUAUGCCUACU--AUCCA-CGCUAUAUGGAUCGCGUUCUCGGCGAUACACAAGCUCAUCGUAUCGUGUACGAUCCUCCGGAAAGACGAGCUCUCGAAUUUCCGUCACUAGCGUGUGCUAAGGAUAAAGA---AACCAUAAAAAGUUU-------- ......((.(((((...--...((-(((((...((((((((....))(((((((...........)))))))...))))))..((((((..(((....)))..))))))....))))))).....))).)).)).---...............-------- ( -38.80, z-score = -1.74, R) >droVir3.scaffold_13049 13881302 144 + 25233164 CUUACCCUAUGCCUA-----UCCA-CGCUAUAUGGAUCGCGUUCUCUGCGAUACACAAGCUCAUCGUAACGUGUACGAUCCUCCGGAAAAACGAGCUCACGAAUUUCCGUCACUAGCGUGUGCUAGAAAGGAAAU---AUAUAGACAUAGUUU-------- ......(((((.(((-----(...-((.....((.((((((.....))))))...))(((((.(((((.....)))))(((...))).....)))))..)).((((((....(((((....)))))...))))))---..)))).)))))...-------- ( -37.20, z-score = -1.67, R) >droMoj3.scaffold_6680 11316452 129 - 24764193 CUUACCCUAUGCCUA-----UCCA-CGCUAUAUGGAUCGCGCUCACGGCGAUACACUAGCUCAUCGUAACGUGUACGAUCCUCCGGAAAGACGAGCUCUCGAAUUUCCGUCACAAGCGUGUGCUAAUGAUCGAUU-------------------------- ...............-----..((-((((....((((((....(((((((((..........)))))..))))..))))))..((((((..(((....)))..)))))).....))))))...............-------------------------- ( -35.40, z-score = -1.28, R) >anoGam1.chr3L 8846180 160 + 41284009 CUUACCCUAUGCCUAUCUAGUGGAUCACCGUAGACGUCUCUACCUCUCCUAUAUACAAGUUCAUCGUAACGCGUUCUGUCCUCGGGGAAAAUUAAUUCCCUUAUUUCGGUCACUAACGUGUGCUGUGUGUGGUGAAAAGGAAAGUGGCAACGUGCCGUUG- .....(((...((........)).((((((((((....))))).......(((((((.((........(((((((.((.((..((((((......))))))......)).))..)))))))))))))))))))))..))).....(((.....)))....- ( -40.20, z-score = -0.76, R) >triCas2.ChLG3 15463946 133 - 32080666 ----CUCUAGGAUCU-----CUUA-UUCUAGAAGUUUCGACAUCCCUUCUGUACACUAAUUCGUCGAAUCUUACGCGAUCUUCGAUGGCUACUAUCUCCCUUAGUUCGGUCAUAAGAGUAUACUAAAAUUCUUCAUCAAUAUU------------------ ----.((((((((..-----...)-)))))))((.((((((.....................)))))).)).....(((.....((((((((((.......))))..))))))((((((........)))))).)))......------------------ ( -21.90, z-score = -0.13, R) >consensus CUUACCCUAUGCCUA_____UCCA_CGCUAUAAGGAUCGCGUUCUCUGCGAUACACGAGCUCAUCGUAUCGCGUGCGAUCCUCGGGGAAAACUAGUUCCCGUAUUUCCGUCACUAGCGUGUGCUAAAAUGGGAAAU_AAUUGAUGUA____UAU_______ ......................((.(((((...((((((........(((((..........)))))........))))))...(((((..(........)..))))).....)))))))......................................... (-14.03 = -14.54 + 0.51)

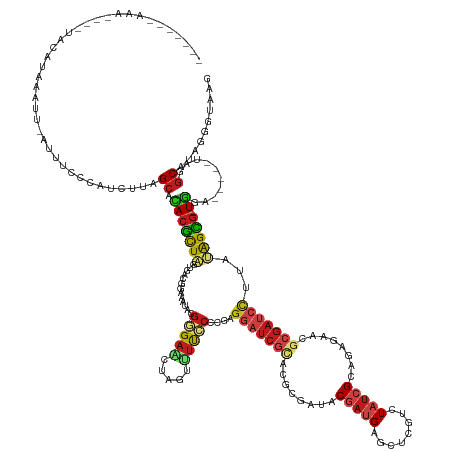
| Location | 13,879,265 – 13,879,416 |
|---|---|
| Length | 151 |
| Sequences | 13 |
| Columns | 161 |
| Reading direction | reverse |
| Mean pairwise identity | 70.79 |
| Shannon entropy | 0.65079 |
| G+C content | 0.46311 |
| Mean single sequence MFE | -45.99 |
| Consensus MFE | -16.58 |
| Energy contribution | -17.58 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.54 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.36 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.20 |
| SVM RNA-class probability | 0.985509 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
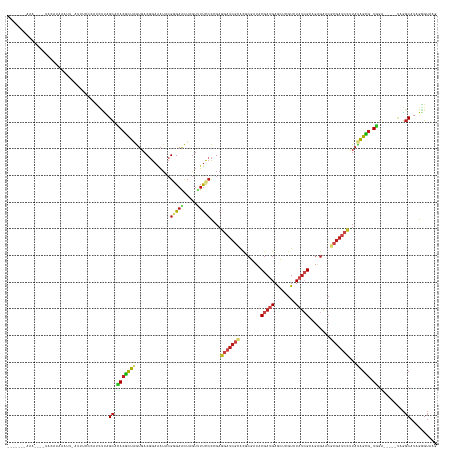
>dm3.chr3L 13879265 151 - 24543557 AAACUUAAUA----UACAUCAAUCAAUUUCCCAUCUUAGCACACGCUAGUGACGGAAAUACGGGAAUUAGUUUUCCCCGAGGAUCGCACGCGAUACGAUGAGCUCGUCUAUCGCAGAGAACGCGAUCCUUAUAGCG-UGGA-----UAGGCAUAGGGUAAG ..........----...............(((((((((...(((((((.............(((((......))))).(((((((((..((((((.((((....)))))))))).(....)))))))))).)))))-))..-----)))).)).))).... ( -51.00, z-score = -4.09, R) >droSim1.chr3L 13234858 150 - 22553184 -AAACUAAUA----UACAUUAAUUAAUUUCUCAUCUUAGCACACGCUAGUGACGGAAAUACGGGAAUUAGUUUUCCCCGAGGAUCGCACGCGAUACGAUGAGCUCGUCUAUCGCAGAGAACGCGAUCCUUAUAGCG-UGGA-----UAGGCAUAGGGUAAG -.........----..................((((((((.(((((((.............(((((......))))).(((((((((..((((((.((((....)))))))))).(....)))))))))).)))))-))..-----...)).))))))... ( -49.80, z-score = -3.90, R) >droSec1.super_0 6021718 150 - 21120651 -AAACUAUUA----UACAUCUAUUAAUUUCCCAUCUUAGCACACGCUAGUGACGGAAAUACGGGAAUUAGUUUUCCCCGAGGAUCGCACGCGAUACGAUGAGCUCGUCUAUCGCAGAGAACGCGAUCCUUAUAGCG-UGGA-----UAGGCAUAGGGUAAG -.........----...............(((((((((...(((((((.............(((((......))))).(((((((((..((((((.((((....)))))))))).(....)))))))))).)))))-))..-----)))).)).))).... ( -51.00, z-score = -3.87, R) >droYak2.chr3L 13963003 153 - 24197627 -AAACUAAUAUGUAUAUAUCAAUU-AUCAUCCAUUUCAGCACACGCUAGUGACGGAAAUACGGGAACUGGUUUUCCCCGAGGAUCGCACGCGAUACGAUGAGCUCGUAUAUCGCAGAGAACGCGAUCCUUAUAGCG-UGGA-----UAGGCAUAGGGUAAG -..(((..(((((...((((....-......(......)..(((((((.............(((((......))))).(((((((((..(((((((((.....)))))..)))).(....)))))))))).)))))-))))-----)).))))).)))... ( -45.60, z-score = -2.05, R) >droEre2.scaffold_4784 13869619 154 - 25762168 -AAACUAAUAUAUUUACAUCAAUAAAUUUCCCAUUGCAGCACACGCUAGUGACGGAAAUACGGGAAUUAGUUUUCCCCGAGGAUCGCACGCGAUACGAUGAGCUCGUCUAUCGCAGAGAACGCGAUCCUUAUAGCG-UGGA-----UAGGCAUAGGGUAAG -............................(((..(((....(((((((.............(((((......))))).(((((((((..((((((.((((....)))))))))).(....)))))))))).)))))-))..-----...)))..))).... ( -52.70, z-score = -4.50, R) >droAna3.scaffold_13337 11230642 144 + 23293914 -------AAAU---UCCAUAAAUC-UUUUCGGUUUCCAGCACACGCUGGUGACGGAAAUCCGGGAACUUGUGUUCCCCGAGGAUCGCACGCGAUACGACGAGUUGGUGUAUCGACGGGAACGCGAUCCUUACAGCG-UGGA-----UAGGCAUAGGGUAAG -------....---(((.......-..(((.(((.(((((....))))).))).)))((((((((((....)))))).(((((((((..(((((((.((......)))))))).)(....))))))))))......-.)))-----).......))).... ( -55.10, z-score = -2.96, R) >dp4.chrXR_group6 5699703 145 - 13314419 ----------CAUUUAUCUGAUUGUUAUACCCCUACCAGCACACGCUAGUGACGGAGAUCCGAGAACUUGUGUUCCCCGAGGAUCGCACGCGAUACGAUGAGCUCGUCUAUCGCCGGGAACGCGAUCCUUAUAGCG-UGGA-----CAGGCAUCGGGUGAG ----------.................(((((...((.(..(((((((....(((....))).((((....))))...(((((((((..((((((.((((....)))))))))).(....)))))))))).)))))-))..-----).))....))))).. ( -54.80, z-score = -2.81, R) >droWil1.scaffold_180949 3471344 140 + 6375548 ------AAAAU---CCCAUAA------UUAAUUUGCCAGCAUACGCUAGUGACGGAAAUUCGUGAACUCGUUUUUCCCGAGGAUCGAACACGCUACGAUGAGUUGGUUUAUCGAAGAGAACGAGAUCCCUAUAGCG-UGGA-----UAGGCAUAGGGUAAG ------.....---(((....------......((((....(((((((.....(((((..((......))..)))))...(((((.......((.(((((((....))))))).)).......)))))...)))))-))..-----..))))..))).... ( -38.14, z-score = -1.45, R) >droGri2.scaffold_15110 9203137 147 + 24565398 --------AAACUUUUUAUGGUU---UCUUUAUCCUUAGCACACGCUAGUGACGGAAAUUCGAGAGCUCGUCUUUCCGGAGGAUCGUACACGAUACGAUGAGCUUGUGUAUCGCCGAGAACGCGAUCCAUAUAGCG-UGGAU--AGUAGGCAUAGGGUAAG --------(((((......))))---)..(((((((..((.(((((((....((((((..(((....)))..))))))..(((((((...(((((((.........)))))))..(....))))))))...)))))-))...--.....))..))))))). ( -45.90, z-score = -1.70, R) >droVir3.scaffold_13049 13881302 144 - 25233164 --------AAACUAUGUCUAUAU---AUUUCCUUUCUAGCACACGCUAGUGACGGAAAUUCGUGAGCUCGUUUUUCCGGAGGAUCGUACACGUUACGAUGAGCUUGUGUAUCGCAGAGAACGCGAUCCAUAUAGCG-UGGA-----UAGGCAUAGGGUAAG --------...((((((((((..---...(((((.(((((....)))))...((((((..((......))..))))))))))).....(((((((..(((.(.((((((.((.....)))))))).)))).)))))-)).)-----)))))))))...... ( -49.70, z-score = -3.18, R) >droMoj3.scaffold_6680 11316452 129 + 24764193 --------------------------AAUCGAUCAUUAGCACACGCUUGUGACGGAAAUUCGAGAGCUCGUCUUUCCGGAGGAUCGUACACGUUACGAUGAGCUAGUGUAUCGCCGUGAGCGCGAUCCAUAUAGCG-UGGA-----UAGGCAUAGGGUAAG --------------------------....((((((((((.(((....))).((((((..(((....)))..))))))....((((((.....))))))..))))))).)))(((.((.((.(.((((((.....)-))))-----).))).)).)))... ( -43.20, z-score = -1.44, R) >anoGam1.chr3L 8846180 160 - 41284009 -CAACGGCACGUUGCCACUUUCCUUUUCACCACACACAGCACACGUUAGUGACCGAAAUAAGGGAAUUAAUUUUCCCCGAGGACAGAACGCGUUACGAUGAACUUGUAUAUAGGAGAGGUAGAGACGUCUACGGUGAUCCACUAGAUAGGCAUAGGGUAAG -.....((....((((.(((((((((((............((.((((..((.((.......(((((......)))))...)).)).)))).))(((((.....)))))....))))))).))))..(((((.((....))..))))).))))....))... ( -37.20, z-score = -0.16, R) >triCas2.ChLG3 15463946 133 + 32080666 ------------------AAUAUUGAUGAAGAAUUUUAGUAUACUCUUAUGACCGAACUAAGGGAGAUAGUAGCCAUCGAAGAUCGCGUAAGAUUCGACGAAUUAGUGUACAGAAGGGAUGUCGAAACUUCUAGAA-UAAG-----AGAUCCUAGAG---- ------------------.((((((((...((((((((.....((((((.(......)))))))........((.(((...))).)).)))))))).....))))))))......(((((.((.............-...)-----).)))))....---- ( -23.79, z-score = 0.94, R) >consensus _______AAA____UACAUAAAUU_AUUUCCCAUCUUAGCACACGCUAGUGACGGAAAUACGGGAACUAGUUUUCCCCGAGGAUCGCACGCGAUACGAUGAGCUCGUCUAUCGCAGAGAACGCGAUCCUUAUAGCG_UGGA_____UAGGCAUAGGGUAAG .........................................(((((((.............(((((.....)))))....(((((((........(((((........)))))........)))))))...))))).))...................... (-16.58 = -17.58 + 1.00)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:24:42 2011