
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 13,607,178 – 13,607,295 |
| Length | 117 |
| Max. P | 0.986105 |

| Location | 13,607,178 – 13,607,295 |
|---|---|
| Length | 117 |
| Sequences | 11 |
| Columns | 136 |
| Reading direction | reverse |
| Mean pairwise identity | 72.12 |
| Shannon entropy | 0.55472 |
| G+C content | 0.52530 |
| Mean single sequence MFE | -40.45 |
| Consensus MFE | -23.53 |
| Energy contribution | -23.82 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.58 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.23 |
| SVM RNA-class probability | 0.986105 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
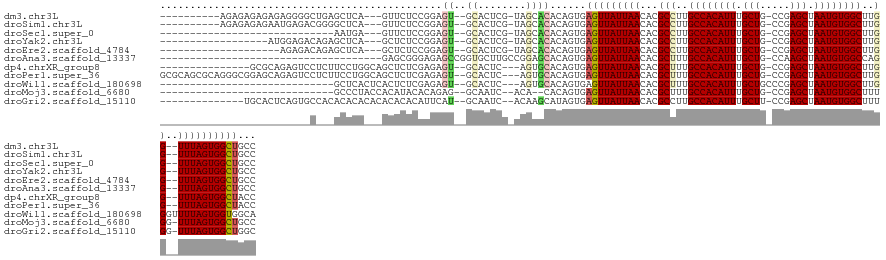
>dm3.chr3L 13607178 117 - 24543557 ----------AGAGAGAGAGAGGGGCUGAGCUCA---GUUCUCCGGAGU--GCACUCG-UAGCACACAGUGAGUUAUUAACACGCCUUGCCACAUUUGCUG-CCGAGCUAAUGUGGCUUGG--UUUAGUGGCUGCC ----------...(.(((((..((((...)))).---.)))))).(((.--...)))(-((((.(((.(((.........)))(((..((((((((.(((.-...))).))))))))..))--)...)))))))). ( -45.70, z-score = -1.83, R) >droSim1.chr3L 12990695 117 - 22553184 ----------AGAGAGAGAAUGAGACGGGGCUCA---GUUCUCCGGAGU--GCACUCG-UAGCACACAGUGAGUUAUUAACACGCCUUGCCACAUUUGCUG-CCGAGCUAAUGUGGCUUGG--UUUAGUGGCUGCC ----------...(.(((((((((......))))---.)))))).(((.--...)))(-((((.(((.(((.........)))(((..((((((((.(((.-...))).))))))))..))--)...)))))))). ( -44.30, z-score = -2.02, R) >droSec1.super_0 5768900 98 - 21120651 -----------------------------AAUGA---GUUCUCCGGAGU--GCACUCG-UAGCACACAGUGAGUUAUUAACACGCCUUGCCACAUUUGCUG-CCGAGCUAAUGUGGCUUGG--UUUAGUGGCUGCC -----------------------------...((---((.(........--).))))(-((((.(((.(((.........)))(((..((((((((.(((.-...))).))))))))..))--)...)))))))). ( -36.50, z-score = -2.42, R) >droYak2.chr3L 13700136 109 - 24197627 ------------------AUGGAGACAGAGCUCA---GCUCUCCGGAGU--GCACUCG-UAGCACACAGUGAGUUAUUAACACGCCUUGCCACAUUUGCUG-CCGAGCUAAUGUGGCUUGG--UUUAGUGGCUGCC ------------------.((....))(((....---((((....))))--...)))(-((((.(((.(((.........)))(((..((((((((.(((.-...))).))))))))..))--)...)))))))). ( -42.60, z-score = -2.20, R) >droEre2.scaffold_4784 13608032 107 - 25762168 --------------------AGAGACAGAGCUCA---GCUCUCCGGAGU--GCACUCG-UAGCACACAGUGAGUUAUUAACACGCCUUGCCACAUUUGCUG-CCGAGCUAAUGUGGCUUGG--UUUAGUGGCUGCC --------------------.......(((....---((((....))))--...)))(-((((.(((.(((.........)))(((..((((((((.(((.-...))).))))))))..))--)...)))))))). ( -40.10, z-score = -1.72, R) >droAna3.scaffold_13337 1459709 96 + 23293914 -------------------------------------GAGCGGGAGAGCCGGUGCUUGCCGGAGCACAGUGAGUUAUUAACACGCUUUGCCACAUUUGCUG-CCAAGCUAAUGUGGCCAGG--UUUAGUGGCUGCC -------------------------------------..((....(..(((((....)))))..)......(((((((((...((((.((((((((.(((.-...))).)))))))).)))--)))))))))))). ( -37.40, z-score = -1.34, R) >dp4.chrXR_group8 2386521 113 + 9212921 ---------------GCGCAGAGUCCUCUUCCUGGCAGCUCUCGAGAGU--GCACUC---AGUGCACAGUGAGUUAUUAACACGCUUUGCCACAUUUGCUG-CCGAGCUAAUGUGGCUUGG--UUUAGUGGCUACC ---------------.((((((((((.......))..))))).....((--((((..---.)))))).)))(((((((((...(((..((((((((.(((.-...))).))))))))..))--))))))))))... ( -41.40, z-score = -1.47, R) >droPer1.super_36 480842 128 - 818889 GCGCAGCGCAGGGCGGAGCAGAGUCCUCUUCCUGGCAGCUCUCGAGAGU--GCACUC---AGUGCACAGUGAGUUAUUAACACGCUUUGCCACAUUUGCUG-CCGAGCUAAUGUGGCUUGG--UUUAGUGGCUACC .((((((((.(((.((((.......)))).))).)).))).......((--((((..---.)))))).)))(((((((((...(((..((((((((.(((.-...))).))))))))..))--))))))))))... ( -48.30, z-score = -0.42, R) >droWil1.scaffold_180698 10806086 102 + 11422946 -----------------------------GCUCACUCACUCUCGAGAGU--GCACUC---AGUGCACAGUGAGUUAUUAACACGCUUUGCCACAUUUGCUGCCCGAGCUAAUGUGGCUUGGGUUUUAGUGGUGGCA -----------------------------(((((((..((....)).((--((((..---.))))))))))))).............((((((.......(((((((((.....))))))))).......)))))) ( -41.14, z-score = -2.90, R) >droMoj3.scaffold_6680 17242282 99 + 24764193 -----------------------------GCCCUACCACAUACACAGAG--GCAAUC--ACA--CACAGUGAGUUAUUAACACGCUUUGCCACAUUUGCUG-CCGAGCUAAUGUGGCUUUGG-UUUAGUGGCUGCC -----------------------------(((((((((........(((--((..((--((.--....))))((.....))..)))))((((((((.(((.-...))).))))))))..)))-)..)).))).... ( -32.00, z-score = -1.96, R) >droGri2.scaffold_15110 17547755 116 - 24565398 --------------UGCACUCAGUGCCACACACACACACACACAUUCAU--GCAAUC--ACAAGCAUAGUGAGUUAUUAACACGCCUUGCCACAUUUGCUU-CCGAGCUAAUGUGGCUUUGG-UUUAGUGGCUGGC --------------..........(((...............((((.((--((....--....))))))))(((((((((...(((..((((((((.(((.-...))).))))))))...))-))))))))))))) ( -35.50, z-score = -1.76, R) >consensus _________________________C___GCUCA___GCUCUCCGGAGU__GCACUCG_UAGCACACAGUGAGUUAUUAACACGCCUUGCCACAUUUGCUG_CCGAGCUAAUGUGGCUUGG__UUUAGUGGCUGCC .............................................((((....))))..............(((((((((....((..((((((((.(((.....))).))))))))..))...)))))))))... (-23.53 = -23.82 + 0.28)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:23:50 2011