| Sequence ID | dm3.chr3L |
|---|---|
| Location | 13,035,868 – 13,036,003 |
| Length | 135 |
| Max. P | 0.965545 |

| Location | 13,035,868 – 13,036,003 |
|---|---|
| Length | 135 |
| Sequences | 12 |
| Columns | 146 |
| Reading direction | forward |
| Mean pairwise identity | 81.50 |
| Shannon entropy | 0.37477 |
| G+C content | 0.41109 |
| Mean single sequence MFE | -35.28 |
| Consensus MFE | -26.67 |
| Energy contribution | -24.19 |
| Covariance contribution | -2.48 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.76 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.75 |
| SVM RNA-class probability | 0.965545 |
| Prediction | RNA |
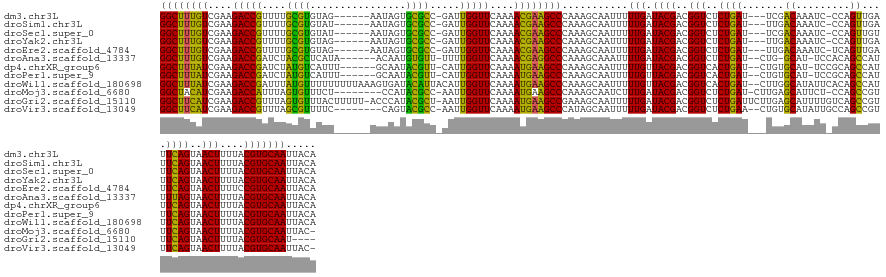
Download alignment: ClustalW | MAF
>dm3.chr3L 13035868 135 + 24543557 GGCUUUGUCGAAGACCGUUUUGCGUGUAG------AAUAGUGCGCC-GAUUGGUUCAAAACGAAGCCCAAAGCAAUUUUUGAUACGACGGUCUCUGAU---UCGACAAAUC-CCAGUUGAUUCAGUAACUUUUACGUGCAAUUACA ((((((((....(((((.((.(((..(..------....)..))).-)).)))))....))))))))...........(((.((((..(((..((((.---(((((.....-...))))).))))..)))....)))))))..... ( -38.90, z-score = -2.21, R) >droSim1.chr3L 12427813 135 + 22553184 GGCUUUGUCGAAGACCGUUUUGCGUGUAU------AAUAGUGCGCC-GAUUGGUUCAAAACGAAGCCCAAAGCAAUUUUUGAUACGACGGUCUCUGAU---UUGACAAAUC-CCAGUUGAUUCAGUAACUUUUACGUGCAAUUACA ((((((((....(((((..(((.((((((------....)))))))-)).)))))....))))))))...........(((.((((..(((..((((.---(..((.....-...))..).))))..)))....)))))))..... ( -37.00, z-score = -2.02, R) >droSec1.super_0 5207835 135 + 21120651 GGCUUUGUCGAAGACCGUUUUGCGUGUAU------AAUAGUGCGCC-GAUUGGUUCAAAACGAAGCCCAAAGCAAUUUUUGAUACGACGGUCUCUGAU---UCGACAAAUC-CCAGUUGUUUCAGUAACUUUUACGUGCAAUUACA ((((((((....(((((..(((.((((((------....)))))))-)).)))))....))))))))...........(((.((((..(((..((((.---.((((.....-...))))..))))..)))....)))))))..... ( -38.00, z-score = -2.17, R) >droYak2.chr3L 13117106 135 + 24197627 GGCUUUGUCGAAGACCGUUUUGCGUGUAG------AAUAGUGCGCC-GAUUGGUUCAAAACGAAGCCCAAAGCAAUUUUUGAUACGACGGUCUCUGAU---UUGACAAAUC-CCAGUUGAUUCAGUAACUUUUACGUGCAAUUACA ((((((((....(((((.((.(((..(..------....)..))).-)).)))))....))))))))...........(((.((((..(((..((((.---(..((.....-...))..).))))..)))....)))))))..... ( -36.70, z-score = -1.69, R) >droEre2.scaffold_4784 13043861 135 + 25762168 GGCUUUGUCGAAGACCGUUUUGCGUGUAG------AAUAGUGCGCC-GAUUGGUUCAAAACGAAGCCCAAAGCAAUUUUUGAUACGACGGUCUCUGAU---UUGACAAAUC-UCAGUUGAUUCAGUAACUUUUCCGUGCAAUUACA ((((((((....(((((.((.(((..(..------....)..))).-)).)))))....))))))))...........(((.((((..(((..((((.---(..((.....-...))..).))))..)))....)))))))..... ( -36.70, z-score = -1.44, R) >droAna3.scaffold_13337 10857502 135 - 23293914 GGCUUUGUCGAAGACCGAUCUACGCUCAUA------ACAAUGUGUU-UUUUGGUUCAAAACGAGGCCCAAAGCAAAUUUUGAUACGACGGUCUCUGAU--CUG-GCAU-UCCACAGCCAUUUUAGUAACUUUUACGUGCAAUUACA ((((((((....((((((...((((.....------.....)))).-..))))))....))))))))...........(((.((((..(((..((((.--.((-((..-......))))..))))..)))....)))))))..... ( -35.70, z-score = -2.30, R) >dp4.chrXR_group6 5306739 136 + 13314419 GGCUUUAUCGAAGACCGAUCUAUGUCAUUU------GCAAUACGUU-CAUUGGUUCAAAAUGAAGCCCAAAGCAAUUUUUGUUACGACGGUCACUGAU--CUGUGCAU-UCCGCAGCCAUUUCAGUAACUUUUACGUGCAAUUACA ((((((((....(((((((..((((.....------.....)))).-.)))))))....))))))))...........((((.(((..(((.(((((.--.((.((..-......))))..))))).)))....)))))))..... ( -32.20, z-score = -1.40, R) >droPer1.super_9 3617052 136 + 3637205 GGCUUUAUCGAAGACCGAUCUAUGUCAUUU------GCAAUACGUU-CAUUGGUUCAAAAUGAAGCCCAAAGCAAUUUUUGUUACGACGGUCACUGAU--CUGUGCAU-UCCGCAGCCAUUUCAGUAACUUUUACGUGCAAUUACA ((((((((....(((((((..((((.....------.....)))).-.)))))))....))))))))...........((((.(((..(((.(((((.--.((.((..-......))))..))))).)))....)))))))..... ( -32.20, z-score = -1.40, R) >droWil1.scaffold_180698 6507099 144 + 11422946 GGCUUUAUCGAAGACCGAUUUAUGUUUUUUUUUAAAGUGAUACAUUACAUUGGUUCAAAAUGAAGCCCAAAGCAAUUUUUGUUACGACGGUCACUGAU--CUUGGCAUAUUCACAGCCAUUUCAGUAACUUUUACGUGCAAUUACA ((((((((....(((((((..((((.((..........)).))))...)))))))....))))))))...........((((.(((..(((.(((((.--..((((.........))))..))))).)))....)))))))..... ( -34.20, z-score = -2.79, R) >droMoj3.scaffold_6680 9695089 134 - 24764193 GGCUACAUCGAAGACCAUUUAGUGUUUCU--------CCAUACGCC-AAUUGGUUCAAAAUGAAGCCCAAAGCAAUCUUUGAUACGACGGUCUCUGAU-CUUGAGCAUUCU-CCAGCCGUUUCAGUAACUUUUACGUGCAAUUAC- ((((.(((....(((((.((.((((....--------....)))).-)).)))))....))).))))(((((....))))).((((..(((..((((.-..((.((.....-...))))..))))..)))....)))).......- ( -29.40, z-score = -1.08, R) >droGri2.scaffold_15110 22984792 140 + 24565398 GGCUUCAUCGAAGACCGUUUAGUGUUUACUUUUU-ACCCAUACGCU-AAUUGGUUCAAAAUGAAGCCGAAAGCAAUUUUUGAUACGACGGUCUCUGAUUCUUGAGCAUUUUGUCAGCCGUUUCAGUAACUUUUACGUGCAAU---- ((((((((....(((((.(((((((.........-......)))))-)).)))))....))))))))....(((.....(((...(((((...(((((.............)))))))))))))(((.....))).)))...---- ( -36.28, z-score = -2.30, R) >droVir3.scaffold_13049 12399163 134 + 25233164 GGCUUCAUCGAAGACCGUUUAGCGUUUUC--------CAGUACGCC-AAUUGGUUCAAAAUGAAGCCCAUAGCAAUUUUUGAUACGACGGUCUCUGAA--CUGUGCAUAUUGCCAGCCGUUUCAGUAACUUUUACGUGCAAUUAC- ((((((((....(((((.((.((((....--------....)))).-)).)))))....))))))))...........(((.((((..(((..(((((--(((.((.....)))))....)))))..)))....)))))))....- ( -36.10, z-score = -2.25, R) >consensus GGCUUUAUCGAAGACCGUUUUGCGUGUAU______AACAAUACGCC_GAUUGGUUCAAAACGAAGCCCAAAGCAAUUUUUGAUACGACGGUCUCUGAU___UGGGCAUAUC_CCAGCCGUUUCAGUAACUUUUACGUGCAAUUACA ((((((((....(((((....((((................)))).....)))))....))))))))...........(((.((((..(((..((((.......((.........))....))))..)))....)))))))..... (-26.67 = -24.19 + -2.48)
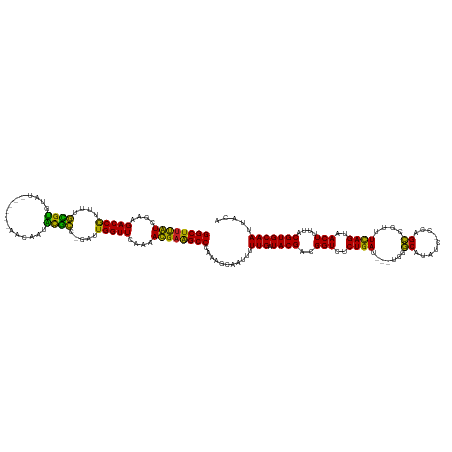
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:22:38 2011