
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 13,013,595 – 13,013,737 |
| Length | 142 |
| Max. P | 0.996852 |

| Location | 13,013,595 – 13,013,706 |
|---|---|
| Length | 111 |
| Sequences | 14 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 74.30 |
| Shannon entropy | 0.57990 |
| G+C content | 0.58658 |
| Mean single sequence MFE | -42.25 |
| Consensus MFE | -15.56 |
| Energy contribution | -15.57 |
| Covariance contribution | 0.01 |
| Combinations/Pair | 1.83 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.37 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.623166 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
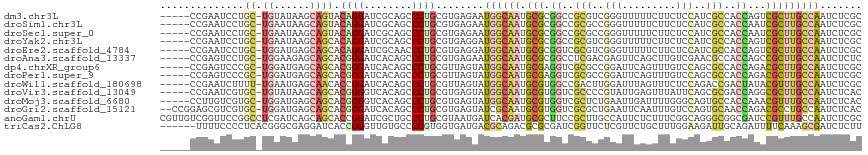
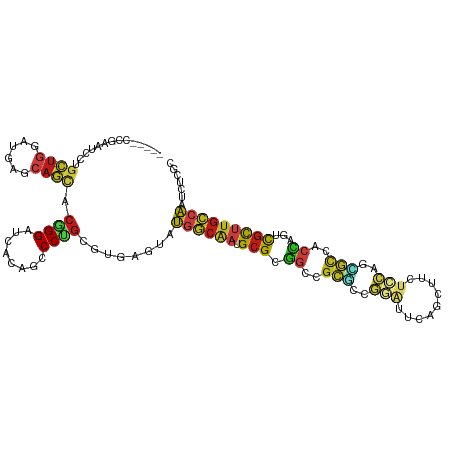
>dm3.chr3L 13013595 111 + 24543557 -----CCGAAUCCUGC-UGUAUAAGCAGUACAGGAUCGCAGCCCUGCGUGAGAAUGGCAAUGCGCGGCCGCGUCGGGUUUUUCUUCUCCAUCGCCACCAGUCGCUUGCCAAUCUCGC -----..((.((((((-(((....))))..)))))))(((....)))((((((.((((((.((((((..(((..(((.........)))..)))..)).).))))))))).)))))) ( -43.50, z-score = -2.73, R) >droSim1.chr3L 12401031 111 + 22553184 -----CCGAAUCCUGC-UGAAUAAGCAGUACAGGAUCGCAGCCCUGCGUGAGAAUGGCAAUGCGCGGCCGCGCCGGGUUUUUCUUCUCCAUCGCCACCAAUCGCUUGCCAAUCUCGC -----..((.((((((-((......)))..)))))))(((....)))((((((.((((((.(((.((..(((..(((.........)))..)))..))...))))))))).)))))) ( -39.60, z-score = -1.54, R) >droSec1.super_0 5185180 111 + 21120651 -----CCGAAUCCUGC-UGAAUAAGCAGUACAGGAUCGCAGCCCUGCGUGAGAAUGGCAAUGCGCGGCCGCGCCGGGUUUUUCUUCUCCAUCGCCACCAAUCGCUUGCCAAUCUCGC -----..((.((((((-((......)))..)))))))(((....)))((((((.((((((.(((.((..(((..(((.........)))..)))..))...))))))))).)))))) ( -39.60, z-score = -1.54, R) >droYak2.chr3L 13092498 111 + 24197627 -----CCGAAUCCUGC-UGAAUAAGCAGCACAGGAUCGCAGCCCUGCGUGAGGAUGGCAAUGCGCGGCCGCGUCGGGUUUUUCUUCUCCAUCGCCACCAGUCGCUUGCCAAUCUCGC -----..((.((((((-((......))))..))))))(((....)))((((((.((((((.((((((..(((..(((.........)))..)))..)).).))))))))).)))))) ( -41.90, z-score = -1.31, R) >droEre2.scaffold_4784 13021128 111 + 25762168 -----CCGAAUCCUGC-UGGAUGAGCAGCACAGGAUCGCAACCCUGCGUGAGGAUGGCAAUGCGCGGUCGCGUCGGGUUUUUCUUCUCCAUCGCCACCAGUCGCUUGCCAAUCUCGC -----..((.((((((-((......))))..))))))(((....)))((((((.((((((.(((((((.(((..(((.........)))..))).))).).))))))))).)))))) ( -43.70, z-score = -1.58, R) >droAna3.scaffold_13337 19156351 111 - 23293914 -----CCGAGUCCUGC-UGGAAGAGCAGCACGGGAUCACAGCCCUGCGUGAGAAUGGCAAUGCGCGGCCUCGACGAGUUCAGCUUGUCGAACGCCACCAGCCGCUUGCCAAUCUCUC -----..((..(((((-((......))))).))..))............((((.((((((.(((((((.((((((((.....))))))))..)))....).))))))))).)))).. ( -44.60, z-score = -1.63, R) >dp4.chrXR_group6 4377285 111 + 13314419 -----CCGAGUCCCGC-UGGAUGAGCAGCACGGGAUCACAGCCCUGCGUUAGUAUGGCAAUGCGAGGUCGCGCCGGAUUCAGUUUGUCCAGCGCCACCAGACGCUUGCCAAUCUCGC -----.((((((((((-((......))))..))))...................((((((.(((.(((.((((.((((.......)))).)))).)))...)))))))))..)))). ( -42.80, z-score = -1.18, R) >droPer1.super_9 2666466 111 + 3637205 -----CCGAGUCCCGC-UGGAUGAGCAGCACGGGAUCACAGCCCUGCGUUAGUAUGGCAAUGCGAGGUCGCGCCGGAUUCAGUUUGUCCAGCGCCACCAGACGCUUGCCAAUCUCGC -----.((((((((((-((......))))..))))...................((((((.(((.(((.((((.((((.......)))).)))).)))...)))))))))..)))). ( -42.80, z-score = -1.18, R) >droWil1.scaffold_180698 6146028 111 - 11422946 -----CCGAAUCUUUU-UGAAUGAGCAACACCGGAUCACAGCCCUGCGUUAGUAUGGCAAUGCGUGGCCGACUUGGAUUUAGUUUCUCCAGACCGACUAUACGUUUGCCAAUCUCGC -----.((((....))-))...(((.(((.(.((........)).).)))....((((((.(((((..((..(((((.........)))))..))....)))))))))))..))).. ( -23.10, z-score = 0.91, R) >droVir3.scaffold_13049 2740841 111 - 25233164 -----CCGAAUCGUGC-UGUAUAAGCAGCACGGGGUCACAGCCCUGCGUGAGUAUGGCAAUGCGUGGUCGCCCCGUAUUGAGUUUAUUCAGCGCGACCAGGCGCUUGCCAAUCUCAC -----......(((((-(((....))))))))((((....))))...(((((..(((((((((.(((((((...((..((((....))))))))))))).))).))))))..))))) ( -51.30, z-score = -4.27, R) >droMoj3.scaffold_6680 7664310 111 - 24764193 -----CCUUGUCGUGC-UGGAUGAGCAGCACGGGGUCACAGCCCUGCGUGAGUAUGGCAAUGCGUGGUCGCUCUGAAUUGAUUUUGGGCAGUGCCACCAAACGUUUGCCAAUCUCAC -----......(((((-((......)))))))((((....))))...(((((..((((((.(((((((.((.(((..(.......)..))).)).)))..))))))))))..))))) ( -43.70, z-score = -1.65, R) >droGri2.scaffold_15121 75770 114 - 279557 --CCGGAGCGUCGUGC-UGGAUGAGCAGCACGGGAUCACAGCCCUGCGUGAGUAUCGCAAUGCGUGGUCGCGCUGAAUUCAAUUUGUCCAGUGCAACCAGACGCCUGCCAAUCUCAC --.(((.(((((((((-((......))))))((((((((.((..((((.......))))..))))))))((((((.((.......)).))))))..)).)))))))).......... ( -47.40, z-score = -2.39, R) >anoGam1.chrU 23959213 117 - 59568033 CGUUGUCGGUUCCGGCCUCGAUCAGCAGCACCGGAUCGCUGCCCUGCGUAAUGAUCACGAUGCGCUUCCGCUUGCCAUUCUCUUUCGGCAGGGCGGCGAUCCGUUUGCCAAUCUCGC .(..((((....))))..)(((..((((...((((((((((((((((....(((....((((.((........)))))).....))))))))))))))))))).))))..))).... ( -50.10, z-score = -2.84, R) >triCas2.ChLG8 9984836 111 + 15773733 ------UUUUCCCCUCACGGGCGAGGAUCACCGGGUUGUGCCCCGUGGUGAUGACGCAGACGCGCGAUCGGUUCUCGUUCUGCUUUGGAAGAUUGCAGAUUUUCAAAGCGAUCUCUU ------............(((((((((((((((((.....)))...)))(((..(((....)))..))))))))))))))(((((((((((........)))))))))))....... ( -37.40, z-score = -0.28, R) >consensus _____CCGAAUCCUGC_UGGAUGAGCAGCACGGGAUCACAGCCCUGCGUGAGUAUGGCAAUGCGCGGCCGCGCCGGAUUCAGCUUCUCCAGCGCCACCAGUCGCUUGCCAAUCUCGC ...........................((.((((........)))).)).....((((((.(((.((..(((..(((.........)))..)))..))...)))))))))....... (-15.56 = -15.57 + 0.01)
| Location | 13,013,595 – 13,013,706 |
|---|---|
| Length | 111 |
| Sequences | 14 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 74.30 |
| Shannon entropy | 0.57990 |
| G+C content | 0.58658 |
| Mean single sequence MFE | -45.89 |
| Consensus MFE | -16.68 |
| Energy contribution | -16.44 |
| Covariance contribution | -0.23 |
| Combinations/Pair | 1.83 |
| Mean z-score | -2.37 |
| Structure conservation index | 0.36 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.57 |
| SVM RNA-class probability | 0.950929 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 13013595 111 - 24543557 GCGAGAUUGGCAAGCGACUGGUGGCGAUGGAGAAGAAAAACCCGACGCGGCCGCGCAUUGCCAUUCUCACGCAGGGCUGCGAUCCUGUACUGCUUAUACA-GCAGGAUUCGG----- ..((((.(((((((((...(((.(((.(((.(........)))).))).))).))).)))))).)))).((((....))))((((((..(((......))-)))))))....----- ( -46.40, z-score = -2.91, R) >droSim1.chr3L 12401031 111 - 22553184 GCGAGAUUGGCAAGCGAUUGGUGGCGAUGGAGAAGAAAAACCCGGCGCGGCCGCGCAUUGCCAUUCUCACGCAGGGCUGCGAUCCUGUACUGCUUAUUCA-GCAGGAUUCGG----- (.((((.(((((((((...(((.(((.(((.(........)))).))).))).))).)))))).)))).)((((((......)))))).(((((.....)-)))).......----- ( -46.00, z-score = -2.00, R) >droSec1.super_0 5185180 111 - 21120651 GCGAGAUUGGCAAGCGAUUGGUGGCGAUGGAGAAGAAAAACCCGGCGCGGCCGCGCAUUGCCAUUCUCACGCAGGGCUGCGAUCCUGUACUGCUUAUUCA-GCAGGAUUCGG----- (.((((.(((((((((...(((.(((.(((.(........)))).))).))).))).)))))).)))).)((((((......)))))).(((((.....)-)))).......----- ( -46.00, z-score = -2.00, R) >droYak2.chr3L 13092498 111 - 24197627 GCGAGAUUGGCAAGCGACUGGUGGCGAUGGAGAAGAAAAACCCGACGCGGCCGCGCAUUGCCAUCCUCACGCAGGGCUGCGAUCCUGUGCUGCUUAUUCA-GCAGGAUUCGG----- ..(((..(((((((((...(((.(((.(((.(........)))).))).))).))).))))))..))).(((((((......)))))))(((((.....)-)))).......----- ( -45.50, z-score = -1.61, R) >droEre2.scaffold_4784 13021128 111 - 25762168 GCGAGAUUGGCAAGCGACUGGUGGCGAUGGAGAAGAAAAACCCGACGCGACCGCGCAUUGCCAUCCUCACGCAGGGUUGCGAUCCUGUGCUGCUCAUCCA-GCAGGAUUCGG----- ..(((..(((((((((...(((.(((.(((.(........)))).))).))).))).))))))..))).((((((((....))))))))(((((.....)-)))).......----- ( -46.80, z-score = -2.34, R) >droAna3.scaffold_13337 19156351 111 + 23293914 GAGAGAUUGGCAAGCGGCUGGUGGCGUUCGACAAGCUGAACUCGUCGAGGCCGCGCAUUGCCAUUCUCACGCAGGGCUGUGAUCCCGUGCUGCUCUUCCA-GCAGGACUCGG----- ..((((.((((((...((..(((((.((((((.((.....)).))))))))))))).)))))).)))).((((....)))).....((.(((((.....)-)))).))....----- ( -46.30, z-score = -0.84, R) >dp4.chrXR_group6 4377285 111 - 13314419 GCGAGAUUGGCAAGCGUCUGGUGGCGCUGGACAAACUGAAUCCGGCGCGACCUCGCAUUGCCAUACUAACGCAGGGCUGUGAUCCCGUGCUGCUCAUCCA-GCGGGACUCGG----- .((((..(((((((((...(((.((((((((.........)))))))).))).))).))))))......((((....)))).((((..((((......))-)))))))))).----- ( -51.10, z-score = -2.95, R) >droPer1.super_9 2666466 111 - 3637205 GCGAGAUUGGCAAGCGUCUGGUGGCGCUGGACAAACUGAAUCCGGCGCGACCUCGCAUUGCCAUACUAACGCAGGGCUGUGAUCCCGUGCUGCUCAUCCA-GCGGGACUCGG----- .((((..(((((((((...(((.((((((((.........)))))))).))).))).))))))......((((....)))).((((..((((......))-)))))))))).----- ( -51.10, z-score = -2.95, R) >droWil1.scaffold_180698 6146028 111 + 11422946 GCGAGAUUGGCAAACGUAUAGUCGGUCUGGAGAAACUAAAUCCAAGUCGGCCACGCAUUGCCAUACUAACGCAGGGCUGUGAUCCGGUGUUGCUCAUUCA-AAAAGAUUCGG----- .((((..((((((.(((...(((((..((((.........))))..))))).)))..)))))).......((((.((((.....)))).)))).......-......)))).----- ( -32.90, z-score = -1.15, R) >droVir3.scaffold_13049 2740841 111 + 25233164 GUGAGAUUGGCAAGCGCCUGGUCGCGCUGAAUAAACUCAAUACGGGGCGACCACGCAUUGCCAUACUCACGCAGGGCUGUGACCCCGUGCUGCUUAUACA-GCACGAUUCGG----- (((((..((((((..((.(((((((.(((.((.......)).))).))))))).)).))))))..)))))...(((......)))(((((((......))-)))))......----- ( -51.50, z-score = -4.80, R) >droMoj3.scaffold_6680 7664310 111 + 24764193 GUGAGAUUGGCAAACGUUUGGUGGCACUGCCCAAAAUCAAUUCAGAGCGACCACGCAUUGCCAUACUCACGCAGGGCUGUGACCCCGUGCUGCUCAUCCA-GCACGACAAGG----- (((((..((((((..((.((((.((.(((.............))).)).)))).)).))))))..)))))...(((......)))(((((((......))-)))))......----- ( -41.92, z-score = -2.60, R) >droGri2.scaffold_15121 75770 114 + 279557 GUGAGAUUGGCAGGCGUCUGGUUGCACUGGACAAAUUGAAUUCAGCGCGACCACGCAUUGCGAUACUCACGCAGGGCUGUGAUCCCGUGCUGCUCAUCCA-GCACGACGCUCCGG-- (((((..(.((((((((..((((((.(((((.........))))).)))))))))).)))).)..)))))((.(((.......)))((((((......))-))))...)).....-- ( -45.90, z-score = -1.99, R) >anoGam1.chrU 23959213 117 + 59568033 GCGAGAUUGGCAAACGGAUCGCCGCCCUGCCGAAAGAGAAUGGCAAGCGGAAGCGCAUCGUGAUCAUUACGCAGGGCAGCGAUCCGGUGCUGCUGAUCGAGGCCGGAACCGACAACG ((..(((..(((..((((((((.(((((((..((.((..((((...((....))...))))..)).))..))))))).))))))))....)))..)))...))((....))...... ( -53.80, z-score = -3.69, R) >triCas2.ChLG8 9984836 111 - 15773733 AAGAGAUCGCUUUGAAAAUCUGCAAUCUUCCAAAGCAGAACGAGAACCGAUCGCGCGUCUGCGUCAUCACCACGGGGCACAACCCGGUGAUCCUCGCCCGUGAGGGGAAAA------ ..((((((((........(((((...........))))).........(((.((((....)))).)))....((((......))))))))).))).(((....))).....------ ( -37.20, z-score = -1.33, R) >consensus GCGAGAUUGGCAAGCGACUGGUGGCGCUGGAGAAAAAAAACCCGGCGCGACCGCGCAUUGCCAUACUCACGCAGGGCUGCGAUCCCGUGCUGCUCAUCCA_GCAGGAUUCGG_____ (((....((((((.......((.(((.(((...........))).))).))......))))))......))).((((.(((......))).))))...................... (-16.68 = -16.44 + -0.23)

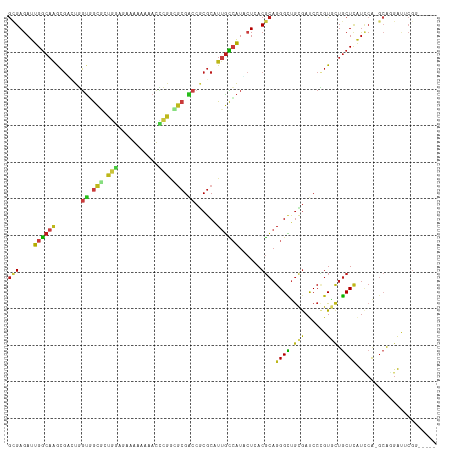
| Location | 13,013,626 – 13,013,737 |
|---|---|
| Length | 111 |
| Sequences | 14 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 71.36 |
| Shannon entropy | 0.62348 |
| G+C content | 0.61483 |
| Mean single sequence MFE | -47.24 |
| Consensus MFE | -17.40 |
| Energy contribution | -18.11 |
| Covariance contribution | 0.71 |
| Combinations/Pair | 1.68 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.37 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.13 |
| SVM RNA-class probability | 0.983328 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
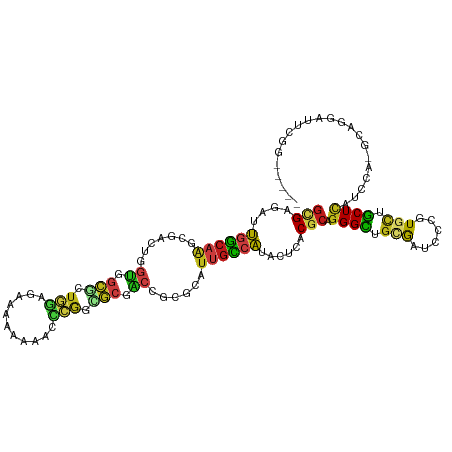
>dm3.chr3L 13013626 111 + 24543557 GCAGCCCUGCG-UGAGAAUGGCAAUGCGCGGCCGCGUCGGGUUUUUCUUCUCCAUCGCCACCAGUCGCUUGCCAAUCUCGCGCAGGUC---CCCGCUAGGCCACUGCUGGGCCUC---- ..(((((((((-(((((.((((((.((((((..(((..(((.........)))..)))..)).).))))))))).)))))))))))..---...)))(((((.(....)))))).---- ( -52.80, z-score = -3.35, R) >droSim1.chr3L 12401062 111 + 22553184 GCAGCCCUGCG-UGAGAAUGGCAAUGCGCGGCCGCGCCGGGUUUUUCUUCUCCAUCGCCACCAAUCGCUUGCCAAUCUCGCGCAGGUC---CCCGCUGGGCCACUGCUGGGCCUC---- .((((((((((-(((((.((((((.(((.((..(((..(((.........)))..)))..))...))))))))).)))))))))))..---...))))((((.(....)))))..---- ( -52.80, z-score = -2.79, R) >droSec1.super_0 5185211 111 + 21120651 GCAGCCCUGCG-UGAGAAUGGCAAUGCGCGGCCGCGCCGGGUUUUUCUUCUCCAUCGCCACCAAUCGCUUGCCAAUCUCGCGCAGGUC---CCCGCUGGGCCACUGCUGGGCCUC---- .((((((((((-(((((.((((((.(((.((..(((..(((.........)))..)))..))...))))))))).)))))))))))..---...))))((((.(....)))))..---- ( -52.80, z-score = -2.79, R) >droYak2.chr3L 13092529 111 + 24197627 GCAGCCCUGCG-UGAGGAUGGCAAUGCGCGGCCGCGUCGGGUUUUUCUUCUCCAUCGCCACCAGUCGCUUGCCAAUCUCGCGCAGGUC---CCCGCUGGGCCACUGCUGGGCCUC---- .((((((((((-(((((.((((((.((((((..(((..(((.........)))..)))..)).).))))))))).)))))))))))..---...))))((((.(....)))))..---- ( -54.30, z-score = -2.68, R) >droEre2.scaffold_4784 13021159 111 + 25762168 GCAACCCUGCG-UGAGGAUGGCAAUGCGCGGUCGCGUCGGGUUUUUCUUCUCCAUCGCCACCAGUCGCUUGCCAAUCUCGCGCAGGUC---CCCGCUGGGCCACUGCUGGGCCUC---- ((...((((((-(((((.((((((.(((((((.(((..(((.........)))..))).))).).))))))))).)))))))))))..---...)).(((((.(....)))))).---- ( -53.60, z-score = -3.41, R) >droAna3.scaffold_13337 19156382 111 - 23293914 ACAGCCCUGCG-UGAGAAUGGCAAUGCGCGGCCUCGACGAGUUCAGCUUGUCGAACGCCACCAGCCGCUUGCCAAUCUCUCGCAGAUC---CCCACUCGGCCAGCUCUGGGCCUC---- ......(((((-.((((.((((((.(((((((.((((((((.....))))))))..)))....).))))))))).)))).)))))...---.......((((.......))))..---- ( -50.30, z-score = -3.79, R) >dp4.chrXR_group6 4377316 111 + 13314419 ACAGCCCUGCG-UUAGUAUGGCAAUGCGAGGUCGCGCCGGAUUCAGUUUGUCCAGCGCCACCAGACGCUUGCCAAUCUCGCGCAGAUC---UUCGGUCGGCCAGCCCUGGGCCGU---- ......(((((-(.((..((((((.(((.(((.((((.((((.......)))).)))).)))...)))))))))..)).))))))...---......(((((.......))))).---- ( -49.20, z-score = -2.55, R) >droPer1.super_9 2666497 111 + 3637205 ACAGCCCUGCG-UUAGUAUGGCAAUGCGAGGUCGCGCCGGAUUCAGUUUGUCCAGCGCCACCAGACGCUUGCCAAUCUCGCGCAGAUC---UUCGGUCGGCCAGCCCUGGGCCGU---- ......(((((-(.((..((((((.(((.(((.((((.((((.......)))).)))).)))...)))))))))..)).))))))...---......(((((.......))))).---- ( -49.20, z-score = -2.55, R) >droWil1.scaffold_180698 6146059 111 - 11422946 ACAGCCCUGCG-UUAGUAUGGCAAUGCGUGGCCGACUUGGAUUUAGUUUCUCCAGACCGACUAUACGUUUGCCAAUCUCGCGCAGAUC---AUCCGUUGGCCAGCCCUGGCCAGU---- ......(((((-(.((..((((((.(((((..((..(((((.........)))))..))....)))))))))))..)).))))))...---.....(((((((....))))))).---- ( -40.00, z-score = -2.56, R) >droVir3.scaffold_13049 2740872 114 - 25233164 ACAGCCCUGCG-UGAGUAUGGCAAUGCGUGGUCGCCCCGUAUUGAGUUUAUUCAGCGCGACCAGGCGCUUGCCAAUCUCACGCAGAUCAUUGUCGGCGGGCCAACCUUGCGCAGU---- ...((.(((((-((((..(((((((((.(((((((...((..((((....))))))))))))).))).))))))..)))))))))..........(((((.....)))))))...---- ( -50.40, z-score = -3.06, R) >droMoj3.scaffold_6680 7664341 114 - 24764193 ACAGCCCUGCG-UGAGUAUGGCAAUGCGUGGUCGCUCUGAAUUGAUUUUGGGCAGUGCCACCAAACGUUUGCCAAUCUCACGCAGAUCCGCAUCGGCGGGCCAGCCUUGCGCCGU---- ...((.(((((-((((..((((((.(((((((.((.(((..(.......)..))).)).)))..))))))))))..)))))))))....))..(((((((....))...))))).---- ( -47.90, z-score = -1.91, R) >droGri2.scaffold_15121 75804 114 - 279557 ACAGCCCUGCG-UGAGUAUCGCAAUGCGUGGUCGCGCUGAAUUCAAUUUGUCCAGUGCAACCAGACGCCUGCCAAUCUCACGCAGAUCCUCAUCGGCGGCCCAGCCUUGUGCCGU---- ......(((((-((((....(((..(((((((.((((((.((.......)).)))))).)))..)))).)))....)))))))))..........(((((.(......).)))))---- ( -44.30, z-score = -2.90, R) >anoGam1.chrU 23959250 115 - 59568033 GCUGCCCUGCG-UAAUGAUCACGAUGCGCUUCCGCUUGCCAUUCUCUUUCGGCAGGGCGGCGAUCCGUUUGCCAAUCUCGCGCAAAUC---UUCCGUGCCGUAGUUGAACUCUUUCGCU ((((((((((.-...(((....((((.((........)))))).....)))))))))))))...........((((..((.(((....---.....)))))..))))............ ( -32.50, z-score = 0.19, R) >triCas2.ChLG8 9984867 111 + 15773733 -GUGCCCCGUGGUGAUGACGCAGACGCGCGAUCGGUUCUCGUUCUGCUUUGGAAGAUUGCAGAUUUUCAAAGCGAUCUCUUUUAAGUC---UUUCGUGCCGAAGUUCUGCUCGUU---- -..(((....)))(((((.(((((...((..(((((...((....((((.(((.((((((...........)))))))))...)))).---...)).))))).))))))))))))---- ( -31.20, z-score = 0.32, R) >consensus ACAGCCCUGCG_UGAGUAUGGCAAUGCGCGGCCGCGCCGGAUUCAGCUUCUCCAGCGCCACCAGUCGCUUGCCAAUCUCGCGCAGAUC___CUCGGUGGGCCAGCCCUGGGCCUU____ ......(((((...((..((((((.(((.((..(((..(((.........)))..)))..))...)))))))))..))..))))).............(.(((....))).)....... (-17.40 = -18.11 + 0.71)

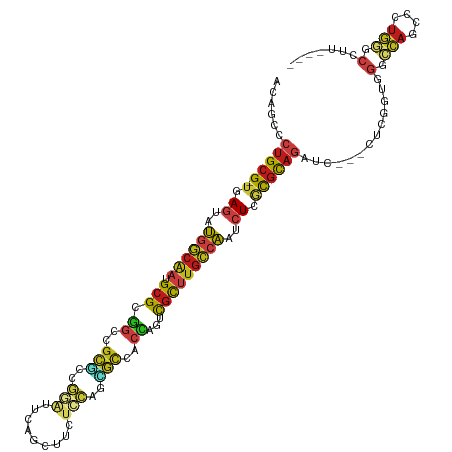
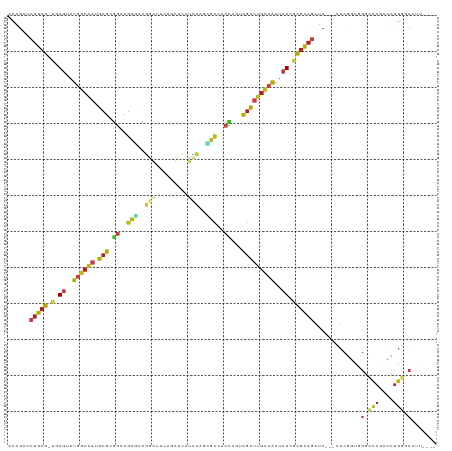
| Location | 13,013,626 – 13,013,737 |
|---|---|
| Length | 111 |
| Sequences | 14 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 71.36 |
| Shannon entropy | 0.62348 |
| G+C content | 0.61483 |
| Mean single sequence MFE | -50.91 |
| Consensus MFE | -19.24 |
| Energy contribution | -19.16 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.73 |
| Mean z-score | -2.87 |
| Structure conservation index | 0.38 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.00 |
| SVM RNA-class probability | 0.996852 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 13013626 111 - 24543557 ----GAGGCCCAGCAGUGGCCUAGCGGG---GACCUGCGCGAGAUUGGCAAGCGACUGGUGGCGAUGGAGAAGAAAAACCCGACGCGGCCGCGCAUUGCCAUUCUCA-CGCAGGGCUGC ----.((((((....).))))).((((.---..((((((.((((.(((((((((...(((.(((.(((.(........)))).))).))).))).)))))).)))).-)))))).)))) ( -58.50, z-score = -4.06, R) >droSim1.chr3L 12401062 111 - 22553184 ----GAGGCCCAGCAGUGGCCCAGCGGG---GACCUGCGCGAGAUUGGCAAGCGAUUGGUGGCGAUGGAGAAGAAAAACCCGGCGCGGCCGCGCAUUGCCAUUCUCA-CGCAGGGCUGC ----(.(((((....).))))).((((.---..((((((.((((.(((((((((...(((.(((.(((.(........)))).))).))).))).)))))).)))).-)))))).)))) ( -57.00, z-score = -3.03, R) >droSec1.super_0 5185211 111 - 21120651 ----GAGGCCCAGCAGUGGCCCAGCGGG---GACCUGCGCGAGAUUGGCAAGCGAUUGGUGGCGAUGGAGAAGAAAAACCCGGCGCGGCCGCGCAUUGCCAUUCUCA-CGCAGGGCUGC ----(.(((((....).))))).((((.---..((((((.((((.(((((((((...(((.(((.(((.(........)))).))).))).))).)))))).)))).-)))))).)))) ( -57.00, z-score = -3.03, R) >droYak2.chr3L 13092529 111 - 24197627 ----GAGGCCCAGCAGUGGCCCAGCGGG---GACCUGCGCGAGAUUGGCAAGCGACUGGUGGCGAUGGAGAAGAAAAACCCGACGCGGCCGCGCAUUGCCAUCCUCA-CGCAGGGCUGC ----(.(((((....).))))).((((.---..((((((.(((..(((((((((...(((.(((.(((.(........)))).))).))).))).))))))..))).-)))))).)))) ( -55.10, z-score = -2.79, R) >droEre2.scaffold_4784 13021159 111 - 25762168 ----GAGGCCCAGCAGUGGCCCAGCGGG---GACCUGCGCGAGAUUGGCAAGCGACUGGUGGCGAUGGAGAAGAAAAACCCGACGCGACCGCGCAUUGCCAUCCUCA-CGCAGGGUUGC ----(.(((((....).))))).((((.---..((((((.(((..(((((((((...(((.(((.(((.(........)))).))).))).))).))))))..))).-)))))).)))) ( -52.50, z-score = -2.94, R) >droAna3.scaffold_13337 19156382 111 + 23293914 ----GAGGCCCAGAGCUGGCCGAGUGGG---GAUCUGCGAGAGAUUGGCAAGCGGCUGGUGGCGUUCGACAAGCUGAACUCGUCGAGGCCGCGCAUUGCCAUUCUCA-CGCAGGGCUGU ----..((((.......))))......(---(..(((((.((((.((((((...((..(((((.((((((.((.....)).))))))))))))).)))))).)))).-)))))..)).. ( -52.30, z-score = -1.82, R) >dp4.chrXR_group6 4377316 111 - 13314419 ----ACGGCCCAGGGCUGGCCGACCGAA---GAUCUGCGCGAGAUUGGCAAGCGUCUGGUGGCGCUGGACAAACUGAAUCCGGCGCGACCUCGCAUUGCCAUACUAA-CGCAGGGCUGU ----(((((((..((....)).......---....((((..((..(((((((((...(((.((((((((.........)))))))).))).))).))))))..))..-))))))))))) ( -55.10, z-score = -3.67, R) >droPer1.super_9 2666497 111 - 3637205 ----ACGGCCCAGGGCUGGCCGACCGAA---GAUCUGCGCGAGAUUGGCAAGCGUCUGGUGGCGCUGGACAAACUGAAUCCGGCGCGACCUCGCAUUGCCAUACUAA-CGCAGGGCUGU ----(((((((..((....)).......---....((((..((..(((((((((...(((.((((((((.........)))))))).))).))).))))))..))..-))))))))))) ( -55.10, z-score = -3.67, R) >droWil1.scaffold_180698 6146059 111 + 11422946 ----ACUGGCCAGGGCUGGCCAACGGAU---GAUCUGCGCGAGAUUGGCAAACGUAUAGUCGGUCUGGAGAAACUAAAUCCAAGUCGGCCACGCAUUGCCAUACUAA-CGCAGGGCUGU ----..((((((....))))))((((..---..((((((..((..((((((.(((...(((((..((((.........))))..))))).)))..))))))..))..-)))))).)))) ( -45.00, z-score = -2.97, R) >droVir3.scaffold_13049 2740872 114 + 25233164 ----ACUGCGCAAGGUUGGCCCGCCGACAAUGAUCUGCGUGAGAUUGGCAAGCGCCUGGUCGCGCUGAAUAAACUCAAUACGGGGCGACCACGCAUUGCCAUACUCA-CGCAGGGCUGU ----((.((.((..(((((....)))))..)).((((((((((..((((((..((.(((((((.(((.((.......)).))).))))))).)).))))))..))))-)))))))).)) ( -56.40, z-score = -4.77, R) >droMoj3.scaffold_6680 7664341 114 + 24764193 ----ACGGCGCAAGGCUGGCCCGCCGAUGCGGAUCUGCGUGAGAUUGGCAAACGUUUGGUGGCACUGCCCAAAAUCAAUUCAGAGCGACCACGCAUUGCCAUACUCA-CGCAGGGCUGU ----.(((((...((....)))))))..((((..(((((((((..((((((..((.((((.((.(((.............))).)).)))).)).))))))..))))-)))))..)))) ( -52.52, z-score = -3.35, R) >droGri2.scaffold_15121 75804 114 + 279557 ----ACGGCACAAGGCUGGGCCGCCGAUGAGGAUCUGCGUGAGAUUGGCAGGCGUCUGGUUGCACUGGACAAAUUGAAUUCAGCGCGACCACGCAUUGCGAUACUCA-CGCAGGGCUGU ----.((((.....))))((((.((.....))..(((((((((..(.((((((((..((((((.(((((.........))))).)))))))))).)))).)..))))-))))))))).. ( -53.20, z-score = -3.96, R) >anoGam1.chrU 23959250 115 + 59568033 AGCGAAAGAGUUCAACUACGGCACGGAA---GAUUUGCGCGAGAUUGGCAAACGGAUCGCCGCCCUGCCGAAAGAGAAUGGCAAGCGGAAGCGCAUCGUGAUCAUUA-CGCAGGGCAGC .((((....((((((((.((((((....---)...))).)))).))))...))...)))).(((((((..((.((..((((...((....))...))))..)).)).-.)))))))... ( -36.60, z-score = -0.34, R) >triCas2.ChLG8 9984867 111 - 15773733 ----AACGAGCAGAACUUCGGCACGAAA---GACUUAAAAGAGAUCGCUUUGAAAAUCUGCAAUCUUCCAAAGCAGAACGAGAACCGAUCGCGCGUCUGCGUCAUCACCACGGGGCAC- ----.....(((((....((((.(....---)..........(((((.(((.....(((((...........)))))....))).))))))).)))))))(((.((.....)))))..- ( -26.40, z-score = 0.21, R) >consensus ____AAGGCCCAGGACUGGCCCACCGAG___GAUCUGCGCGAGAUUGGCAAGCGACUGGUGGCGCUGGAGAAAAAAAACCCGGCGCGACCGCGCAUUGCCAUACUCA_CGCAGGGCUGU .................................((((((..((..((((((.......((.(((.(((...........))).))).))......))))))..))...))))))..... (-19.24 = -19.16 + -0.08)
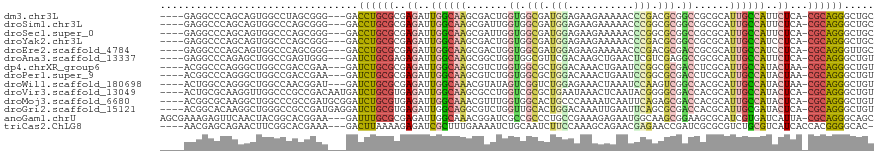

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:22:36 2011