| Sequence ID | dm3.chr3L |
|---|---|
| Location | 11,969,309 – 11,969,419 |
| Length | 110 |
| Max. P | 0.956923 |

| Location | 11,969,309 – 11,969,419 |
|---|---|
| Length | 110 |
| Sequences | 12 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 70.60 |
| Shannon entropy | 0.62182 |
| G+C content | 0.53049 |
| Mean single sequence MFE | -35.22 |
| Consensus MFE | -18.84 |
| Energy contribution | -17.61 |
| Covariance contribution | -1.23 |
| Combinations/Pair | 1.57 |
| Mean z-score | -1.36 |
| Structure conservation index | 0.53 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.64 |
| SVM RNA-class probability | 0.956923 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 11969309 110 + 24543557 ----GUUACUUUUCUCGUAUCCAGCUACUCUGCUCACUGUGGUGGCACUCACUGCAGUGGUU---GCCCUAGUUGGAGCCGCCGAUCCUGAAUGCAUAUACCGGAGGACUAGGAACC ----......(((((.((.((((((((....((.(((((..(((.....)))..))))))).---....)))))))).((.(((.................))).)))).))))).. ( -34.03, z-score = -0.58, R) >droSim1.chr3L 11335333 110 + 22553184 ----GUUACUUUUCUCGUAUCCAGCUACUCUGCUCACUGUGGUGGCACUCACUGCAGUGGCU---GCCCUAGUUGGAGCCGCCGAUCCUGAAUGCAUAUACCGGAGGACUAGGAACC ----......(((((.((.((((((((....((.(((((..(((.....)))..))))))).---....)))))))).((.(((.................))).)))).))))).. ( -35.63, z-score = -0.91, R) >droSec1.super_0 4155586 110 + 21120651 ----GUUACUUUUCUCGUAUCCAGCUACUCUGCUCACUGUGGUGGCAAUCACUGCAGUGGCU---GCCCUAGUUGGAGCCGCCGAUCCCGAAUGCAUAUACCGGAGGACUAGGAACC ----(((.((.(((((...((((((((....((.(((((..(((.....)))..))))))).---....))))))))((...((....))...))........)))))..)).))). ( -35.80, z-score = -1.13, R) >droYak2.chr3L 12014464 110 + 24197627 ----GUUACUUUUCUCACAUCCAGCUACUCUGCUCACUGUGGUGGCACUCACUGCAGUGGCU---GCCCUAGUUGGAGCCGCCGAUCCCGAAUGCCUAUACCGGAGGACGAGGAACC ----......((((((...((((((((....((.(((((..(((.....)))..))))))).---....))))))))(((.(((.................))).)).))))))).. ( -38.53, z-score = -1.72, R) >droEre2.scaffold_4784 11956303 113 + 25762168 -GUUUCUACUUUUCUCGUAUCCAGCUACUCUGCUCACUGUGGUGGCACUCACUGCAGUGGCU---GCUCUAGUUGGAGCCGCCGAUCCUGAAUGUAUCUACCGGAGGACUAGGAAGC -.((((((.((((((.(((((((((((....((.(((((..(((.....)))..))))))).---....))))))))......(((.(.....).)))))).)))))).)))))).. ( -38.80, z-score = -1.47, R) >droAna3.scaffold_13337 6219156 113 + 23293914 ----AUUUCUGUUUCCACCAUCAGUUACUCUGCUAGCUGUGGUGACACUCACUGCAGUGACCCUCGCCGUGGUGGAUGCCGUCGACCCUGAAUGCCUUUACCGGAAGAUACGCAACC ----.((((((..((((((((((((((......)))))).(((((...((((....))))...))))))))))))).((..(((....)))..))......)))))).......... ( -32.60, z-score = -1.27, R) >dp4.chrXR_group6 1968084 110 + 13314419 ----CUCACGAUUUCAUUUUUUAGUUGCUCUGCUAGCUGUGGUGACACUCACUACAGUGGCGGU---CCUUGUCGGAGCUGCCGAUCCUGAAUGCGUCUACCGCAAAUUGCGUGGUC ----.(((((..((((.....((((......))))(((((((((.....)))))))))((((((---((.....))).))))).....))))((((.....)))).....))))).. ( -36.80, z-score = -1.98, R) >droPer1.super_9 259068 110 + 3637205 ----CUCACGAUUUCAUUUUUUAGUUGCUCUGCUAGCUGUGGUGACACUCACUACAGUGGCGGU---CCUUGUCGGAGCUGCCGAUCCUGAAUGCGUCUACCGCAAAUUGCGUGGUC ----.(((((..((((.....((((......))))(((((((((.....)))))))))((((((---((.....))).))))).....))))((((.....)))).....))))).. ( -36.80, z-score = -1.98, R) >droWil1.scaffold_180698 8147581 110 - 11422946 -----UUUCCAUUUG--CCAUUAGUUACUCUGCUAGCUGUGGUGGCACUGACUACAGUGGCAAUUGCCCUAGUCGGAGCAGCCGAUCCUGAAUGCGUUUACCGCAGACUACGUGGUC -----.........(--(((((((((.(((((((((..(..(((.(((((....))))).).))..).)))).)))))(((......)))..((((.....))))))))).))))). ( -34.50, z-score = -0.82, R) >droVir3.scaffold_13049 4155717 114 - 25233164 ACUUUUUCUAACUCUUCCCAAUAGUUACUCUGCUUGCUGUGGUUACAUUGACCGCAGUGGCAUC---CCUUGCCGGCGCUGCUGAUCCUGAAUGCGUUUACCGCAAGCUACGUGGAC .................(((.(((((.....(((.(((((((((.....)))))))))((((..---...)))))))...............((((.....)))))))))..))).. ( -31.20, z-score = -1.42, R) >droMoj3.scaffold_6680 9314686 109 - 24764193 -----UUUCGUUUUCCCCUAACAGUUACUCUGCUUACUGUGGUUACGUUGACCGCAGUGGCAGC---AAUUGUCGGUGCUGCCGAUCCUGAAUGCGUUUACCGCAAGCUACGUGGCC -----..................(((((...(((((((((((((.....)))))))))((((((---(........)))))))..........(((.....)))))))...))))). ( -35.90, z-score = -1.96, R) >droGri2.scaffold_15110 22609673 114 + 24565398 UCUUUCUCCAUACCUUUACAACAGUUACUCUGCUUACUGUGGUUACAUUCGCCGCAGUGGCAAC---CCUUGCCAGCGCUGCCGAUCCUGAGUGCGUCUACAGGAAAUUGCGUGGAU ......(((((....................(((.((((((((.......))))))))((((..---...)))))))((......(((((((.....)).)))))....))))))). ( -32.10, z-score = -1.09, R) >consensus ____GUUACUUUUCUCGUAUCCAGUUACUCUGCUCACUGUGGUGACACUCACUGCAGUGGCU___GCCCUAGUCGGAGCCGCCGAUCCUGAAUGCGUAUACCGGAAGAUACGUAACC .......................((...((.(((.(((((((((.....))))))))))))..........(((((.....)))))...))..))...................... (-18.84 = -17.61 + -1.23)

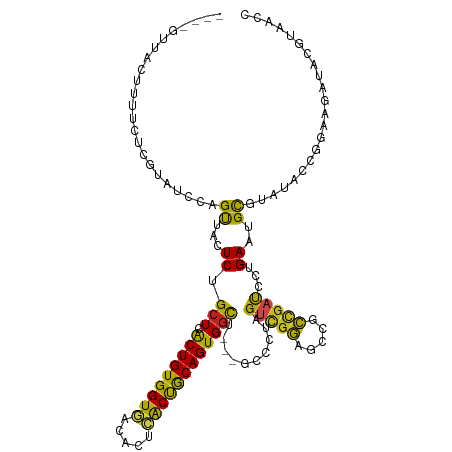

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:20:04 2011