
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 11,033,518 – 11,033,614 |
| Length | 96 |
| Max. P | 0.550977 |

| Location | 11,033,518 – 11,033,614 |
|---|---|
| Length | 96 |
| Sequences | 12 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 70.69 |
| Shannon entropy | 0.58313 |
| G+C content | 0.61719 |
| Mean single sequence MFE | -41.14 |
| Consensus MFE | -15.03 |
| Energy contribution | -14.30 |
| Covariance contribution | -0.73 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.37 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.12 |
| SVM RNA-class probability | 0.550977 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
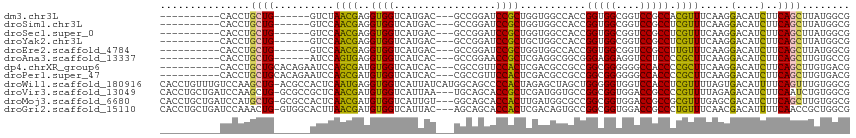
>dm3.chr3L 11033518 96 + 24543557 ----------CACCUGCUG------GUCUAACGAGGUGGUCAUGAC---GCCGGAUCCGCUGGUGGCCACCGGUGGCGGUCCGCCACGUUUCAAGGACAUCUUCAGCUUAUGGCG ----------..((.((((------((((((((.(((((((((.(.---((.(....)))).)))))))))(((((....))))).))))....)))).....))))....)).. ( -36.20, z-score = -0.33, R) >droSim1.chr3L 10431197 96 + 22553184 ----------CACCUGCUG------GUCCAACGAGGUGGUCAUGAC---GCCGGAUCCGCUGGUGGCCACCGGUGGCGGUCCGCCUCGUUUCAAGGACAUCUUCAGCUUAUGGCG ----------..((.((((------((((((((((((((.(....(---(((((.....))))))((((....)))).).))))))))))....)))).....))))....)).. ( -42.60, z-score = -1.98, R) >droSec1.super_0 3261251 96 + 21120651 ----------CACCUGCUG------GUCCAACGAGGUGGUCAUGAC---GCCGGAUCCGCUGGUGGCCACCGGUGGCGGUCCGCCUCGUUUCAAGGACAUCUUCAGCUUAUGGCG ----------..((.((((------((((((((((((((.(....(---(((((.....))))))((((....)))).).))))))))))....)))).....))))....)).. ( -42.60, z-score = -1.98, R) >droYak2.chr3L 11061807 96 + 24197627 ----------CACCUGCUG------GUCCAACGAGGUGGUCAUGAC---GCCGGAUCCGCUGCUGGCCACCGGUGGCGGUCCGCCUCGUUUCAAGGACAUCUUCAGCUUAUGGCG ----------..((.((((------(((((((((((((((......---)))(((.((((..((((...))))..)))))))))))))))....)))).....))))....)).. ( -44.30, z-score = -2.74, R) >droEre2.scaffold_4784 11042366 96 + 25762168 ----------CACCUGCUG------GUCCAACGAGGUGGUCAUGAC---GCCGGAUCCGCUGGUGGCCACCGGUGGCGGUCCGCCUUGUUUCAAGGACAUCUUCAGCUUAUGGCG ----------..((.((((------((((((((((((((.(....(---(((((.....))))))((((....)))).).))))))))))....)))).....))))....)).. ( -40.50, z-score = -1.50, R) >droAna3.scaffold_13337 15946914 96 + 23293914 ----------CACCUGCUG------AUCCAGUGAGGUGGUCAUCAC---GCCGGAACCGCUCGAGGCGGCGGGAGGAGGUCCUCCCCGCUUCAAGGACAUCUUCAGCUUGUGCCG ----------(((..((((------((((..((((((((......(---((((...((......)))))))(((((....))))))))))))).))).....)))))..)))... ( -41.80, z-score = -1.47, R) >dp4.chrXR_group6 7323785 102 - 13314419 ----------CACCUGCUGCACAGAAUCCAGCGAUGUGGUCAUCAC---CGCCGUUCCACUCGACGCCGCCGGCGGGGGGCCACCCCGCUUCAAGGACAUCUUCAGCUUGUGACG ----------(((..((((...(((.(((((((..((((((.((.(---(((((................))))))))))))))..))))....)))..))).))))..)))... ( -38.09, z-score = -1.22, R) >droPer1.super_47 101710 102 - 592741 ----------CACCUGCUGCACAGAAUCCAGCGAUGUGGUCAUCAC---CGCCGUUCCACUCGACGCCGCCGGCGGGGGGCCACCCCGCUUCAAGGACAUCUUCAGCUUGUGACG ----------(((..((((...(((.(((((((..((((((.((.(---(((((................))))))))))))))..))))....)))..))).))))..)))... ( -38.09, z-score = -1.22, R) >droWil1.scaffold_180916 163343 114 - 2700594 CACCUGUUUGUCCAAGCUG-ACGCCACUCAAUGAGGUGGUCAUUAUCAUGGCAGCCCCACUAGAGCUAGCUGGGGGUGGUCCACCUCGUUUUAGUGACAUUUUCAGUUUGUGGCG .........((((((((((-(.(.((((.((((((((((((((....))))((.((((((((....))).))))).))..))))))))))..)))).)....)))))))).))). ( -44.80, z-score = -2.86, R) >droVir3.scaffold_13049 15721729 111 + 25233164 CACCUGCUGAUCCAAGCUG-GCGCCGCUCAACGAUGUGGUCAUUAA---UGCAGCACCGCUCGAUGGUGCCGGCGGUGGACCGCCCCGUUUUAGAGACAUCUUCAAUCUGUGGCG ..((.(((......))).)-)((((((.....((....(((.((((---.((.((((((.....)))))).(((((....)))))..)).)))).)))....)).....)))))) ( -40.10, z-score = -0.80, R) >droMoj3.scaffold_6680 3644605 111 - 24764193 CACCUGCUGAUCCAUGCUG-GCGCCACUCAACGAUGUGGUCAUUGU---GGCAGCACCACUUGAUGGCGCCGGCGGUGGACCGCCGCGUUUGAGCGACAUCUUCAGCUUGUGGCG (((..((((((((((((((-((((((.((((.((((....))))((---((.....)))))))))))))))))).)))))..(.(((......))).)....)))))..)))... ( -50.30, z-score = -2.24, R) >droGri2.scaffold_15110 11652277 111 - 24565398 CACCUGCUGAUCCAAACUG-GUGGCACUUAACGAUGUGGUCAUUAC---AGCAGCACCACUCGACAGUGCCGGCGGUGGACCGCCCUGUUUCAACGACAUUUUCAACCGCUGGCG ..((.(((((..((.....-..((((((...(((.((((((.....---....).))))))))..))))))(((((....))))).))..)))..((.....))....)).)).. ( -34.30, z-score = -0.47, R) >consensus __________CACCUGCUG______AUCCAACGAGGUGGUCAUGAC___GCCGGAUCCGCUCGAGGCCGCCGGCGGCGGUCCGCCCCGUUUCAAGGACAUCUUCAGCUUGUGGCG ...............((((..........((((..((((.................))))...........(((((....))))).)))).............))))........ (-15.03 = -14.30 + -0.73)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:17:39 2011