| Sequence ID | dm3.chr3L |
|---|---|
| Location | 10,312,886 – 10,312,987 |
| Length | 101 |
| Max. P | 0.999592 |

| Location | 10,312,886 – 10,312,987 |
|---|---|
| Length | 101 |
| Sequences | 14 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 82.49 |
| Shannon entropy | 0.38756 |
| G+C content | 0.46185 |
| Mean single sequence MFE | -39.76 |
| Consensus MFE | -32.24 |
| Energy contribution | -32.58 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.13 |
| Mean z-score | -5.14 |
| Structure conservation index | 0.81 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 4.05 |
| SVM RNA-class probability | 0.999592 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
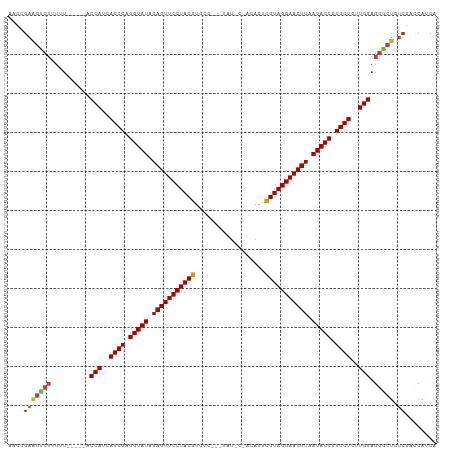
>dm3.chr3L 10312886 101 + 24543557 AACCGAAGUCUUUUU-----ACCAUCAGCGAGGUAUAGAGUUCCUACGUUCC---UAUAU-UCAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCAGCA ...(((((((((...-----.(((..((((.(((((.(((((((((((...(---(....-..)).))))))))))).))))).))))..))))))))).)))....... ( -39.50, z-score = -5.23, R) >anoGam1.chr2L 18422515 96 + 48795086 --------GGUGACU-----GCCAUCAGCGAGGUAUAGAGUUCCUACGGUAAUCGAUUGAAACU-UUGUAGGAACUUCAUACCGUGCUCUUGGAUAGCCGUUUACCAGCG --------(((((((-----((((..((((.(((((..(((((((((((...((....))....-)))))))))))..))))).))))..))).)))....))))).... ( -37.50, z-score = -4.44, R) >droGri2.scaffold_15110 11992969 103 - 24565398 UAACGAAGUCUAUUU-----ACCAUUAGCGAGGUAUAGAGUUCCUACGUGCA--AAUUGCAAUAAUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCGACCAGCA ...((((((((....-----.(((..((((.(((((.((((((((((((((.--....))).....))))))))))).))))).))))..))).))))).)))....... ( -40.40, z-score = -5.24, R) >droMoj3.scaffold_6680 4085877 103 - 24764193 UAACGAAGUCUAUAU-----ACCAUCAGCGAGGUAUAGAGUUCCUACGUGCA--AAUUGCAAUAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCGACCAACA ...((((((((....-----.(((..((((.(((((.((((((((((((((.--....))).....))))))))))).))))).))))..))).))))).)))....... ( -40.10, z-score = -5.09, R) >droVir3.scaffold_13049 16093215 103 + 25233164 UACCGAAGUCUAUAU-----ACCAUCAGCGAGGUAUAGAGUUCCUACGUGCA--AAUUGCAAUAAUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCGACCAGCA ...((((((((....-----.(((..((((.(((((.((((((((((((((.--....))).....))))))))))).))))).))))..))).))))).)))....... ( -39.90, z-score = -4.83, R) >droWil1.scaffold_181024 1461207 103 - 1751709 AAACGAAGCCUAUUU-----ACCAUAAGCGAGGUAUAGAGUUCCUACGUGCA--AAUUGCAAUAAUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCGAACAACA ...((((((((....-----.(((..((((.(((((.((((((((((((((.--....))).....))))))))))).))))).))))..))).))))).)))....... ( -43.30, z-score = -6.82, R) >droSim1.chr3L 9702912 99 + 22553184 AACCGAAGUCUUUUU-----ACCAUCAGCGAGGUAUAGAGUUCCUACGUUCC---UAU---UCAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCAUCA ...(((((((((...-----.(((..((((.(((((.(((((((((((....---...---.....))))))))))).))))).))))..))))))))).)))....... ( -39.30, z-score = -5.41, R) >droSec1.super_0 2526761 99 + 21120651 AACCGAAGUCUUUUU-----ACCAUCAGCGAGGUAUAGAGUUCCUACGUUCC---UAU---UCAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCAUCA ...(((((((((...-----.(((..((((.(((((.(((((((((((....---...---.....))))))))))).))))).))))..))))))))).)))....... ( -39.30, z-score = -5.41, R) >droYak2.chr3L 10302146 99 + 24197627 AACCGAAGUCUUUUU-----ACCAUCAGCGAGGUAUAGAGUUCCUACGUUCC---UAU---UCAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCAUCA ...(((((((((...-----.(((..((((.(((((.(((((((((((....---...---.....))))))))))).))))).))))..))))))))).)))....... ( -39.30, z-score = -5.41, R) >droEre2.scaffold_4784 10306182 99 + 25762168 AACCGAAGUCUUUUU-----ACCAUCAGCGAGGUAUAGAGUUCCUACGUUCC---UAU---UCAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCAUCA ...(((((((((...-----.(((..((((.(((((.(((((((((((....---...---.....))))))))))).))))).))))..))))))))).)))....... ( -39.30, z-score = -5.41, R) >droAna3.scaffold_13337 15233378 103 + 23293914 AACCGAAGCCUAUUU-----GCCAUCAGCGAGGUAUAGAGUUCCUACGUGCCA--CUUAAACUAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCGACCAUCA ...((((((((....-----.(((..((((.(((((.(((((((((((....(--((......)))))))))))))).))))).))))..))).))))).)))....... ( -42.10, z-score = -5.09, R) >dp4.chrXR_group6 8706034 101 + 13314419 AAGCGAAGCCUCUUU-----ACCAUCAGCGAGGUAUAGAGUUCCUACGUGCCG--UAUCC-AAAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCCAUCAAC- ..(.((((((.((..-----.(((..((((.(((((.(((((((((((.((..--.....-...))))))))))))).))))).))))..))))))))).)))......- ( -38.20, z-score = -4.31, R) >droPer1.super_27 1146109 101 + 1201981 AAGCGAAGCCUCUUU-----ACCAUCAGCGAGGUAUAGAGUUCCUACGUGCCG--UAUCC-AAAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCCAUCAAC- ..(.((((((.((..-----.(((..((((.(((((.(((((((((((.((..--.....-...))))))))))))).))))).))))..))))))))).)))......- ( -38.20, z-score = -4.31, R) >triCas2.ChLG3 18613773 102 - 32080666 AACGGCGCUCUUUUUGGUAGACCACUAGCGAGGUAUAGAGUUCCUACGUGAU---UUU---UCGGCUGUAGGAACUUCAUACCGUGCUCUUGGAU--UUACCGACCAUCA .............(((((((((((..((((.(((((..(((((((((((((.---...---)))..))))))))))..))))).))))..))).)--)))))))...... ( -40.30, z-score = -5.02, R) >consensus AACCGAAGUCUUUUU_____ACCAUCAGCGAGGUAUAGAGUUCCUACGUGCC___UAU_C_ACAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCGACCAUCA ....(((((((..........(((..((((.(((((.(((((((((((..................))))))))))).))))).))))..))).))))).))........ (-32.24 = -32.58 + 0.33)


| Location | 10,312,886 – 10,312,987 |
|---|---|
| Length | 101 |
| Sequences | 14 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 82.49 |
| Shannon entropy | 0.38756 |
| G+C content | 0.46185 |
| Mean single sequence MFE | -35.21 |
| Consensus MFE | -26.72 |
| Energy contribution | -26.86 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.59 |
| Structure conservation index | 0.76 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.81 |
| SVM RNA-class probability | 0.999341 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
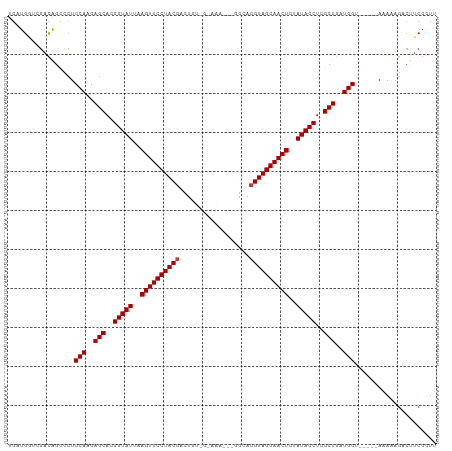
>dm3.chr3L 10312886 101 - 24543557 UGCUGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUGA-AUAUA---GGAACGUAGGAACUCUAUACCUCGCUGAUGGU-----AAAAAGACUUCGGUU ......((((.((((..(((..(((..(((((..((((((((((.(((.-...))---)...))))))))))..)))))..)))..))).-----.....)))))))).. ( -34.90, z-score = -3.33, R) >anoGam1.chr2L 18422515 96 - 48795086 CGCUGGUAAACGGCUAUCCAAGAGCACGGUAUGAAGUUCCUACAA-AGUUUCAAUCGAUUACCGUAGGAACUCUAUACCUCGCUGAUGGC-----AGUCACC-------- ....(((..((.((((((....(((..((((((.(((((((((..-....((....)).....))))))))).))))))..)))))))))-----.)).)))-------- ( -32.10, z-score = -3.54, R) >droGri2.scaffold_15110 11992969 103 + 24565398 UGCUGGUCGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGAUUAUUGCAAUU--UGCACGUAGGAACUCUAUACCUCGCUAAUGGU-----AAAUAGACUUCGUUA .((((.....))))...(((..(((..(((((..((((((((((.....(((....--.)))))))))))))..)))))..)))..))).-----............... ( -35.90, z-score = -4.48, R) >droMoj3.scaffold_6680 4085877 103 + 24764193 UGUUGGUCGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUAUUGCAAUU--UGCACGUAGGAACUCUAUACCUCGCUGAUGGU-----AUAUAGACUUCGUUA ....((((.........(((..(((..(((((..((((((((((.....(((....--.)))))))))))))..)))))..)))..))).-----.....))))...... ( -33.46, z-score = -3.22, R) >droVir3.scaffold_13049 16093215 103 - 25233164 UGCUGGUCGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGAUUAUUGCAAUU--UGCACGUAGGAACUCUAUACCUCGCUGAUGGU-----AUAUAGACUUCGGUA .((((.....))))...(((..(((..(((((..((((((((((.....(((....--.)))))))))))))..)))))..)))..))).-----......((....)). ( -37.50, z-score = -4.06, R) >droWil1.scaffold_181024 1461207 103 + 1751709 UGUUGUUCGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGAUUAUUGCAAUU--UGCACGUAGGAACUCUAUACCUCGCUUAUGGU-----AAAUAGGCUUCGUUU .......(((.((((..(((.((((..(((((..((((((((((.....(((....--.)))))))))))))..)))))..)))).))).-----.....)))))))... ( -39.10, z-score = -5.74, R) >droSim1.chr3L 9702912 99 - 22553184 UGAUGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUGA---AUA---GGAACGUAGGAACUCUAUACCUCGCUGAUGGU-----AAAAAGACUUCGGUU ......((((.((((..(((..(((..(((((..((((((((((.((..---..)---)...))))))))))..)))))..)))..))).-----.....)))))))).. ( -34.30, z-score = -3.29, R) >droSec1.super_0 2526761 99 - 21120651 UGAUGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUGA---AUA---GGAACGUAGGAACUCUAUACCUCGCUGAUGGU-----AAAAAGACUUCGGUU ......((((.((((..(((..(((..(((((..((((((((((.((..---..)---)...))))))))))..)))))..)))..))).-----.....)))))))).. ( -34.30, z-score = -3.29, R) >droYak2.chr3L 10302146 99 - 24197627 UGAUGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUGA---AUA---GGAACGUAGGAACUCUAUACCUCGCUGAUGGU-----AAAAAGACUUCGGUU ......((((.((((..(((..(((..(((((..((((((((((.((..---..)---)...))))))))))..)))))..)))..))).-----.....)))))))).. ( -34.30, z-score = -3.29, R) >droEre2.scaffold_4784 10306182 99 - 25762168 UGAUGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUGA---AUA---GGAACGUAGGAACUCUAUACCUCGCUGAUGGU-----AAAAAGACUUCGGUU ......((((.((((..(((..(((..(((((..((((((((((.((..---..)---)...))))))))))..)))))..)))..))).-----.....)))))))).. ( -34.30, z-score = -3.29, R) >droAna3.scaffold_13337 15233378 103 - 23293914 UGAUGGUCGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUAGUUUAAG--UGGCACGUAGGAACUCUAUACCUCGCUGAUGGC-----AAAUAGGCUUCGGUU ......((((.((((..(((..(((..(((((..(((((((((((((......))--)....))))))))))..)))))..)))..))).-----.....)))))))).. ( -35.90, z-score = -2.66, R) >dp4.chrXR_group6 8706034 101 - 13314419 -GUUGAUGGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUUU-GGAUA--CGGCACGUAGGAACUCUAUACCUCGCUGAUGGU-----AAAGAGGCUUCGCUU -.......((.(((((((((..(((..(((((..((((((((((...((-(....--)))..))))))))))..)))))..)))..))).-----..)).)))))).... ( -36.00, z-score = -3.05, R) >droPer1.super_27 1146109 101 - 1201981 -GUUGAUGGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUUU-GGAUA--CGGCACGUAGGAACUCUAUACCUCGCUGAUGGU-----AAAGAGGCUUCGCUU -.......((.(((((((((..(((..(((((..((((((((((...((-(....--)))..))))))))))..)))))..)))..))).-----..)).)))))).... ( -36.00, z-score = -3.05, R) >triCas2.ChLG3 18613773 102 + 32080666 UGAUGGUCGGUAA--AUCCAAGAGCACGGUAUGAAGUUCCUACAGCCGA---AAA---AUCACGUAGGAACUCUAUACCUCGCUAGUGGUCUACCAAAAAGAGCGCCGUU .((((((.((((.--..(((..(((..((((((.(((((((((......---...---.....))))))))).))))))..)))..)))..)))).........)))))) ( -34.94, z-score = -3.93, R) >consensus UGAUGGUCGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUGU_G_AAA___GGCACGUAGGAACUCUAUACCUCGCUGAUGGU_____AAAAAGACUUCGGUU .................(((..(((..(((((..((((((((((..................))))))))))..)))))..)))..)))..................... (-26.72 = -26.86 + 0.14)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:15:53 2011