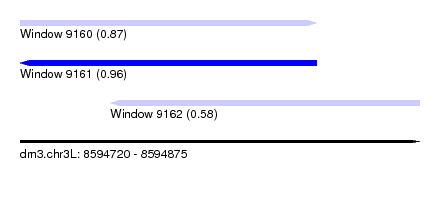
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 8,594,720 – 8,594,875 |
| Length | 155 |
| Max. P | 0.960826 |

| Location | 8,594,720 – 8,594,835 |
|---|---|
| Length | 115 |
| Sequences | 14 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 75.24 |
| Shannon entropy | 0.57891 |
| G+C content | 0.46453 |
| Mean single sequence MFE | -30.06 |
| Consensus MFE | -18.96 |
| Energy contribution | -19.77 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.40 |
| Mean z-score | -0.86 |
| Structure conservation index | 0.63 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.97 |
| SVM RNA-class probability | 0.865037 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
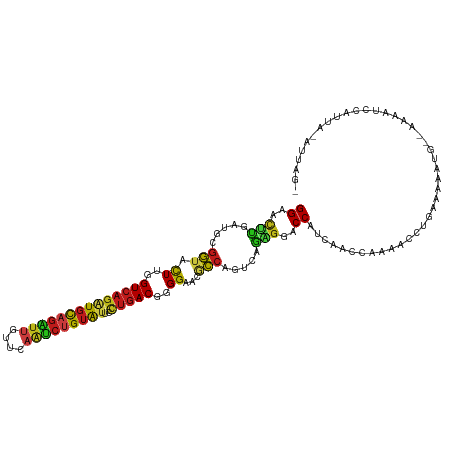
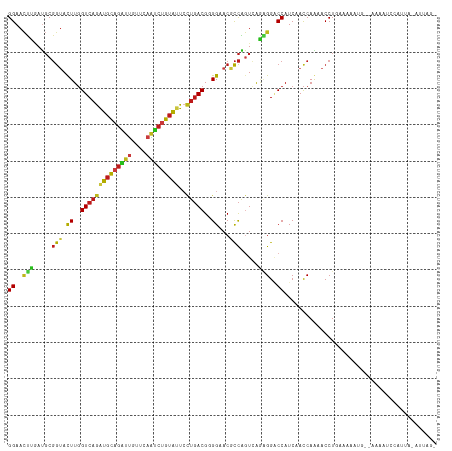
>dm3.chr3L 8594720 115 + 24543557 ACUAAU-UAAUGGCUUUU--CACUUUUCAGGUUCUGGUAGAUGGUCCUCUGACUGGCGUGCCCCGUCAGGAAUACAGAUUGAACAAUCUGCAUCUGACCAAGUACCGCAUCAAGUUCC ......-...........--.((((....(((.((((((((((....((((((.((.....)).))))))....((((((....))))))))))).))).)).))).....))))... ( -28.80, z-score = -0.51, R) >droWil1.scaffold_180949 4991603 113 + 6375548 ----AC-UAAUUUGUUUUGCAUUACCGCAGGUUUUGGUUGAUGGCCCUCUCACCGGUGUUCCCCGUCAGGAAUACAGAUUGAACAAUCUGCAUCUGACCAAGUACCGCAUCAAGUUCC ----..-.((((((...(((......)))((((((((((((((............(((((((......)))))))(((((....))))).)))).))))))).)))....)))))).. ( -30.40, z-score = -1.56, R) >droSim1.chr3L 8036719 115 + 22553184 ACUAAU-UAAUGGCUUUU--CACUCUUCAGGUUCUGGUAGAUGGUCCUCUGACUGGCGUGCCCCGUCAGGAAUACAGAUUGAACAAUCUGCAUCUGACCAAGUACCGCAUCAAGUUCC ......-...........--.........(((.((((((((((....((((((.((.....)).))))))....((((((....))))))))))).))).)).)))............ ( -28.10, z-score = -0.18, R) >droSec1.super_0 873580 115 + 21120651 ACUAAU-UAAUGGCUUUU--CACUCUUCAGGUUCUGGUAGAUGGUCCUCUGACUGGCGUGCCCCGUCAGGAAUACAGAUUAAACAAUCUGCAUCUGACCAAGUACCGCAUCAAGUUCC ......-...........--.........(((.((((((((((....((((((.((.....)).))))))....((((((....))))))))))).))).)).)))............ ( -27.20, z-score = -0.35, R) >droYak2.chr3L 20878931 117 - 24197627 ACUAAU-UACUGGCUUAUCUCACUCUUCAGGUGCUGGUCGAUGGUCCUCUGACUGGCGUGCCCCGUCAGGAAUACAGAUUGAACAAUCUGCACCUGACCAAGUACCGCAUCAAGUUCC ......-......................(((((((((((..(((..((((((.((.....)).))))))....((((((....)))))).)))))))).))))))............ ( -31.90, z-score = -0.79, R) >droEre2.scaffold_4784 20309653 115 - 25762168 ACUAAU-UACUGGCUUAU--CACUCUGCAGGUUCUGGUUGAUGGUCCCCUGACUGGCGUGCCCCGUCAGGAAUACAGAUUGAACAAUCUGCAUCUGACCAAGUACCGCAUCAAGUUCC ......-...........--.....(((.(((.((((((((((....((((((.((.....)).))))))....((((((....)))))))))).)))).)).))))))......... ( -32.60, z-score = -1.07, R) >droAna3.scaffold_13337 15248687 114 - 23293914 GCUAAU-GAUUGCUCUUU---GAUCCAUAGGUUCUGGUCGAUGGCCCUCUGACUGGCGUGCCCCGUCAGGAAUACAGGCUGAGCAACCUGCACCUGACCAAGUACCGCAUCAAGUUCC ((....-....))..(((---(((.....(((.(((((((..((((......((((((.....)))))).......))))..((.....))...))))).)).)))..)))))).... ( -29.42, z-score = 0.75, R) >dp4.chrXR_group8 203941 115 - 9212921 -ACAAC-UAAGC-AUGUUCUUCUCUUUUAGGUUUUGGUUGAUGGUCCUUUGACCGGUGUUCCACGUCAGGAAUACAGAUUGAACAAUCUGCAUCUGACCAAAUACCGCAUUAAGUUCC -.((((-((((.-....(((........))).))))))))..((..(((....(((((((....((((((....((((((....))))))..))))))..)))))))....)))..)) ( -28.50, z-score = -1.26, R) >droPer1.super_50 197594 115 + 557249 -ACAAC-UGAGC-AUGUUCUUCUCUUUUAGGUUUUGGUUGAUGGUCCUUUGACCGGUGUUCCACGUCAGGAAUACAGAUUGAACAAUCUGCAUCUGACCAAAUACCGCAUUAAGUUCC -..(((-((((.-...)))..........(((((((((((((((((....)))).(((((((......)))))))(((((....)))))..))).))))))).)))......)))).. ( -29.30, z-score = -1.12, R) >droVir3.scaffold_13049 6251994 115 + 25233164 -UUAAC-UAUAU-AUACAUAUUGAUUGCAGGUUUUGGUUGAUGGUCCGCUGACUGGUGUUCCACGUCAGGAAUACAGAUUGAACAAUCUGCAUCUGACCAAAUACCGCAUCAAGUUCC -.....-.....-.......(((((.((.((((((((((((((.....((((((((....))).))))).....((((((....)))))))))).))))))).))))))))))..... ( -35.80, z-score = -3.22, R) >droMoj3.scaffold_6680 4567628 115 + 24764193 UUGAAC-UAAUUUAUUUAU--UUCUUACAGGUUUUGGUUGAUGGACCUCUGACUGGUGUUCCACGUCAGGAAUACAGAUUGAACAAUCUGCAUCUGACCAAGUACCGCAUCAAGUUCC ..((((-((((......))--).......((((((((((((((....(((((((((....))).))))))....((((((....)))))))))).))))))).)))......))))). ( -32.30, z-score = -2.41, R) >droGri2.scaffold_15110 18080723 118 - 24565398 AUUAACAUAUUUGAUAUUCAUAAAUUGCAGGUAUUGGUUGAUGGGCCAUUGACCGGUGUUCCACGUCAGGAGUACAGAUUGAACAAUCUGCAUCUGACCAAAUACCGCAUUAAGUUCC .........................(((.((((((((((((((.((..(((((.((....))..)))))..)).((((((....)))))))))).)))).)))))))))......... ( -30.20, z-score = -1.27, R) >anoGam1.chr2L 23999809 114 + 48795086 ----UCCGGCAUCUCUCUCGCUAUCCGCAGGUACUGAUCGAUGGCCCCACCAGCGGCGUUCCCCGUCAACAGUACGCCGUGAACCAUCUGCAUCUGACGAAGUUCCGCGUGCGCUUCC ----((((((.........((.....))..((((((...(.(((.....))).)((((.....))))..)))))))))).)).......((((.((.........)).))))...... ( -27.50, z-score = 1.32, R) >triCas2.ChLG3 28476502 115 + 32080666 ---ACUGUGUUACAUUUUAAUUUUUUCCAGGUGUUGGUUGAUGGGCCUGCUAGUAAGGUACCCCGUGGCCAACUCCGCUUGAAUCAGCUACAUUUGACCAAAUUUAGGCUGAAAUUCC ---..........................((.(((((((.((((((((.......)))...)))))))))))).)).......((((((..((((....))))...))))))...... ( -28.80, z-score = -0.40, R) >consensus _CUAAC_UAAUGGAUUUU__CACUCUUCAGGUUCUGGUUGAUGGUCCUCUGACUGGCGUUCCCCGUCAGGAAUACAGAUUGAACAAUCUGCAUCUGACCAAGUACCGCAUCAAGUUCC .............................(((..(((((((((.....(((((.((....))..))))).....((((((....)))))))))).)))))...)))............ (-18.96 = -19.77 + 0.81)
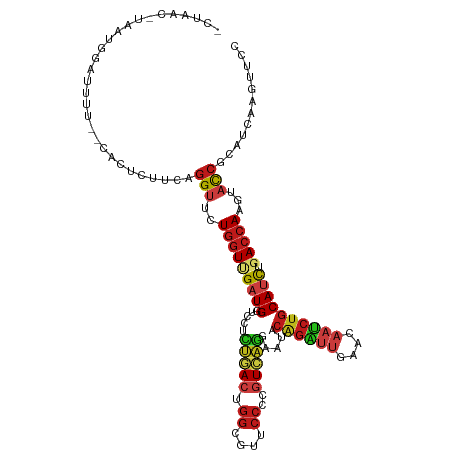
| Location | 8,594,720 – 8,594,835 |
|---|---|
| Length | 115 |
| Sequences | 14 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 75.24 |
| Shannon entropy | 0.57891 |
| G+C content | 0.46453 |
| Mean single sequence MFE | -34.04 |
| Consensus MFE | -19.73 |
| Energy contribution | -19.55 |
| Covariance contribution | -0.18 |
| Combinations/Pair | 1.62 |
| Mean z-score | -1.40 |
| Structure conservation index | 0.58 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.69 |
| SVM RNA-class probability | 0.960826 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 8594720 115 - 24543557 GGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGACCAUCUACCAGAACCUGAAAAGUG--AAAAGCCAUUA-AUUAGU .....(((((((((((..(((((.((((((((((....)))))))))).(((((..((....)).)))))..)))))..))))....(((....)).)--....).)))))-)..... ( -38.40, z-score = -2.90, R) >droWil1.scaffold_180949 4991603 113 - 6375548 GGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGAACACCGGUGAGAGGGCCAUCAACCAAAACCUGCGGUAAUGCAAAACAAAUUA-GU---- ((..(((....((((.(((.((((((((((((((....)))))))))..))))).)))...))))....)))..)).............((((....))))..........-..---- ( -33.40, z-score = -1.32, R) >droSim1.chr3L 8036719 115 - 22553184 GGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGACCAUCUACCAGAACCUGAAGAGUG--AAAAGCCAUUA-AUUAGU ............((((..(((((.((((((((((....)))))))))).(((((..((....)).)))))..)))))..)))).....((((..((((--......)))).-.)))). ( -38.20, z-score = -2.57, R) >droSec1.super_0 873580 115 - 21120651 GGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUUAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGACCAUCUACCAGAACCUGAAGAGUG--AAAAGCCAUUA-AUUAGU ............((((..(((((.((((((((((....)))))))))).(((((..((....)).)))))..)))))..)))).....((((..((((--......)))).-.)))). ( -37.20, z-score = -2.56, R) >droYak2.chr3L 20878931 117 + 24197627 GGAACUUGAUGCGGUACUUGGUCAGGUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGACCAUCGACCAGCACCUGAAGAGUGAGAUAAGCCAGUA-AUUAGU .........((((((...(((((.((((((((((....)))))))))).(((((..((....)).)))))..)))))...))).))).((((..(.((........)).).-.)))). ( -36.80, z-score = -1.06, R) >droEre2.scaffold_4784 20309653 115 + 25762168 GGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGGGGACCAUCAACCAGAACCUGCAGAGUG--AUAAGCCAGUA-AUUAGU ((.((((..((((((.(((((((.((((((((((....))))))))))((((((..((....)).)))))).)))))......)).))).))))))).--.....))....-...... ( -39.10, z-score = -1.94, R) >droAna3.scaffold_13337 15248687 114 + 23293914 GGAACUUGAUGCGGUACUUGGUCAGGUGCAGGUUGCUCAGCCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGGCCAUCGACCAGAACCUAUGGAUC---AAAGAGCAAUC-AUUAGC ((..(((((((((...(((.((((((((((((((....))))))))..)))))).)))..)))..)))).))..))...(((((.......))).))---...((....))-...... ( -40.10, z-score = -1.28, R) >dp4.chrXR_group8 203941 115 + 9212921 GGAACUUAAUGCGGUAUUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCGGUCAAAGGACCAUCAACCAAAACCUAAAAGAGAAGAACAU-GCUUA-GUUGU- ..((((....(((((...(((((.((((((((((....))))))))))..((((.(((....)))))))...)))))...))).....((.....))........-))..)-)))..- ( -30.00, z-score = -1.41, R) >droPer1.super_50 197594 115 - 557249 GGAACUUAAUGCGGUAUUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCGGUCAAAGGACCAUCAACCAAAACCUAAAAGAGAAGAACAU-GCUCA-GUUGU- ..((((......(((...(((((.((((((((((....))))))))))..((((.(((....)))))))...)))))...)))...........(((........-.))))-)))..- ( -29.90, z-score = -1.20, R) >droVir3.scaffold_13049 6251994 115 - 25233164 GGAACUUGAUGCGGUAUUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCAGUCAGCGGACCAUCAACCAAAACCUGCAAUCAAUAUGUAU-AUAUA-GUUAA- .....(((((((((..((((((..((((((((((....))))))..((((((((.(((....)))))))).))).)))).))))))..)))).))))).......-.....-.....- ( -34.20, z-score = -2.60, R) >droMoj3.scaffold_6680 4567628 115 - 24764193 GGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCAGUCAGAGGUCCAUCAACCAAAACCUGUAAGAA--AUAAAUAAAUUA-GUUCAA .(((((...(((((...(((((..((((((((((....)))))))))).(((((.(((....))))))))..........)))))...)))))(...--.).........)-)))).. ( -30.60, z-score = -1.89, R) >droGri2.scaffold_15110 18080723 118 + 24565398 GGAACUUAAUGCGGUAUUUGGUCAGAUGCAGAUUGUUCAAUCUGUACUCCUGACGUGGAACACCGGUCAAUGGCCCAUCAACCAAUACCUGCAAUUUAUGAAUAUCAAAUAUGUUAAU .........(((((((((.((((((.((((((((....))))))))...)))))((((..((........))..))))...).)))))).)))......................... ( -28.50, z-score = -0.96, R) >anoGam1.chr2L 23999809 114 - 48795086 GGAAGCGCACGCGGAACUUCGUCAGAUGCAGAUGGUUCACGGCGUACUGUUGACGGGGAACGCCGCUGGUGGGGCCAUCGAUCAGUACCUGCGGAUAGCGAGAGAGAUGCCGGA---- ((...(.(.(((.....((((.(((.(((.((((((((.((((((.(((....)))...))))))......)))))))).....))).)))))))..))).)...)...))...---- ( -36.30, z-score = 1.13, R) >triCas2.ChLG3 28476502 115 - 32080666 GGAAUUUCAGCCUAAAUUUGGUCAAAUGUAGCUGAUUCAAGCGGAGUUGGCCACGGGGUACCUUACUAGCAGGCCCAUCAACCAACACCUGGAAAAAAUUAAAAUGUAACACAGU--- ..(((((...((.....(((((....(((.((..((((.....))))..)).)))((((..((....))...))))....))))).....))...)))))...............--- ( -23.80, z-score = 0.89, R) >consensus GGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGAACGCCAGUCAGAGGACCAUCAACCAAAACCUGAAAAAUG__AAAAUCCAUUA_AUUAG_ ((..(((.....(((.((..((((((((((((((....)))))))))..)))))..))...))).....)))..)).......................................... (-19.73 = -19.55 + -0.18)

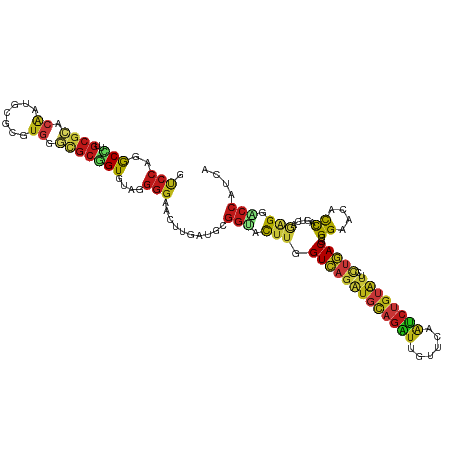
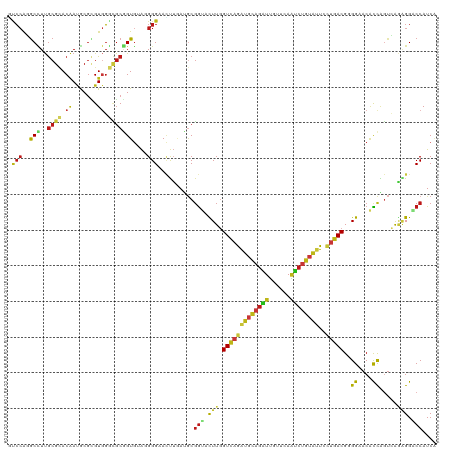
| Location | 8,594,755 – 8,594,875 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.82 |
| Shannon entropy | 0.46287 |
| G+C content | 0.55222 |
| Mean single sequence MFE | -45.86 |
| Consensus MFE | -22.54 |
| Energy contribution | -23.04 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.67 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.49 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.581727 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 8594755 120 - 24543557 GUCCAGGCCUUGCGCACGAUGCGCGUGGGCGCGGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGACCAUCU ((((..(((((((((.....))))).))))(((...((((....)))).))))).)).(((((.((((((((((....)))))))))).(((((..((....)).)))))..)))))... ( -50.90, z-score = -2.11, R) >droWil1.scaffold_180949 4991636 120 - 6375548 UGCCACACUUUGCGCACAAUGCGUGUGGGCGCUGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGAACACCGGUGAGAGGGCCAUCA ..((((((..(((.((((.....)))).)))..)))).))((..(((....((((.(((.((((((((((((((....)))))))))..))))).)))...))))....)))..)).... ( -47.10, z-score = -1.98, R) >apiMel3.GroupUn 341474276 120 + 399230636 UCCCAUGCUUUACGUACAACACGGGUGGAUCCAGUAUAUGGGAAACGUAUACGGAAUUUAGUUAAAUGAAGAUCAUUUAAUCUCAUUUCACAACGUGGAACAUUUGAAGAAGGUCCAUCA ..((...(((((.((....((((.((((((((.(((((((.....)))))))))).....((((((((.....)))))))).....)))))..))))....)).)))))..))....... ( -29.30, z-score = -1.48, R) >droSim1.chr3L 8036754 120 - 22553184 GUCCAGGCCUUGCGCACGAUGCGCGUGGGCGCGGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGACCAUCU ((((..(((((((((.....))))).))))(((...((((....)))).))))).)).(((((.((((((((((....)))))))))).(((((..((....)).)))))..)))))... ( -50.90, z-score = -2.11, R) >droSec1.super_0 873615 120 - 21120651 GUCCAGGCCUUGCGCACGAUGCGCGUGGGCGCGGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUUAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGACCAUCU ((((..(((((((((.....))))).))))(((...((((....)))).))))).)).(((((.((((((((((....)))))))))).(((((..((....)).)))))..)))))... ( -49.90, z-score = -2.12, R) >droYak2.chr3L 20878968 120 + 24197627 GUCCAGGCCUUGCGCACAAUGCGCGUGGGCGCGGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGGUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGACCAUCG ((((...((((((((....(((((....)))))))))))))....((((((((...(((.((((((((((((((....))))))))..)))))).)))..)))..))))).))))..... ( -51.70, z-score = -2.33, R) >droEre2.scaffold_4784 20309688 120 + 25762168 GUCCAGGCCUUGCGCACGAUGCGCGUGGGCGCGGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGGGGACCAUCA ((((..(((((((((.....))))).))))(((...((((....)))).))))).)).(((((.((((((((((....))))))))))((((((..((....)).)))))).)))))... ( -54.20, z-score = -2.83, R) >droAna3.scaffold_13337 15248721 120 + 23293914 GUCCAGGCCUUGCGGACAAUGCGCGUGGGCGCGGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGGUGCAGGUUGCUCAGCCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGGCCAUCG .....((((((.(.(((..((.((((((.((((...((((....)))).)))).).(((.((((((((((((((....))))))))..)))))).))))))))))))).))))))).... ( -54.60, z-score = -1.76, R) >dp4.chrXR_group8 203976 120 + 9212921 GUCCAGGCCUUGCGCACAAUGCGCGUGGGCGCGGUGAAUGGGAACUUAAUGCGGUAUUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCGGUCAAAGGACCAUCA ((((..(((((((((.....))))).)))).(((((...(....)...............((((((((((((((....)))))))))..)))))......)))))......))))..... ( -44.00, z-score = -1.64, R) >droPer1.super_50 197629 120 - 557249 GUCCAGGCCUUGCGCACAAUGCGCGUGGGCGCGGUGAAUGGGAACUUAAUGCGGUAUUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCGGUCAAAGGACCAUCA ((((..(((((((((.....))))).)))).(((((...(....)...............((((((((((((((....)))))))))..)))))......)))))......))))..... ( -44.00, z-score = -1.64, R) >droVir3.scaffold_13049 6252029 120 - 25233164 ACCCAGGCCUUGCGCACAAUGCGUGUGGGCGCGGUGUAUGGGAACUUGAUGCGGUAUUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCAGUCAGCGGACCAUCA .((((.(((.(((.((((.....)))).))).)))...))))................(((((.((((((((((....))))))))))((((((.(((....)))))))).))))))... ( -45.90, z-score = -1.55, R) >droMoj3.scaffold_6680 4567663 120 - 24764193 ACCCAGGCUUUGCGCACAAUGCUUGUGGGCGCGGUGUAUGGGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCAGUCAGAGGUCCAUCA .((((.(((.(((.((((.....)))).))).)))...))))..........((.((((.....((((((((((....)))))))))).(((((.(((....)))))))))))))).... ( -42.00, z-score = -0.93, R) >droGri2.scaffold_15110 18080761 120 + 24565398 ACCCAGGCCUUGCGAACAAUGCGCGUGGCCGCGGUGUAGGGGAACUUAAUGCGGUAUUUGGUCAGAUGCAGAUUGUUCAAUCUGUACUCCUGACGUGGAACACCGGUCAAUGGCCCAUCA (((..((((.((((.......)))).))))(((...((((....)))).))))))....(((((((((((((((....)))))))).)).((((.(((....))))))).)))))..... ( -43.00, z-score = -0.90, R) >anoGam1.chr2L 23999843 120 - 48795086 UCGAGGGCAUCGCGCACCAAACGGGUACGGGCAGUAUGCGGGAAGCGCACGCGGAACUUCGUCAGAUGCAGAUGGUUCACGGCGUACUGUUGACGGGGAACGCCGCUGGUGGGGCCAUCG ......(((((((((..(...((.((((.....)))).)).)..))))..((((....))))..))))).((((((((.((((((.(((....)))...))))))......)))))))). ( -46.40, z-score = -0.61, R) >triCas2.ChLG3 28476537 120 - 32080666 GUCCACGCUUUGCGCACGAUACGAGUCGAAGCUGUGAACGGGAAUUUCAGCCUAAAUUUGGUCAAAUGUAGCUGAUUCAAGCGGAGUUGGCCACGGGGUACCUUACUAGCAGGCCCAUCA ((.(((((((((.((.((...)).)))))))).))).))(((.......((((.....(((((((.....(((......)))....)))))))..)))).(((.......)))))).... ( -34.00, z-score = 0.34, R) >consensus GUCCAGGCCUUGCGCACAAUGCGCGUGGGCGCGGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGAACACCAGUCAGAGGACCAUCA .(((..(((..((((.((.......)).)))))))....)))..........(((.(((.(((((((((((((......))))))))..)))))..((....)).....))).))).... (-22.54 = -23.04 + 0.50)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:11:58 2011